Automatic Segmentation of Covid-19 Infected Regions in Chest CT Images Based on 2D/3D Model Ensembling
-
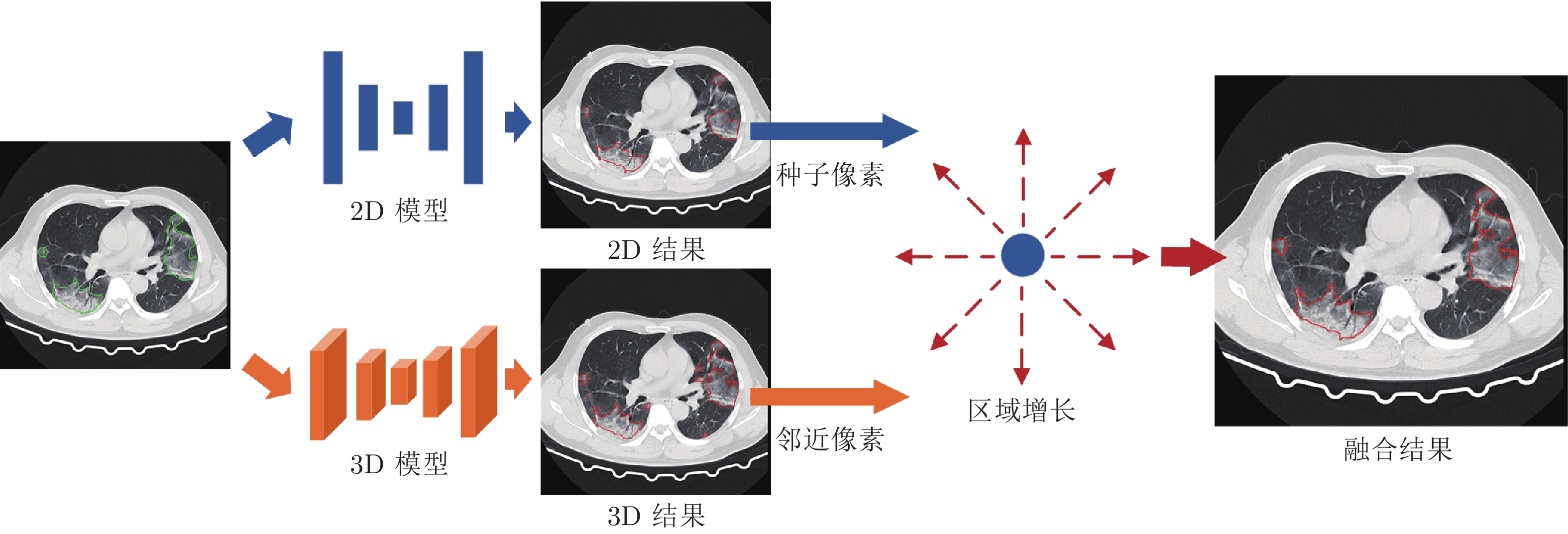
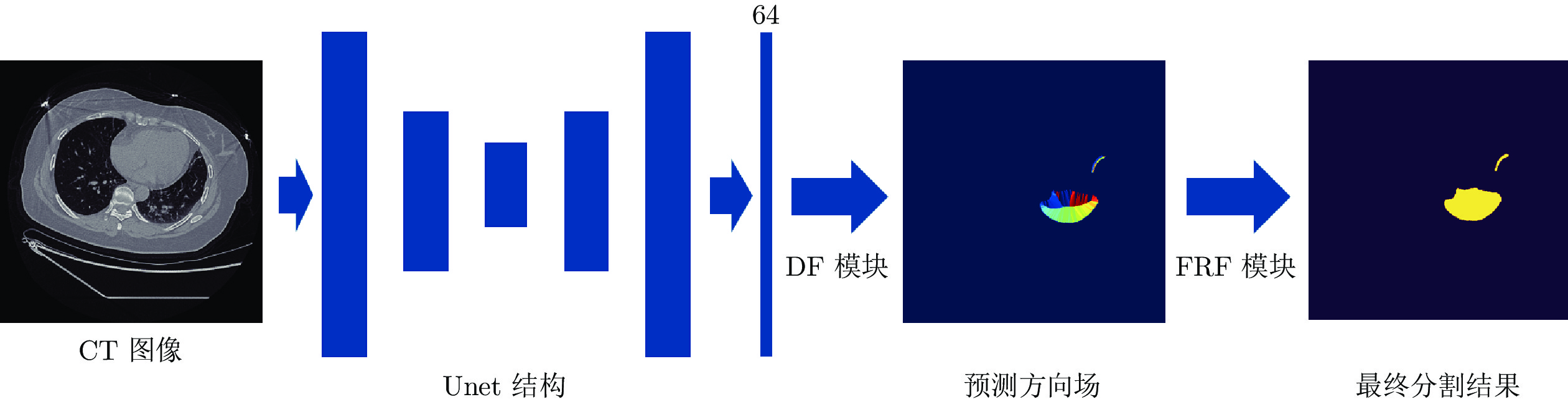
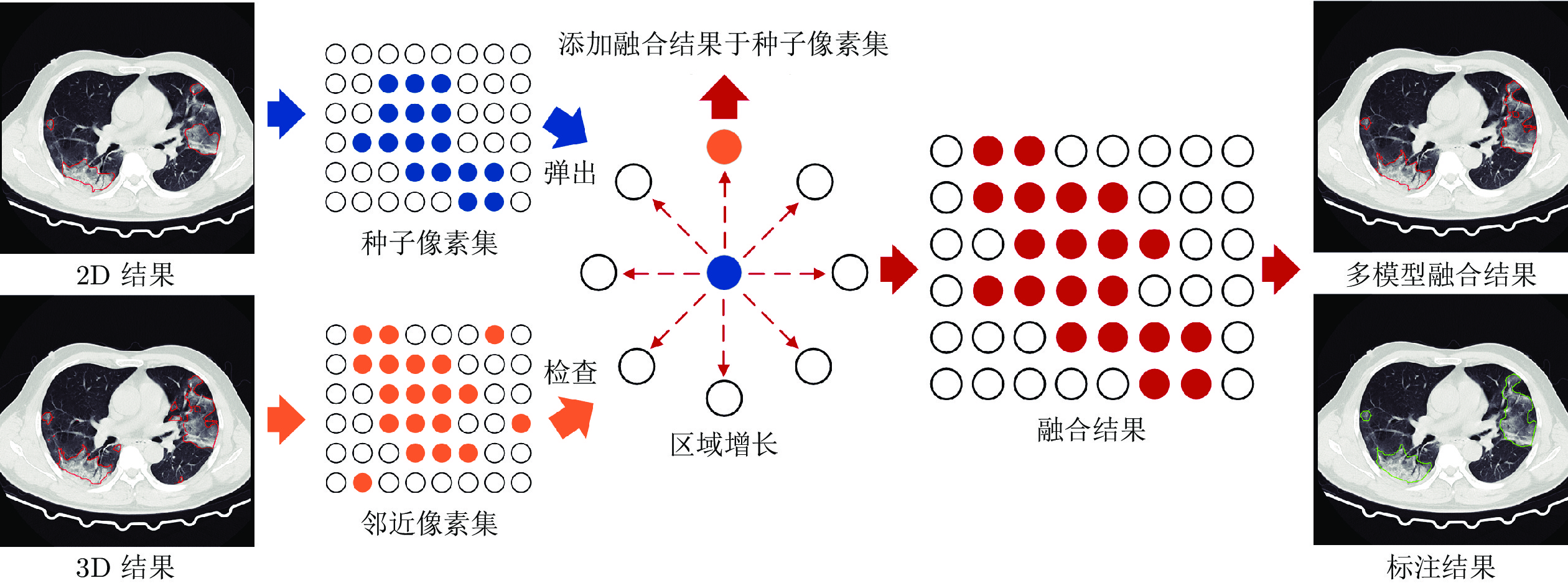
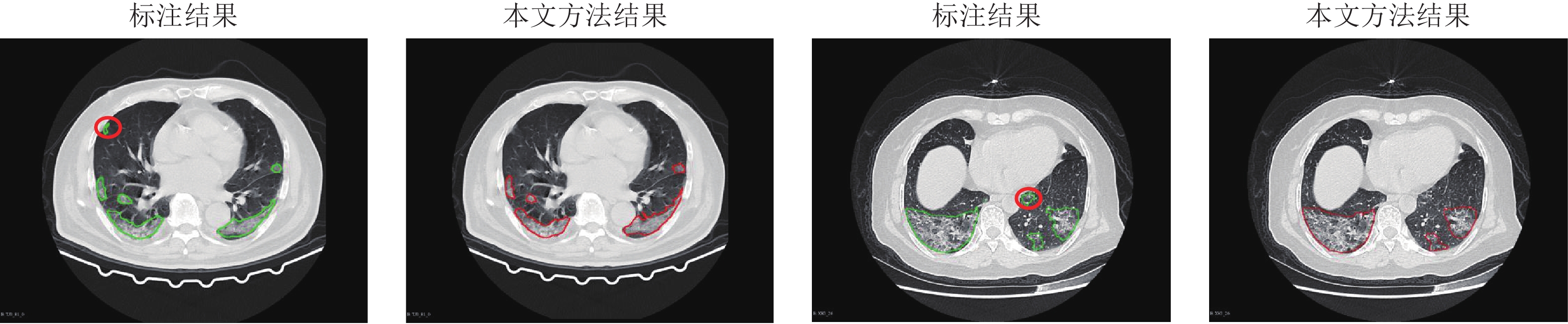
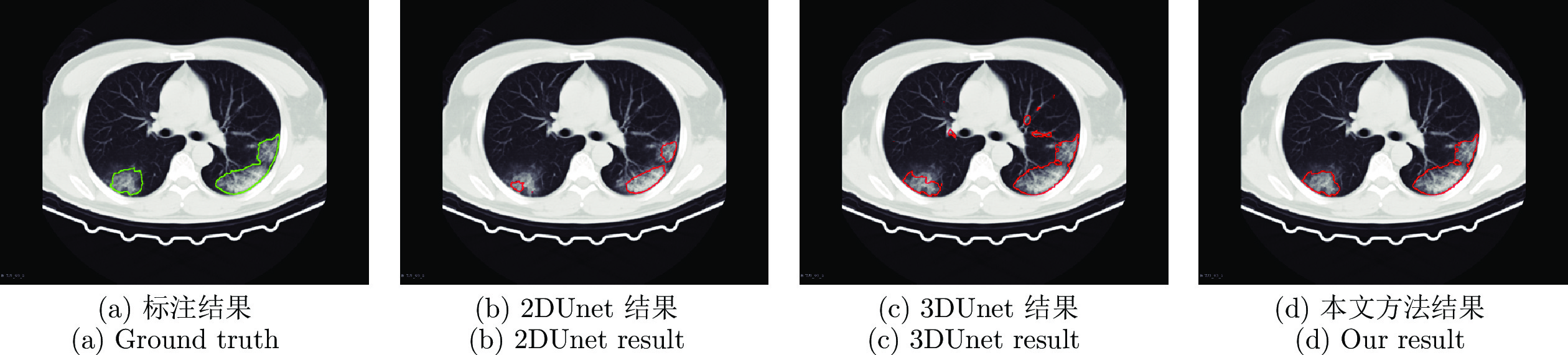
摘要: 自2019年末以来, 全球蔓延的新型冠状病毒(Coronavirus disease 2019, Covid-19)已经给世界人民造成了严重的健康威胁. 其中新型冠状病毒患者的计算机断层扫描(Computer tomography, CT)图像通过肺炎病灶分割技术可以为医学诊断提供有价值的量化信息. 虽然目前基于深度学习的方法已经在新型冠状病毒肺炎病灶分割任务上取得了良好的效果, 但是在面对不同中心数据的情况下分割效果往往会大幅下降. 因此, 研究一种具有更好泛化性能的新型冠状病毒肺炎病灶分割算法具有重要意义. 提出一种新冠肺炎病灶多模型融合分割方法. 通过训练3DUnet模型和2DUnet结合方向场(Direction field, DF)模型, 利用多种模型各自优点进行分割结果的融合, 得到更好的泛化性能. 通过同中心和跨中心数据集的实验, 证明该方法能够有效提高新冠肺炎病灶分割的泛化性能, 为医学诊断分析提供帮助.
-
关键词:
- 新冠肺炎 /
- 计算机断层扫描影像分割 /
- 深度学习 /
- 泛化性能
Abstract: Currently, the global pandemic of coronavirus disease 2019 (Covid-19) causes serious health risks. The coronavirus disease computer tomography (CT) image pneumonia lesion segmentation can provide quantitative information and greatly help for the diagnosis. Although existing deep learning-based methods have achieved good performance on coronavirus disease pneumonia lesion segmentation, the performance usually drops a lot while meeting different center datasets, which is especially common in the coronavirus disease global pandemic. Therefore, it is of great interest to propose an algorithm with better generalization performance for coronavirus disease pneumonia lesion segmentation. In this paper, we present a novel method to fuse multi-models for improving the generalization performance of lesion segmentation. Specifically, we train the 3DUnet and the 2DUnet combined with direction field (2DUnetDF). Then, we fuse 2D and 3D segmentation results to make a better generalization result with the advantage of different models. The in-dataset and cross-dataset experiments show that our method can significantly improve the generalization performance and provide effective help for doctors to analyze the CT images of patients in actual clinical usage.-
Key words:
- Covid-19 /
- CT segmnetation /
- deep learning /
- generalizability
-
表 1 XH150训练在XH40和TJ40测试的Dice结果
Table 1 Dice comparison of methods trained on XH150 and test on XH40 and TJ40 datasets
方法 XH40 TJ40 3DUnet 80.93 77.95 2DUnet 86.94 74.64 3D + 2DUnet (FMM) 87.57 81.90 3D + 2DUnetDF (FMM) 87.93 82.90 表 2 TJ145训练在XH40和TJ40测试的Dice结果
Table 2 Dice comparison of methods trained on TJ145 and test on XH40 and TJ40 datasets
方法 XH40 TJ40 3DUnet 70.17 79.81 2DUnet 78.25 87.08 3D + 2DUnet (FMM) 80.93 87.52 3D + 2DUnetDF (FMM) 82.43 87.62 表 3 不同方法在同数据集测试的Hausdorff95结果
Table 3 In-dataset evaluation of different methods for Hausdorff95
Method XH40 TJ40 3DUnet 4.03 5.59 2DUnet 2.73 4.17 3D + 2DUnet (FMM) 2.74 4.19 3D + 2DUnetDF (FMM) 2.72 4.24 表 4 不同方法在交叉数据集测试的Hausdorff95结果
Table 4 Cross-dataset evaluation of different methods for Hausdorff95
方法 XH40 TJ40 3DUnet 5.39 5.69 2DUnet 3.46 4.68 3D + 2DUnet (FMM) 3.38 4.48 3D + 2DUnetDF (FMM) 3.23 4.46 表 5 XH150训练, 不同融合方法在XH40和TJ40测试Dice的对比结果
Table 5 Dice comparison of methods trained on XH150 test on XH40 and TJ40 datasets with different fusion methods
方法 XH40 TJ40 3DUnet 80.93 77.95 3DUnet-Voting 81.55 79.10 2DUnet 86.94 74.64 2DUnet-Voting 86.81 72.49 3D + 2DUne (FMM) 87.57 81.90 Multi3D2DUnet-Voting 85.05 74.35 Multi3D2DUnet-Voting (FMM) 88.19 82.36 表 6 TJ145训练, 不同融合方法在XH40和TJ40测试Dice的对比结果
Table 6 Dice comparison of methods trained on TJ145 test on XH40 and TJ40 datasets with different fusion methods
方法 XH40 TJ40 3DUnet 70.17 79.81 3DUnet-Voting 73.88 80.54 2DUnet 78.25 87.08 2DUnet-Voting 80.94 87.16 3D + 2DUnet (FMM) 80.93 87.52 Multi3D2DUnet-Voting 82.51 86.73 Multi3D2DUnet-Voting (FMM) 85.48 87.48 表 7 XH150训练在XH40和TJ40上测试的消融实验结果
Table 7 Ablation studies for Dice trained on XH150 test on XH40 and TJ40 datasets
方法 XH40 TJ40 3DUnet 80.93 77.95 2DUnet 86.94 74.64 2DUnetDF 87.83 76.84 3D + 2DUnet (FMM) 87.57 81.90 3D + 2DUnetDF (FMM) 87.93 82.90 表 8 TJ145训练在XH40和TJ40上测试的消融实验结果
Table 8 Ablation studies for Dice trained on TJ145 test on XH40 and TJ40 datasets
方法 XH40 TJ40 3DUnet 70.17 79.81 2DUnet 78.25 87.08 2DUnetDF 80.52 87.57 3D + 2DUnet (FMM) 80.93 87.52 3D + 2DUnetDF (FMM) 82.43 87.62 -
[1] Wang Chen, Horby Peter W, Hayden Frederick G, Gao George F. A novel coronavirus outbreak of global health concern. The Lancet, 2020, 395(10223): 470-473. doi: 10.1016/S0140-6736(20)30185-9 [2] Huang Chao-Lin, Wang Ye-Ming, Li Xing-Wang, Ren Li-Li, Zhao Jian-Ping, Hu Yi, Zhang Li, Fan Guo-Hui, Xu Jiu-Yang, Gu Xiao-Ying, Cheng Zhen-Shun, Yu Ting, Xia Jia-An, Wei Yuan, Wu Wen-Juan, Xie Xue-Lei, Yin Wen, Li Hui, Liu Min, Xiao Yan, Gao Hong, Guo Li, Xie Jun-Gang, Wang Guang-Fa, Jiang Rong-Meng, Gao, Zhan-Cheng, Jin Qi, Wang Jian-wei, Cao Bin. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. The Lancet, 2020, 395(10223): 497-506. doi: 10.1016/S0140-6736(20)30183-5 [3] Coronavirus Covid-19 global cases by the center for systems science and engineering at johns hopkins university [Online], available: https://coronavirus.jhu.edu/map.html, May 7, 2020 [4] Benvenuto Domenico, Giovanetti Marta, Salemi Marco, Prosperi Mattia, De Flora Cecilia, Junior Alcantara Luiz Carlos, Angeletti Silvia, Ciccozzi Massimo. The global spread of 2019-nCoV: a molecular evolutionary analysis. Pathogens and Global Health, 2020, 114(2): 64-67. doi: 10.1080/20477724.2020.1725339 [5] Kissler Stephen M, Tedijanto Christine, Goldstein Edward, Grad Yonatan H, Lipsitch Marc. Projecting the transmission dynamics of SARS-CoV-2 through the postpandemic period. Science, 2020, 368(6493): 860-868. doi: 10.1126/science.abb5793 [6] Ai Tao, Yang Zhen-Lu, Hou Hong-Yan, Zhan Chen-Ao, Chen Chong, Lv Wen-Zhi, Tao Qian, Sun Zi-Yong, Xia Li-Ming. Correlation of chest CT and RT-PCR testing in coronavirus disease 2019 (COVID-19) in China: a report of 1014 cases. Radiology, 2020, 200642. [7] Fang Yi-Cheng, Zhang Huang-Qi, Xie Ji-Cheng, Lin Min-Jie, Ying Ling-Jun, Pang Pei-Pei, Ji Wen-Bin. Sensitivity of chest CT for COVID-19: comparison to RT-PCR. Radiology, 2020, 296(2): E115-E117. doi: 10.1148/radiol.2020200432 [8] Lei Jun-Qiang, Li Jun-Feng, Li Xun, Qi Xiao-Long. CT imaging of the 2019 novel coronavirus (2019-nCoV) pneumonia. Radiology, 2020, 295(1): 18. doi: 10.1148/radiol.2020200236 [9] Li Lin, Qin Li-Xin, Xu Ze-guo, Yin You-Bing, Wang Xin, Kong Bin, Bai Jun-Jie, Lu Yi, Fang Zheng-Han, Song Qi, Cao Kun-lin, Liu Da-liang, Wang Gui-Shen, Xu Qi-Zhong, Fang Xi-Sheng, Zhang Shi-Qin, Xia Juan, Xia Jun. Artificial intelligence distinguishes COVID-19 from community acquired pneumonia on chest CT. Radiology, 2020, 200905. [10] Ng Ming-Yen, Lee Elaine YP, Yang Jin, Yang Fang-Fang, Li Xia, Wang Hong-Xia, Liu Macy Mei-sze, Lo Christine Shing-Yen, Leung Barry, Khong Pek-Lan, Hui Christopher Kim-Ming, Yuen, Kwok-Yung, Kuo MichaelD. Imaging profile of the COVID-19 infection: radiologic findings and literature review. Radiology: Cardiothoracic Imaging, 2020, 2(1): e200034. doi: 10.1148/ryct.2020200034 [11] Shi Feng, Wang Jun, Shi Jun, Wu Zi-Yan, Wang Qian, Tang Zhen-Yu, He Ke-Lei, Shi Ying-Huan, Shen Ding-Gang. Review of artificial intelligence techniques in imaging data acquisition, segmentation and diagnosis for COVID-19. IEEE Reviews in Biomedical Engineering, 2020, 14: 4-15. [12] Kang Heng-Yuan, Xia Li-Ming, Yan Fu-Hua, Wan Zhi-Bin, Shi Feng, Yuan Huan, Jiang Hui-Ting, Wu Di-Jia, Sui He, Zhang Chang-Qing, Shen Ding-Gang. Diagnosis of coronavirus disease 2019 (COVID-19) with structured latent multi-view representation learning. IEEE Transactions on Medical Imaging, 2020, 38(8): 2606-2614 [13] Fang Cong, Bai Song, Chen Qian-Lan, Zhou Yu, Xia Li-Ming, Qin Li-Xin, Gong Shi, Xie Xu-Dong, Zhou Chun-Hua, Tu Dan-Dan, Zhang Chang-Zheng, Liu Xiao-Wu, Chen Wei-Wei, Bai Xiang, Philip H S Torr. Deep learning for predicting COVID-19 malignant progression. Medical Image Analysis, 2021, 72: 102096. doi: 10.1016/j.media.2021.102096 [14] Li Jin-Peng, Zhao Gang-Ming, Tao Ya-Ling, Zhai Peng-Hua, Chen Hao, He Hui-Guang, Cai Ting. Multi-task contrastive learning for automatic CT and X-ray diagnosis of COVID-19. Pattern Recognition, 2021, 114: 107848. doi: 10.1016/j.patcog.2021.107848 [15] Chen Jun, Wu Lian-Lian, Zhang Jun, Zhang Liang, Gong De-Xin, Zhao Yi-Lin, Chen Qiu-Xiang, Huang Shu-Lan, Yang Ming, Yang Xiao, Hu Shan, Wang Yong-Gui, Hu Xiao, Zheng Bi-Qing, Zhang Kuo, Wu Hui-Ling, Dong Ze-Hua, Xu You-Ming, Zhu Yi-Jie, Chen Xi, Zhang, Meng-Jiao, Yu Li-Lei, Cheng Fan, Yu Hong-Gang. Deep learning-based model for detecting 2019 novel coronavirus pneumonia on high-resolution computed tomography. Scientific reports, 2020, 10(1): 1-11. doi: 10.1038/s41598-019-56847-4 [16] Song Ying, Zheng Shuang-Jia, Li Liang, Zhang Xiang, Zhang Xiao-Dong, Huang Zi-Wang, Chen Jian-Wen, Zhao Hui-Ying, Wang Rui-Xuan, Chong Yutian, Shen Jun, Zha Yun-Fei, Yang Yue-Dong. Deep learning enables accurate diagnosis of novel coronavirus (COVID-19) with CT images. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2021, 18(6): 2775-2780. [17] Xu Xiao-Wei, Jiang Xian-Gao, Ma Chun-Lian, Du Peng, Li Xu-Kun, Lv Shuang-Zhi, Yu Liang, Ni Qin, Chen Yan-Fei, Su, Jun-Wei, Lang Guan-Jing, Li Yong-Tao, Zhao Hong, Liu Jun, Xu Kai-Jin, Ruan Ling-Xiang, Sheng Ji-Fang, Qiu Yun-Qing, Wu Wei, Liang Ting-Bo, Li Lan-Juan. Deep learning system to screen coronavirus disease 2019 pneumoni. Engineering, 2020, 6(10): 1122-1129. doi: 10.1016/j.eng.2020.04.010 [18] Wang Xing-Gang, Deng Xian-Bo, Fu Qing, Zhou Qiang, Feng Jia-Pei, Ma Hui, Liu Wen-Yu, Zheng Chuan-Sheng. A weakly-supervised framework for COVID-19 classification and lesion localization from chest CT. IEEE Transactions on Medical Imaging, 2020, 39(8): 2615-2625. doi: 10.1109/TMI.2020.2995965 [19] Fan Deng-Ping, Zhou Tao, Ji Ge-Peng, Zhou Yi, Chen Geng, Fu Hua-Zhu, Shen Jian-Bing, Shao Ling. Inf-net: automatic COVID-19 lung infection segmentation from CT images. IEEE Transactions on Medical Imaging, 2020, 39(8): 2626-2637. doi: 10.1109/TMI.2020.2996645 [20] Xie Wei-Yi, Jacobs Colin, Charbonnier Jean-Paul, Van Ginneken Bram. Relational modeling for robust and efficient pulmonary lobe segmentation in CT scans. IEEE Transactions on Medical Imaging, 2020, 39(8): 2664-2675. doi: 10.1109/TMI.2020.2995108 [21] Wu Yu-Huan, Gao Shang-Hua, Mei Jie, Xu Jun, Fan Deng-Ping, Zhang Rong-Guo, Cheng Ming-Ming. JCS: An explainable COVID-19 diagnosis system by joint classification and segmentation. IEEE Transactions on Image Processing, 2021, 30: 3113-3126. doi: 10.1109/TIP.2021.3058783 [22] Zhao Xiang-Yu, Zhang Peng, Song Fan, Fan Guang-Da, Sun Yang-Yang, Wang Yu-Jia, Tian Zhe-Yuan, Zhang Lu-Qi, Zhang Guang-Lei. D2A U-net: automatic segmentation of COVID-19 CT slices based on dual attention and hybrid dilated convolution. Computers in Biology and Medicine, 2021, 6: 104526. [23] He K M, Zhang X Y, Ren S Q, Sun J. Deep residual learning for image recognition. In: Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Las Vegas, USA: IEEE, 2016. 770−778 [24] Vaswani A, Shazeer N, Parmar N, Uszkoreit J, Jones L, Gomez A N, et al. Attention is all you need. In: Proceedings of the Advances in Neural Information Processing Systems. Long Beach, USA: MIT Press, 2017. 5998−6008 [25] Qiu Y, Liu Y, Li S J, Xu J. MiniSeg: An extremely minimum network for efficient Covid-19 segmentation. In: Proceedings of the AAAI Conference on Artificial Intelligence. Virtual Event: AAAI Press, 2021. 4846−4854 [26] Wang Guo-Tai, Liu Xing-Long, Li Chao-Ping, Xu Zhi-Yong, Ruan Jiu-Gen, Zhu Hai-Feng, Meng Tao, Li Kang, Huang Ning, Zhang Shao-Ting. A noise-robust framework for automatic segmentation of COVID-19 pneumonia lesions from CT images. IEEE Transactions on Medical Imaging, 2020, 39(8): 2653-2663. doi: 10.1109/TMI.2020.3000314 [27] Jiang Yi-Fan, Chen Han, Loew Murray, Ko Hanseok. Covid-19 CT image synthesis with a conditional generative adversarial network. IEEE Journal of Biomedical and Health Informatics, 2020, 25(2): 441-452. [28] Chaganti Shikha, Grenier Philippe, Balachandran Abishek, Chabin Guillaume, Cohen Stuart, Flohr Thomas, Georgescu Bogdan, Grbic Sasa, Liu Si-Qi, Mellot François, Murray Nicolas, Nicolaou Savvas, Parker William, Re Thomas, Sanelli Pina, W Sauter Alexander, Xu Zhou-Bing, Yoo Young-Jin, Ziebandt Valentin, Comaniciu Dorin. Automated quantification of CT patterns associated with COVID-19 from chest CT. Radiology: Artificial Intelligence, 2020, 2(4): e200048. doi: 10.1148/ryai.2020200048 [29] Shan Fei, Gao Yao-Zong, Wang Jun, Shi Wei-Ya, Shi Nan-Nan, Han Miao-Fei, Xue Zhong, Shen Ding-Gang, Shi Yu-Xin. Abnormal lung quantification in chest CT images of COVID-19 patients with deep learning and its application to severity prediction. Medical Physics, 2021, 48(4): 1633-1645. doi: 10.1002/mp.14609 [30] Han Zhong-Yi, Wei Ben-Zheng, Hong Yan-Fei, Li Tian-Yang, Cong Jin-Yu, Zhu Xue, Wei Hai-Feng, Zhang Wei. Accurate screening of COVID-19 using attention-based deep 3D multiple instance learning. IEEE Transactions on Medical Imaging, 2020, 39(8): 2584-2594. doi: 10.1109/TMI.2020.2996256 [31] Cao Yu-Kun, Xu Zhan-Wei, Feng Jian-Jiang, Jin Cheng, Han Xiao-Yu, Wu Han-Ping, Shi He-Shui. Longitudinal assessment of covid-19 using a deep learning-based quantitative ct pipeline: Illustration of two cases. Radiology: Cardiothoracic Imaging, 2020, 2(2): e200082. doi: 10.1148/ryct.2020200082 [32] Huang Lu, Han Rui, Ai Tao, Yu Peng-Xin, Kang Han, Tao Qian, Xia Li-Ming. Serial quantitative chest CT assessment of covid-19: deep-learning approach. Radiology: Cardiothoracic Imaging, 2020, 2(2): e200075. doi: 10.1148/ryct.2020200075 [33] Yue Hong-Mei, Yu Qian, Liu Chuan, Huang Yi-Fei, Jiang Zi-Cheng, Shao Chu-Xiao, Zhang Hong-Guang, Ma Bao-Yi, Wang Yuan-Cheng, Xie Guang-Hang, Zhang Hai-Jun, Li, Xiao-Guo, Kang Ning, Meng Xiang-Pan, Huang Shan, Xu Dan, Lei Jun-Qiang, Huang Hui-Hong, Yang Jie, Ji Jiang-Song, Pan Hong-Qiu, Zou Sheng-Qiang, Ju Sheng-Hong, Qi Xiao-Long. Machine learning-based CT radiomics method for predicting hospital stay in patients with pneumonia associated with SARS-CoV-2 infection: a multicenter study. Annals of Translational Medicine, 2020, 8(14). [34] LAmyar Amine, Modzelewski Romain, Li Hua, Ruan Su. Multi-task deep learning based CT imaging analysis for COVID-19 pneumonia: Classification and segmentation. Computers in Biology and Medicine, 2020, 126: 104037. doi: 10.1016/j.compbiomed.2020.104037 [35] Yan Q, Wang B, Gong D, Luo C, Zhao W, Shen J H, et al. Covid-19 chest CT image segmentation — A deep convolutional neural network solution. arXiv preprint arXiv: 2004.10987, 2020. [36] Oulefki Adel, Agaian Sos, Trongtirakul Thaweesak, Laouar Azzeddine Kassah. Automatic COVID-19 lung infected region segmentation and measurement using CT-scans images. Pattern Recognition, 2021, 114: 107747. doi: 10.1016/j.patcog.2020.107747 [37] Zhou Long-Xi, Li Zhong-Xiao, Zhou Jue-Xiao, Li Hao-Yang, Chen Yu-Peng, Huang Yu-Xin, Xie De-Xuan, Zhao Lin-Tao, Fan Ming, Hashmi Shahrukh, Abdelkareem Faisal, Eiada Riham, Xiao Xi-Gang, Li Li-Hua, Qiu Zhao-Wen, Gao Xin. A rapid, accurate and machine-agnostic segmentation and quantification method for CT-based COVID-19 diagnosis. IEEE Transactions on Medical Imaging, 2020, 39(8): 2638-2652. doi: 10.1109/TMI.2020.3001810 [38] Yao Q S, Xiao L, Liu P H, Zhou S K. Label-free degmentation of Covid-19 lesions in lung CT. IEEE Transactions on Medical Imaging, 2021, 40(10): 2808-2819. [39] Çiçek Ö, Abdulkadir A, Lienkam S S, Brox T, Ronneberger O. 3D U-net: Learning dense volumetric segmentation from sparse annotation. In: Proceedings of the International Conference on Medical Image Computing and Computer-assisted Intervention. Athens, Greece: 2016. 424−432 [40] Ronneberger O, Fischer P, Brox T. U-net: Convolutional networks for biomedical image segmentation. In: Proceedings of the International Conference on Medical Image Computing and Computer-Assisted Intervention. Munich, Germany: 2015. 234− 241 [41] Tang Zhen-Yu, Zhao Wei, Xie Xing-Zhi, Zhong Zheng, Shi Feng, Ma Tian-Min, Liu Jun, Shen Ding-Gang. Severity assessment of COVID-19 using CT image features and laboratory indices. Physics in Medicine & Biology, 2021, 66(3): 035015. [42] Akbari Younes, Hassen Hanadi, Al-Maadeed Somaya, Zughaier Susu M. COVID-19 lesion segmentation using lung CT scan images: comparative study based on active contour models. Applied Sciences, 2020, 11(17): 8039. [43] Shen Cong, Yu Nan, Cai Shu-Bo, Zhou Jie, Sheng Jie-Xin, Liu Kang, Zhou He-Ping, Guo You-Min, Niu Gan. Quantitative computed tomography analysis for stratifying the severity of coronavirus disease 2019. Journal of Pharmaceutical Analysis, 2020, 10(2): 123-129. doi: 10.1016/j.jpha.2020.03.004 [44] Xu Yong-Chao, Wang Yu-Kang, Zhou Wei, Wang Yong-Pan, Yang Zhi-Bo, Bai Xiang. TextField: learning a deep direction field for irregular scene text detection. IEEE Transactions on Image Processing, 2019, 28(11): 5566-5579. doi: 10.1109/TIP.2019.2900589 [45] Wan J Q, Liu Y, Wei D L, Bai X, Xu Y C. Super-BPD: Super boundary-to-pixel direction for fast image segmentation. In: Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Virtual Event: IEEE, 2020. 9253−9262 [46] Cheng F, Chen C, Wang Y L, Shi H S, Cao Y K, Tu D D, et al. Learning directional feature maps for cardiac MRI segmentation. In: Proceedings of the International Conference on Medical Image Computing and Computer-Assisted Intervention. Lima, Peru: 2020. 108−117 [47] Quinlan J R. Bagging, boosting, and C4. In: Proceedings of the AAAI Conference on Artificial Intelligence. Oregon, Porland: AAAI Press, 1996. 725−730 [48] Breiman Leo. Bagging predictors. Machine learning, 1996, 24(2): 123-140. [49] Freund Yoav, Schapire Robert E. A desicion-theoretic generalization of on-line learning and an application to boosting. Journal of Computer and System Sciences, 1997, 55(1): 119-139. doi: 10.1006/jcss.1997.1504 [50] Hastie T, Rosset S, Zhu J, et al. Multi-class adaboost[J]. Statistics and its Interface, 2009, 2(3): 349-360 [51] 赖小波, 许茂盛, 徐小媚. 基于多模型融合和区域迭代生长的视网膜血管自动分割. 电子学报, 2019, 47(12): 2611-2621.Lai Xiao-bo, Xu Mao-Sheng, Xu Xiao-Mei. Automatic retinal vessel segmentation based on multi-model fusion and region iterative growth. Acta Electronica Sinica, 2019, 47(12): 2611-2621. [52] Milletari F, Navab N, Seyed-Ahmad A. V-net: Fully convolutional neural networks for volumetric medical image segmentation. In: Proceedings of the International Conference on 3D Visi-on. Stanford, USA: IEEE, 2016. 565−571 [53] Baid U, Shah N, Talbar S. Brain tumor segmentation with cascaded deep convolutional neural network. In: Proceedings of the International Conference on Medical Image Computing and Computer-assisted Intervention Brainlesion Workshop. Shenzh-en, China: 2019. 90−98 [54] Kingma D P, Ba J. Adam: A method for stochastic optimization. In: Proceedings of the International Conference on Learning Representations. San Diego, USA: 2015. -




 下载:
下载: