-
摘要: 针对特征子集区分度准则(Discernibility of feature subsets, DFS)没有考虑特征测量量纲对特征子集区分能力影响的缺陷, 引入离散系数, 提出GDFS (Generalized discernibility of feature subsets)特征子集区分度准则. 结合顺序前向、顺序后向、顺序前向浮动和顺序后向浮动4种搜索策略, 以极限学习机为分类器, 得到4种混合特征选择算法. UCI数据集与基因数据集的实验测试, 以及与DFS、Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR和FSSC-SD的实验比较和统计重要度检测表明: 提出的GDFS优于DFS, 能选择到分类能力更好的特征子集.Abstract: To overcome the deficiencies of the discernibility of feature subsets (DFS) which cannot take into account the influences from different attribute scales on the discernibility of a feature subset, the generalized DFS, shorted as GDFS, is proposed in this paper by introducing the coefficient of variation. The GDFS is combined with four search strategies, including sequential forward search (SFS), sequential backward search (SBS), sequential forward floating search (SFFS) and sequential backward floating search (SBFS) to develop four hybrid feature selection algorithms. The extreme learning machine (ELM) is adopted as a classification tool to guide feature selection process. We test the classification capability of the feature subsets detected by GDFS on the datasets from UCI machine learning repository and on the classic gene expression datasets, and compare the performance of the ELM classifiers based on the feature subsets by GDFS, DFS and classic feature selection algorithms including Relief, DRJMIM, mRMR, LLE Score, AVC, SVM-RFE, VMInaive, AMID, AMID-DWSFS, CFR, and FSSC-SD respectively. The statistical significance test is also conducted between GDFS, DFS, Relief, DRJMIM, mRMR, LLE Score, AVC, SVM-RFE, VMInaive, AMID, AMID-DWSFS, CFR, and FSSC-SD. Experimental results demonstrate that the proposed GDFS is superior to the original DFS. It can detect the feature subsets with much better capability in classification performance.
-
大数据时代的数据不仅样本量剧增, 维数也日益剧增, 引发维数灾难[1], 增加计算复杂度, 而且冗余和不相关特征使得分类器性能较差, 给数据分析带来挑战. 因此, 特征选择及其评价成为一个研究热点[2-6].
特征选择旨在发现具有强分类能力且互不相关或尽可能互不相关的少量特征构成特征子集. 特征搜索策略包括完全搜索、随机搜索和启发式搜索3大类[7]. 特征选择算法可分为: Filter[8], Wrapper[9], Embedded[10], Hybrid[11-13], 以及Ensemble[14]几大类. Filter方法根据独立于分类器的特征重要性评价准则, 如卡方检验等来判断特征的分类能力, 选择分类性能强的特征构成特征子集. Filter方法独立于学习过程, 速度快, 但需要阈值作为停止准则, 且准确率较低. Wrapper方法依赖于分类器, 需要将训练样本分为训练子集和验证子集两部分, 特征选择则过程中, 以分类器在验证子集的性能判断相应特征子集的分类能力, 选择分类能力强的特征子集. 构建基于特征子集的分类模型, 以测试集对模型进行评价, 从而评价特征子集和相应特征选择算法的性能. Wrapper方法中, 特征选择过程中使用的学习算法完全是一个“黑匣子”. 因此, Wrapper方法依赖于学习过程, 准确率较高, 但计算量大, 且存在过适应风险. Embedded方法通过优化一个目标函数实现特征选择, 特征选择在优化目标函数过程中完成, 不需要将训练样本分成训练子集和验证子集, 但构造合适的优化目标函数困难. Hybrid方法集成Filter方法和Wrapper方法的优势, 采用Filter方法独立于分类器的准则度量特征分类能力大小, 以一定的启发式策略来搜索特征子集, 采用Wrapper方法的以分类器分类性能评价相应特征子集的分类能力. 因此, Hybrid方法得到广泛关注. Ensemble方法集成不同特征选择算法实现特征选择, 一般情况下具有较好性能, 能选择到分类能力较好的特征子集, 但需要训练多个不同分类器.
Relief算法[15]是经典的Filter方法, 但只适用于二分类问题. Relief-F[16]算法将Relief由二分类扩展到多分类问题. LVW (Las Vegas wrapper)算法[17]在拉斯维加斯方法(Las Vegas method)框架下使用随机搜索策略实现特征选择. SVM-RFE (SVM-recursive feature elimination)[18]基于SVM (Support vector machine)和后向剔除思想实现特征选择, 是经典的Embedded特征选择算法, 是为解决超高维基因选择问题提出的算法, 但若每次只剔除一个基因, 时间消耗将成为瓶颈. 为此, 作者Guyon指出, 对于超高维基因选择, 每次迭代, 可一次剔除上百个基因, 但她没有给出到底一次剔除多少个基因合适的理论依据和实践指导. mRMR (Max-relevance, min-redundancy)[19]基于特征相关性, 旨在选择到分类能力强且冗余度最小的特征构成特征子集, 但不同的相关性度量可能会得到不同的结果. F-score[20]是衡量特征在两类间分辨能力的有效准则. Xie等将F-score推广用于任意类分类问题[13, 21], 并提出考虑特征测量量纲的改进F-score特征重要度评价准则D-score[22], 用于皮肤病诊断. 针对F-score和D-score仅考虑单个特征区分能力, 没有考虑特征联合贡献的问题, 谢等提出了考虑特征联合贡献的特征子集区分度衡量准则DFS (Discernibility of feature subsets)[23], 从而获得分类能力更优的特征子集. LLE Score (Locally linear embedding score)[24]算法通过局部线性嵌入, 实现非线性维约简[25], 进行肿瘤基因选择. AVC (Feature selection with AUC-based variable complementarity)算法[26]通过最大化变量互补性实现特征选择. 最大化ROC曲线下面积的基因选择算法[27]实现了非平衡基因数据的特征选择. 特征选择算法DRJMIM (Dynamic relevance and joint mutual information maximization)[28]充分考虑特征相关性和特征相互依赖性, 采用动态相关性和最大化联合互信息实现特征选择. 基于邻域粗糙集的特征选择算法[29]基于邻域熵的不确定性度量, 从基因表达数据集中选择差异表达基因实现癌症分类. 谢等对非平衡基因数据的差异表达基因选择进行了系统研究[30], 提出了16种针对非平衡基因数据的特征选择算法. Li等[31]从数据视图角度对特征选择算法进行总结, 将特征选择算法分为基于相似度的方法、基于信息论的方法、基于稀疏学习的方法, 以及基于统计的方法4大类.
特征选择研究已引起研究者广泛关注, 是高维小样本癌症基因数据分析的首要步骤, 也是其他高维数据分析的基础. 然而, 现有特征选择算法对特征分类能力的评价, 多数仅考虑单个特征的分类贡献, 并忽略了特征测量量纲的影响, DFS[23]准则考虑了特征的联合贡献, 但其没有考虑不同测量量纲对特征分类贡献的影响, 值域差异悬殊的特征, 相当于被赋予了差异悬殊的权重, 无法准确度量特征对分类的贡献量. 为此, 提出GDFS (Generalized discernibility of feature subsets)新准则, 引入离散系数对DFS准则进行改进, 客观度量特征子集的分类能力. 以ELM (Extreme learning machine)为分类工具评估特征子集的分类性能. UCI (University of California in Irvine)机器学习数据库数据集和基因数据集的实验测试, 以及与DFS和现有经典特征选择算法的实验比较与统计显著性检测表明, 提出的GDFS特征子集区分度评价准则是一种有效的特征子集分类能力度量准则, 能选择到分类性能很好的特征子集.
1. GDFS特征子集区分度
设数据集
${\boldsymbol{X}}$ 包含$l\left( {l \geq 2} \right)$ 个类, 第$c\left( {c = 1, \cdots ,l} \right)$ 类样本数为${n_c}$ .1.1 DFS特征子集区分度
DFS特征子集区分度衡量准则[23]考虑特征子集所包含特征的联合作用, 评价特征子集的类别间区分能力大小. 则含有
$i$ 个特征的特征子集的区分度DFS定义为式(1).$$DF{S_i} = \frac{{\sum\limits_{c = 1}^l {\sum\limits_{j = 1}^i {{{\left( {\bar x_j^c - {{\bar x}_j}} \right)}^2}} } }}{{\sum\limits_{c = 1}^l {\frac{1}{{{n_c} - 1}}\sum\limits_{k = 1}^{{n_c}} {\sum\limits_{j = 1}^i {\left( {{{\left( {x_{k,j}^c - \bar x_j^c} \right)}^2}} \right)} } } }}$$ (1) 式(1)分子的
$ {\overline{x}}_{j}^{c}, {\overline{x}}_{j}$ 分别表示第$c$ 类质心(第$c$ 类样本均值)在第$j$ 个特征的取值, 以及整个数据集质心(全部样本均值)在第$j$ 个特征的取值, 因此, 分子表示对应当前$i$ 个特征的特征子集, 样本集$l$ 个类的质心(类中心)到样本集质心(样本集中心)的距离和, 表示类别间的可分性, 值越大表示类别间越疏. 式(1)分母的$x_{k,j}^c$ 表示第$c$ 类的第$k$ 个样本在第$j$ 个特征的取值, 因此, 分母表示对应当前$i$ 个特征的特征子集, 样本$l$ 个类的类内方差之和, 表示类内可聚性, 值越小表示类内越聚[23]. 因此, 式(1)$DF{S_i}$ 的值越大表明包含当前$i$ 个特征的特征子集的分类能力越强[23].1.2 GDFS特征子集区分度
离散系数(变异系数)是样本标准差与样本均值之比, 消除了特征测量量纲对度量样本离散程度的标准差大小的影响, 离散系数越大表明数据离散程度越大, 反之越小[32].
DFS没有考虑特征测量量纲对特征重要度的影响, 不同特征取值范围差异悬殊情况下, 相当于对取值较大特征赋予了较大权重, 使其容易被选择到, 从而影响特征选择结果的客观性. 为了客观度量每个特征的分类能力, 避免特征测量量纲不同带来的影响, 提出GDFS特征子集区分能力度量准则, 克服DFS的缺陷, 以便发现真正具有区分能力的特征. GDFS定义为式(2).
$$GDF{S_i} = \frac{{\frac{1}{{l - 1}}\sum\limits_{c = 1}^l {\left( {\sum\limits_{j = 1}^i {\frac{{{{\left( {\bar x_j^c - {{\bar x}_j}} \right)}^2}}}{{{{\bar x}_j}}}} } \right)} }}{{\sum\limits_{c = 1}^l {\frac{1}{{{n_c} - 1}}\sum\limits_{k = 1}^{{n_c}} {\left( {\sum\limits_{j = 1}^i {\frac{{{{\left( {x_{k,j}^c - \bar x_j^c} \right)}^2}}}{{\bar x_j^c}}} } \right)} } }}$$ (2) 式(2)中分子表示
$l$ 个类别对应当前$i$ 个特征的类别间离散系数, 其值越大, 表示各类别间的分散程度越好; 分母表示$l$ 个类别对应当前$i$ 个特征的类内离散系数之和, 其值越小, 表示各类别越紧凑. 因此, 式(2)的值越大, 表明当前$i$ 个特征构成的特征子集的分类能力越强.1.3 GDFS正确性理论分析
GDFS针对DFS没有考虑特征测度对特征区分能力影响的缺陷提出采用离散系数对DFS进行改进, 因此, 若能证明离散系数不受测度影响, 而标准差受测度影响, 则可证明GDFS正确. 为此, 提出下面的定理, 并进行理论证明.
定理1. 不妨设有包含N个样本, 每个样本拥有n个不同测度特征的数据集
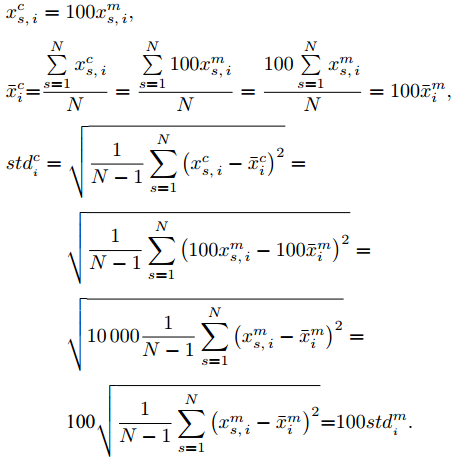
${\boldsymbol{X}} = \{ {{\boldsymbol{x}}_s}| s = 1, \cdots , N \} \in $ $ {{\bf {R}} ^{N \times n}}$ , 如果某一特征${f_i}\left( {i = 1, \cdots ,n} \right)$ 采用米作为度量测度, 则${f_i} \in \left[ {0.5,2} \right]$ , 而若采用厘米作为测度, 则${f_i} \in \left[ {50,200} \right]$ .$std_i^c,std_i^m$ 分别表示特征${f_i}$ 采用厘米和米作为度量测度时的标准差,${\rm{\sigma}} _i^c,{\rm{\sigma}} _i^m$ 分别表示特征${f_i}$ 采用厘米和米作为测度时的离散系数, 则${\rm{\sigma}} _i^c{\rm{ = }}{\rm{\sigma}} _i^m$ ,$std_i^c \ne std_i^m$ .证明. 不妨将特征
${f_i}$ 在数据集${\boldsymbol{X}}$ 的均值记为${\bar x_i}$ , 标准差记为$st{d_i}$ , 离散系数记为${{\rm{\sigma}} _i}$ , 则:$$ st{d_i} = \sqrt {\frac{\sum\limits_{s = 1}^N {\left( {{x_{s,i}} - {{\bar x}_i}} \right)} ^2}{N-1}} ,\;\;\;\;\;{{\rm{\sigma}} _i}{\rm{ = }}\frac{{st{d_i}}}{{{{\bar x}_i}}} $$ 不妨记
${f_i} \in \left[ {50,200} \right]$ 时的标准差为$std_i^c$ , 样本值为$x_{s,i}^c$ , 各样本在特征${f_i}$ 的均值记为$\bar x_i^c$ ;${f_i} \in $ $ \left[ {0.5,2} \right]$ 的标准差为$std_i^m$ , 样本值记为$x_{s,i}^m$ , 各样本在特征${f_i}$ 的均值记为$\bar x_i^m$ . 则:$$ \begin{split} &x_{s,i}^c = 100x_{s,i}^m,\\ &\bar x_i^c{\rm{ = }}\frac{{\sum\limits_{s = 1}^N {x_{s,i}^c} }}{N} = \frac{{\sum\limits_{s = 1}^N {100x_{s,i}^m} }}{N} = \frac{{100\sum\limits_{s = 1}^N {x_{s,i}^m} }}{N} = 100\bar x_i^m,\\ &std_{_i}^c = \sqrt {{\frac{1}{N-1}\sum\limits_{s = 1}^N {\left( {x_{s,i}^c - \bar x_i^c} \right)} ^2}}=\\ &\quad\qquad\sqrt {{\frac{1}{N-1}\sum\limits_{s = 1}^N \left( {100x_{s,i}^m - 100\bar x_i^m} \right) ^2}} = \\ &\quad\qquad \sqrt {10\,000{\frac{1}{N-1}\sum\limits_{s = 1}^N {\left( {x_{s,i}^m - \bar x_i^m} \right)} ^2}} =\\ &\quad\qquad{\rm{ 100}}\sqrt {{\frac{1}{N-1}\sum\limits_{s = 1}^N {\left( {x_{s,i}^m - \bar x_i^m} \right)} ^2}} {\rm{ = }}100std_{_i}^m . \end{split} $$ 特征
${f_i}$ 的离散系数${{\rm{\sigma}} _i}=\dfrac{{st{d_i}}}{{{{\bar x}_i}}}$ . 则:${\rm{\sigma}} _i^c= \dfrac{{std_i^c}}{{\bar x_ \cdot ^c}}= $ $ \dfrac{{100std_i^m}}{{100\bar x_i^m}} = \dfrac{{std_i^m}}{{\bar x_i^m}} = {\rm{\sigma}} _i^m$ .因此,
${\rm{\sigma}} _i^c{\rm{ = }}{\rm{\sigma}} _i^m$ ,$std_i^c \ne std_i^m$ 成立, 即离散系数与特征测度无关, 但方差与标准差均受到特征测度影响. 由此可见, 提出的GDFS在理论上是正确的. □2. 极限学习机
极限学习机ELM是基于单隐层前馈神经网络的机器学习算法[33]. ELM随机产生输入层和隐藏层之间的连接权重和隐藏层阈值, 只需要设定隐藏层结点数便能获得唯一最优的隐藏层到输出层的连接权重.
假设有
$N$ 个训练样本对$\left( {{{\boldsymbol{x}}_i},{{\boldsymbol{t}}_i}} \right)$ ,${{\boldsymbol{x}}_i} \in {{\bf{R}} ^n}$ ,${{\boldsymbol{t}}_i} \in $ $ {{\bf{R}} ^m}$ , 激活函数为$g\left( \cdot \right)$ , 则有$\tilde N$ 个隐结点的单隐层前馈神经网络的数学模型描述为式(3).$$\sum\limits_{j = 1}^{\tilde N} {{{\boldsymbol{\beta }}_j}g\left( {{{\boldsymbol{w}}_j} \cdot {{\boldsymbol{x}}_i} + {b_j}} \right)} = {{\boldsymbol{t}}_i}$$ (3) 其中,
${{\boldsymbol{w}}_{\boldsymbol{j}}}$ 表示第$j$ 个隐结点和所有输入结点间的权重向量,${{\boldsymbol{\beta }}_j}$ 表示第$j$ 个隐结点和所有输出结点间的权重向量,${b_j}$ 是第$j$ 个隐结点的阈值.带有
$\tilde N$ 个隐结点的ELM, 激活函数$g\left( \cdot \right)$ 能够以零误差逼近$N$ 个训练样本, 即存在${{\boldsymbol{\beta }}_j},{{\boldsymbol{w}}_j},{b_j}$ , 使式(3)成立. 式(3)可简写为式(4)矩阵形式.$${\boldsymbol{H\beta }} = {\boldsymbol{T}}$$ (4) 其中,
$$ \begin{array}{l} {\boldsymbol{H}}\left( {{{\boldsymbol{w}}_{1}} ,\cdot \cdot \cdot ,{{\boldsymbol{w}}_{\tilde N}},{b_{1}}, \cdot \cdot \cdot ,{b_{\tilde N}},{{\boldsymbol{x}}_{1}}, \cdot \cdot \cdot ,{{\boldsymbol{x}}_N}} \right)= \\ {\left[ {\begin{array}{*{20}{c}} {g\left( {{{\boldsymbol{w}}_1} \cdot {{\boldsymbol{x}}_1} + {b_1}} \right)}& \cdots &{g\left( {{{\boldsymbol{w}}_{\tilde N}} \cdot {{\boldsymbol{x}}_1} + {b_{\tilde N}}} \right)} \\ \vdots & \cdots & \vdots \\ {g\left( {{{\boldsymbol{w}}_1} \cdot {{\boldsymbol{x}}_N} + {b_1}} \right)}& \cdots &{g\left( {{{\boldsymbol{w}}_{\tilde N}} \cdot {{\boldsymbol{x}}_N} + {b_{\tilde N}}} \right)} \end{array}} \right]_{N \times \tilde N}} \end{array} , $$ ${\boldsymbol{\beta }} = {\left[ {\begin{aligned} {{\boldsymbol{\beta }}_1^{\rm{T}}} \\ \vdots\;\; \\ {{\boldsymbol{\beta }}_{\tilde N}^{\rm{T}}} \end{aligned}} \right]_{\tilde N \times m}},$ ${\boldsymbol{T}} = {\left[ {\begin{aligned} {{\boldsymbol{t}}_1^{\rm{T}}} \\ \vdots \;\; \\ {{\boldsymbol{t}}_N^{\rm{T}}} \end{aligned}} \right]_{N \times m}} .$ ${\boldsymbol{H}}$ 是隐藏层输出矩阵,${\boldsymbol{\beta }}$ 是隐藏层与输出层之间的权值向量矩阵,${\boldsymbol{T}}$ 是输出矩阵.求解式(4)的最小二乘解, 可转化为求解式(5). 根据最小范数准则, ELM的最小二乘解为
$\hat {\boldsymbol{\beta }}= $ $ {{\boldsymbol{H}}^{{ + }}}{\boldsymbol{T}}$ ,${{\boldsymbol{H}}^{{ + }}}$ 为${\boldsymbol{H}}$ 的广义逆矩阵.$$\begin{split} &\left\| {{\boldsymbol{H}}\left( {{{\boldsymbol{w}}_1}, \cdot \cdot \cdot, {{\boldsymbol{w}}_{\tilde N}},{b_1}, \cdot \cdot \cdot ,{b_{\tilde N}}} \right)\hat {\boldsymbol{\beta }} - {\boldsymbol{T}}} \right\|= \\ & \qquad \mathop {\min }\limits_\beta \left\| {{\boldsymbol{H}}\left( {{{\boldsymbol{w}}_1}, \cdot \cdot \cdot ,{{\boldsymbol{w}}_{\tilde N}},{b_1}, \cdot \cdot \cdot ,{b_{\tilde N}}} \right){\boldsymbol{\beta }} - {\boldsymbol{T}}} \right\| \end{split} $$ (5) 3. 基于GDFS的特征选择算法
假设S为包含n个特征的特征全集, C是选择的特征子集, C初始化为空集, 划分数据集为训练集和测试集, 在训练集进行特征选择, 采用SFS, SBS, SFFS和SBFS特征搜索策略, 以GDFS评价特征子集性能, 得到算法1 ~ 4描述的4种混合特征选择算法: GDFS+SFS, GDFS+SBS, GDFS+SFFS, GDFS+SBFS.
算法1. GDFS+SFS特征选择算法
输入: 训练集
${\boldsymbol{X}} \in {{\bf{R}} ^{m \times n}}$ ,${\boldsymbol{S}} = \left\{ {{f_i}\left| {i = 1, \cdots ,n} \right.} \right\},{\boldsymbol{C}} = \Phi .$ //$ \Phi $ 表示空集输出: 特征子集
${\boldsymbol{C}}$ 步骤 1. 计算特征
${f_i}\left( {i = 1, \cdots ,n} \right)$ 的$D{\rm{ - }}score$ 值, 令$K{\rm{ = }}\mathop {{\rm{argmax}}}\limits_{i = 1, \cdots ,n} \left\{ {D{\rm{ - }}score\left( i \right)} \right\}$ , C = C + K, S = S − K;步骤 2. 5-折交叉验证训练ELM分类器, 训练集样本只含有C中全部特征, 记录5-折交叉验证的平均分类准确率
$Acctrain$ ;步骤 3. 判断S是否为空, 若S不空, 将S中的特征逐一与C组合, 构成比当前C特征数多1的临时特征子集tempC, 根据式(2)计算tempC的GDFS值, 选择GDFS值最大的特征子集tempC对应的特征K加入到C, 令S = S − K, 转步骤2; 若S为空, 则算法结束.
取
$Acctrain$ 不再提高时对应的特征子集C为被选择特征子集. 以C中所含特征在训练集构建ELM分类器, 计算测试集的各项指标, 评价特征子集C的分类性能.算法2. GDFS+SBS特征选择算法
输入: 训练集
${\boldsymbol{X}} \in {{\bf{R}} ^{m \times n}}$ ,${\boldsymbol{S}} = \left\{ {{f_i}\left| {i = 1, \cdots ,n} \right.} \right\},{\boldsymbol{C}} = \Phi .$ //$ \Phi $ 表示空集输出: 特征子集
${\boldsymbol{C}}$ 步骤 1. 令C = S;
步骤 2. 计算S的规模
$\left\| {\boldsymbol{S}} \right\|$ , 若$\left\| {\boldsymbol{S}} \right\| \ne 0$ , 则5-折交叉验证训练ELM, 训练样本包括S中全部特征, 记录5-折交叉验证的平均分类准确率$Acctrain$ , 若$\left\| {\boldsymbol{S}} \right\| = 0$ , 则算法结束;步骤 3. 尝试删除S中每一个特征, 计算
$\left\| {\boldsymbol{S}} \right\|$ 个特征数为$\left\| {\boldsymbol{S}} \right\|{\rm{ - }}1$ 的临时特征子集tempS的GDFS, 删除使GDFS值最大的tempS对应特征K, 令S = S − K, C = C − K, 转步骤2.取
$Acctrain$ 不再提高时的特征子集C为被选特征子集. 以C中所含特征在训练集构建ELM分类器, 通过测试集来评价特征子集C的分类性能.算法3. GDFS+SFFS特征选择算法
输入: 训练集
${\boldsymbol{X}} \in {{\bf{R}} ^{m \times n}}$ ,${\boldsymbol{S}} = \left\{ {{f_i}\left| {i = 1, \cdots ,n} \right.} \right\},{\boldsymbol{C}} = \Phi .$ //$ \Phi $ 表示空集输出: 特征子集
${\boldsymbol{C}}$ 步骤 1. 计算特征
${f_i}\left( {i = 1, \cdots ,n} \right)$ 的$D{\rm{ - }}score$ , 令$K{\rm{ = }}\mathop {{\rm{argmax}}}\limits_{i = 1, \cdots ,n} \left\{ {D{\rm{ - }}score\left( i \right)} \right\}$ , C = C + K, S = S − K;步骤 2. 5-折交叉验证训练ELM, 训练集样本只含有C中全部特征, 记录5-折交叉验证的平均分类准确率
$Acctrain$ ;步骤 3. 若
$\left\| {\boldsymbol{S}} \right\| \ne 0$ , 将S中的每一个特征与特征子集C组合, 构成特征数增1的临时特征子集tempC, 计算tempC的GDFS值, 选择GDFS值最大的特征子集tempC对应特征K加入到C, 令S = S − K; 否则, 算法结束;步骤 4. 训练ELM, 训练样本只含有C中全部特征, 并记录相应的
$Acctrain$ ;步骤 5. 若
$Acctrain$ 上升, 则转步骤3; 否则, 从C中删除刚加入的特征K, 然后转步骤3.算法结束时的特征子集
${\boldsymbol{C}} $ 为选择的特征子集. 以C中所含特征构建ELM模型, 通过测试集来评价特征子集的分类性能.算法4. GDFS+SBFS特征选择算法
输入: 训练集
${\boldsymbol{X}} \in {{\bf{R}} ^{m \times n}}$ ,${\boldsymbol{S}}{\rm{ = }}\left\{ {{f_i}\left| {i = 1, \cdots ,n} \right.} \right\},{\boldsymbol{C}} = \Phi .$ //$ \Phi $ 表示空集输出: 特征子集
${\boldsymbol{C}}$ 步骤 1. 令C = S, 5-折交叉验证训练ELM, 训练样本含有S中全部特征, 记录平均分类准确率
$Acctrain$ ;步骤 2. 若
$\left\| {\boldsymbol{S}} \right\| \ne 0$ , 尝试删除S中每一特征, 得到$\left\| {\boldsymbol{S}} \right\|$ 个特征数为$\left\| {\boldsymbol{S}} \right\|{\rm{ - }}1$ 的临时特征子集tempS, 计算tempS的GDFS, 从S中删除GDFS值最大的临时特征子集tempS对应特征K, 即令S = S − K; 否则, 算法结束;步骤 3. 训练ELM, 训练集样本含有当前S中全部特征, 记录相应的
$Acctrain$ ;步骤 4. 若
$Acctrain$ 上升或者保持不变, 则令C = C − K;步骤 5. 转步骤2.
算法结束时,
${\boldsymbol{C}} $ 为选择的特征子集, 构建基于C的ELM, 通过测试集来评价特征子集C的分类性能.4. 实验结果与分析
实验分为4部分, 第1部分验证采用ELM分类器的合理性; 第2部分比较提出的GDFS与原始DFS的性能; 第3部分比较提出的4种特征选择算法与经典算法的性能; 第4部分是算法的统计重要性检测. 其中, 第1部分实验采用原始DFS特征子集评价准则, 以便选择与DFS结合最优的分类器, 这样使第2部分比较提出的GDFS与DFS时, 选择使DFS性能最佳的分类器, 能更凸显提出的GDFS的优越性.
为了避免实验结果受不同数据集划分的影响, 采用5-折交叉验证实验, 以获得平均的实验结果. 并在实验前, 随机打乱样本获得随机实验数据. 打乱方法为: 随机生成一个足够大2维数组, 数组元素的取值为1~数据集规模之间的一个随机数, 交换数组每行两个元素值对应样本.
4.1 ELM与SVM性能比较
本小节采用DFS特征子集评价准则, 结合SFS, SBS, SFFS和SBFS特征搜索策略, 分别采用ELM和SVM分类工具引导特征选择过程, 比较基于相应特征子集的ELM和SVM分类器的性能, 选择分类性能好的分类器. 实验采用UCI机器学习数据库[34]的iris, thyroid-disease, glass, wine, Heart Disease, WDBC (Wisconsin diagnostic breast cancer), WPBC (Wisconsin prognostic breast cancer), dermatology, ionosphere和Handwrite数据集. 数据集描述见表1. thyroid-disease是thyroid gland data数据集; Heart Disease为processed Cleveland, 删掉6个含有缺失数据的样本, 样本数由303变为297; WPBC删掉了4个含有缺失数据的样本, 样本数由198变为194; dermatology删掉了8个含有缺失数据的样本, 因此样本数由366变为358; Handwrite选择了前2类进行实验.
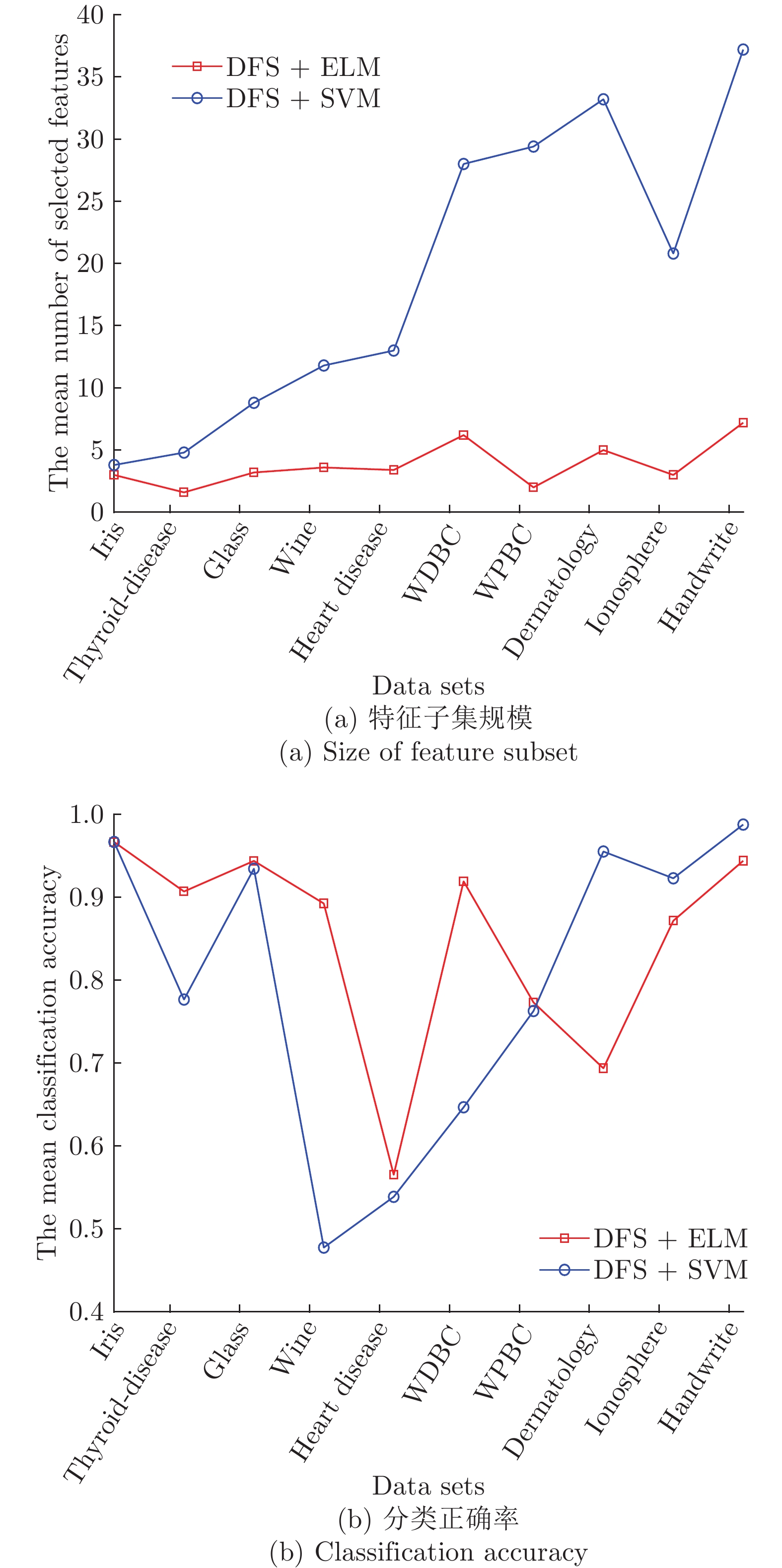
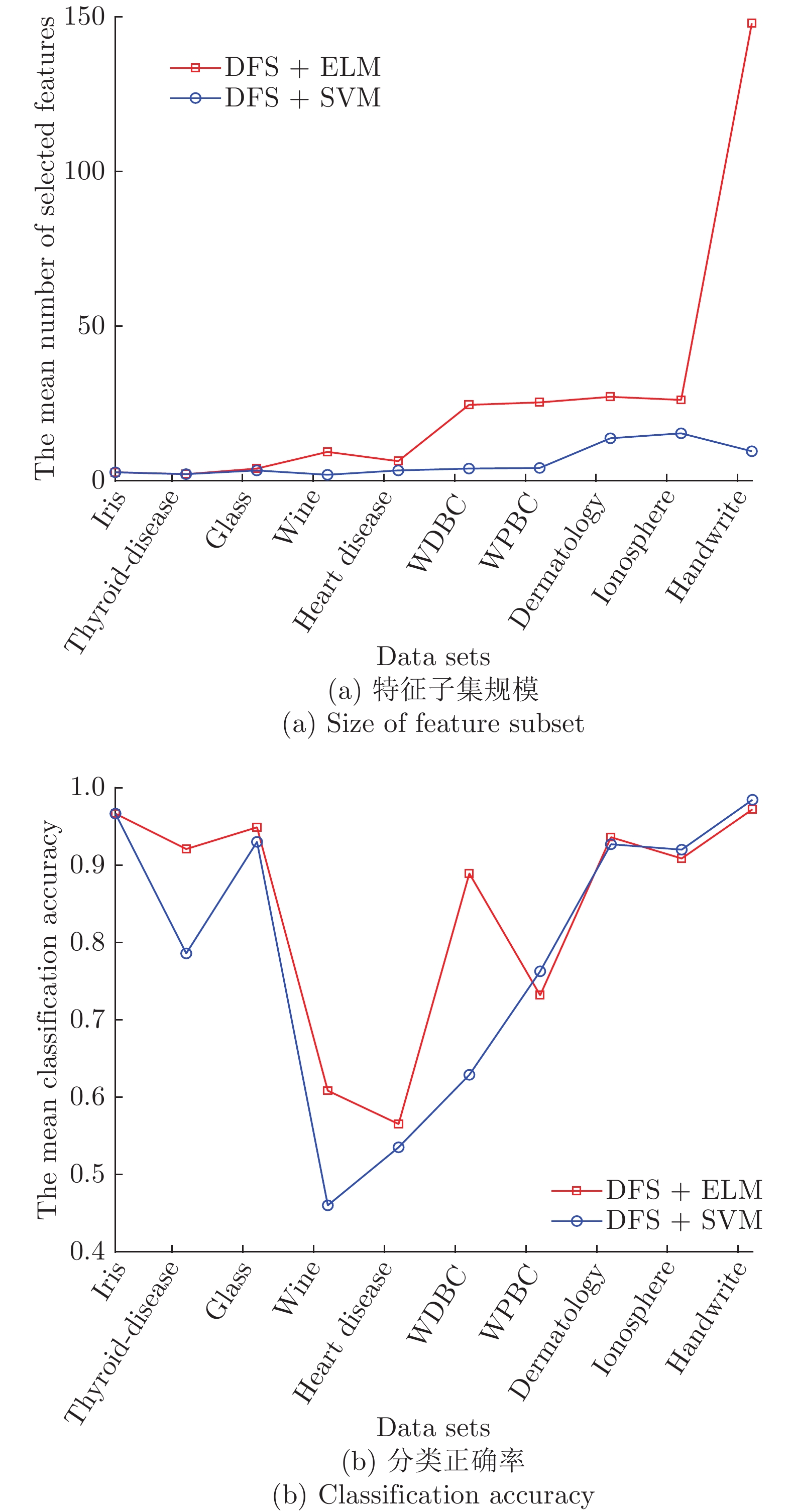
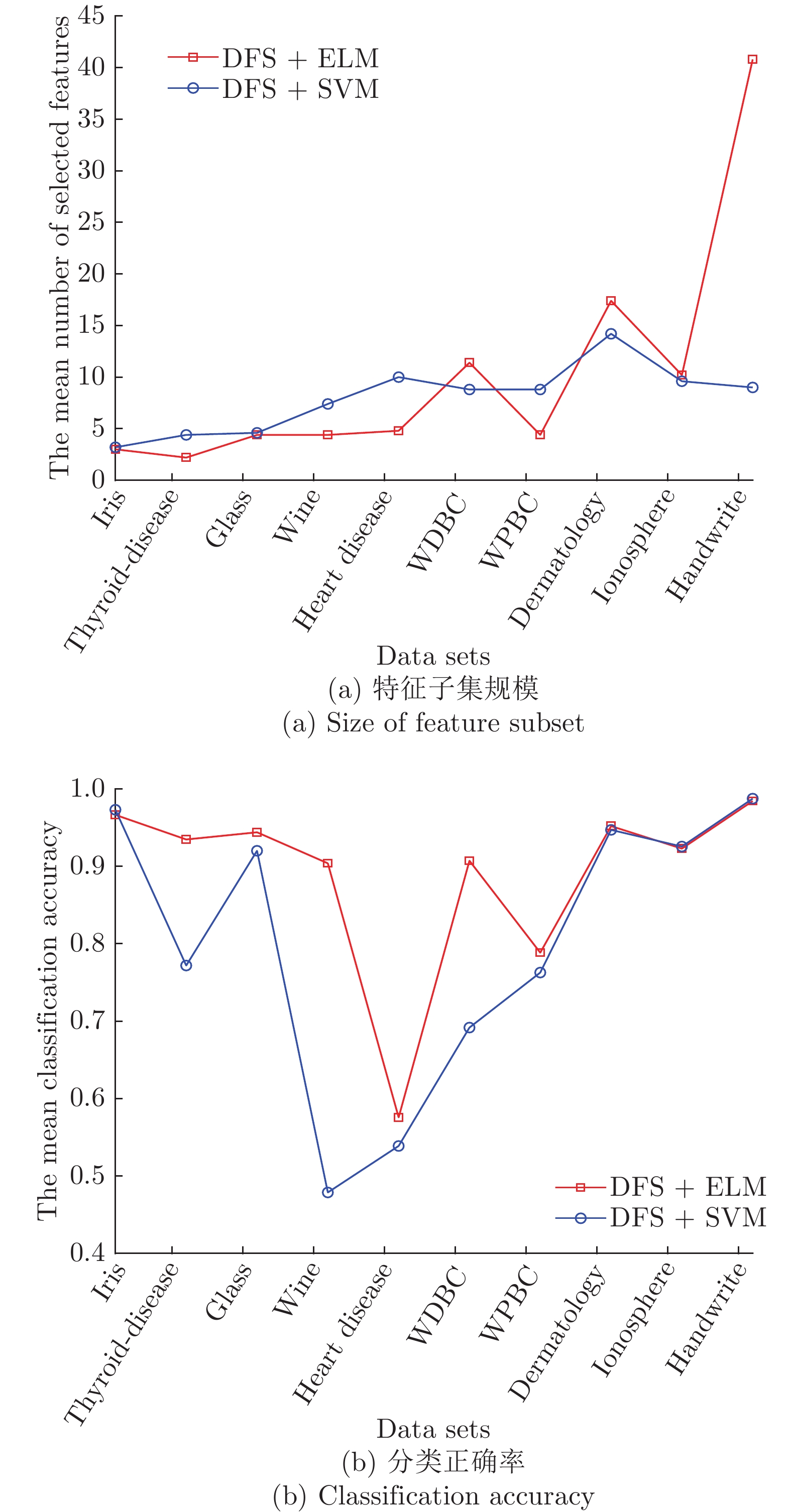
表 1 实验用UCI数据集描述Table 1 Descriptions of datasets from UCI数据集 样本个数 特征数 类别数 iris 150 4 3 thyroid-disease 215 5 3 glass 214 9 2 wine 178 13 3 Heart Disease 297 13 3 WDBC 569 30 2 WPBC 194 33 2 dermatology 358 34 6 ionosphere 351 34 2 Handwrite 323 256 2 SVM分类器采用林智仁等[35]开发的SVM工具箱, 核函数采用RBF (Radial basis function)核函数[36], 参数采用默认值. ELM采用RBF核函数, 参数为默认值, 隐藏层结点数以5为步长增加, 根据交叉验证结果选择最优隐结点数[33]. 为避免ELM的随机初始输入权重向量和隐结点阈值影响实验结果, 实验中设定阈值为0.01, 当训练数据集的分类正确率在一定范围内波动时, 认为分类正确. 图1 ~ 4展示了分别采用ELM与SVM为分类器, 以DFS度量特征子集性能的5-折交叉验证实验平均结果.
图1实验结果显示: 采用SFS搜索策略, 以ELM分类器引导特征选择过程得到的特征子集不仅规模小, 且在绝大部分数据集上的分类性能更好. 图2 ~ 图3实验结果显示, 采用SBS和SFFS搜索策略, 以ELM或SVM为分类器, 除了Handwrite数据集, 其他数据集的特征数量差别不大, 但ELM分类器得到的特征子集分类能力更强. 图4的实验结果显示: ELM分类器选择的特征子集的规模在多数数据集上比SVM得到的特征子集规模稍大, 但ELM分类器得到的特征子集的分类性能优于SVM选择的特征子集的分类性能.
特征选择的目标是: 发现规模小且分类性能好的特征子集. 综合图2 ~ 图4的实验结果可见, 采用ELM分类器能够获得分类能力更好的特征子集.
4.2 GDFS与DFS性能比较
本小节在第4.1节实验基础上, 选择使DFS性能更优的ELM分类器, 测试提出的GDFS特征子集性能评价准则的优越性. 提出的4种特征选择算法GDFS+SFS, GDFS+SBS, GDFS+SFFS, GDFS+SBFS与原DFS+SFS, DFS+SBS, DFS+SFFS, DFS+SBFS在表1数据集的5-折交叉验证的实验结果如表2 ~ 表5所示, 加粗和加下划线表示最优实验结果.
表 2 GDFS+SFS与DFS+SFS算法的5-折交叉验证实验结果Table 2 The 5-fold cross-validation experimental results of GDFS+SFS and DFS+SFS algorithmsData sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.2 3 0.9733 0.9667 thyroid-disease 5 1.4 1.6 0.9163 0.9070 glass 9 2.4 3.2 0.9346 0.9439 wine 13 3.6 3.6 0.9272 0.8925 Heart Disease 13 2.8 3.4 0.5889 0.5654 WDBC 30 3.4 6.2 0.9227 0.9193 WPBC 33 1.8 2 0.7835 0.7732 dermatology 34 4.6 5 0.7151 0.6938 ionosphere 34 4.4 3 0.9029 0.8717 Handwrite 256 7.4 7.2 0.9657 0.9440 平均 43.1 3.4 3.82 0.8630 0.8478 表 5 GDFS+SBFS与DFS+SBFS算法的5-折交叉验证实验结果Table 5 The 5-fold cross-validation experimental results of GDFS+SBFS and DFS+SBFS algorithmsData sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.4 2.8 0.98 0.9667 thyroid-disease 5 2.4 2.2 0.9395 0.9209 glass 9 5.4 4 0.8979 0.9490 wine 13 9.2 9.4 0.6519 0.6086 Heart Disease 13 5.4 6.4 0.5757 0.5655 WDBC 30 22.8 24.6 0.8911 0.8893 WPBC 33 24.6 25.4 0.7681 0.7319 dermatology 34 28.2 27.2 0.9444 0.9362 ionosphere 34 28.4 26.2 0.9174 0.9087 Handwrite 256 137.4 148 0.9938 0.9722 平均 43.1 26.62 27.62 0.8560 0.8449 表 3 GDFS+SBS与DFS+SBS算法的5-折交叉验证实验结果Table 3 The 5-fold cross-validation experimental results of GDFS+SBS and DFS+SBS algorithmsData sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.6 3.2 0.9867 0.9733 thyroid-disease 5 2.8 3.2 0.9269 0.9070 glass 9 8.2 6.8 0.9580 0.9375 wine 13 12 11.6 0.6855 0.6515 Heart Disease 13 11.8 11.8 0.5490 0.5419 WDBC 30 28 28.8 0.8981 0.8616 WPBC 33 30.8 31.6 0.7785 0.7633 dermatology 34 31 31 0.9443 0.9303 ionosphere 34 31.8 32.2 0.9031 0.8947 Handwrite 256 245 248.6 1 0.9936 平均 43.1 40.4 40.88 0.8630 0.8455 表 4 GDFS+SFFS与DFS+SFFS算法的5-折交叉验证实验结果Table 4 The 5-fold cross-validation experimental results of GDFS+SFFS and DFS+SFFS algorithmsData sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.8 3 0.9867 0.9667 thyroid-disease 5 2.2 2.2 0.9395 0.9349 glass 9 4.2 4.4 0.9629 0.9442 wine 13 4.2 4.4 0.9261 0.9041 Heart Disease 13 4.4 4.8 0.5928 0.5757 WDBC 30 11 11.4 0.9385 0.9074 WPBC 33 5.8 4.4 0.7943 0.7886 dermatology 34 16.8 17.4 0.9522 0.9552 ionosphere 34 9.6 10.2 0.9173 0.9231 Handwrite 256 42.2 40.8 0.9907 0.9846 平均 43.1 10.32 10.3 0.8992 0.8885 表2 ~ 表5的5-折交叉验证实验结果显示: GDFS+SFS, GDFS+SBS, GDFS+SFFS和GDFS+SBFS选择的特征子集的分类能力均分别优于DFS+SFS, DFS+SBS, DFS+SFFS和DFS+SBFS算法选择的特征子集的分类能力. 因此, GDFS比DFS选择的特征子集的分类能力更强. 从各算法选择的特征子集规模来看, GDFS+SFS选择的特征子集规模最小, 接着是GDFS+SFFS和GDFS+SBFS算法, GDFS+SBS算法选择的特征子集规模较大. 另外, GDFS+SFS, GDFS+SBS, GDFS+SBFS比DFS+SFS, DFS+SBS, DFS+SBFS选择的特征子集规模平均值略小, GDFS+SFFS与DFS+SFFS选择的特征子集规模基本相当, 前者略大一点.
表2 ~ 表5的5-折交叉验证实验结果还显示, GDFS+SFFS算法选择的特征子集的分类性能最好, GDFS+SFS和GDFS+SBS选择的特征子集的分类能力相当, 不如GDFS+SFFS, 但优于GDFS+SBFS算法选择的特征子集的分类能力.
综上分析可见, 提出的GDFS比原始DFS更优, 能选择到分类能力好且规模较小的特征子集. 其中, GDFS+SFFS算法选择的特征子集分类能力最优, 且规模较小. 因此后面对比实验中仅选择GDFS+SFFS算法与现有经典算法进行比较.
4.3 GDFS与其他特征选择算法的比较
本小节用6个经典基因数据集Colon[37]、Prostate[38]、Myeloma[39]、Gas2[40-41]、SRBCT[42]和Carcinoma[31]进一步测试提出的特征子集性能评价准则GDFS的优越性. 数据集详细信息见表6. 实验将比较提出的GDFS+SFFS与现有特征选择算法DFS+SFFS[23], Relief[15-16], DRJMIM[28], mRMR[19], LLE Score[24], AVC[26], SVM-RFE[18], VMInaive (Variational mutual information)[43], AMID (AUC and mutual information difference)[30], AMID-DWSFS (Dynamic weighted SFS using dynamic AUC and mutual information difference)[30], CFR (Composition of feature relevancy)[44], FSSC-SD (Feature selection by spectral clustering based on standard deviation)[45]选择的特征子集的ELM分类器的分类准确率Accuracy、查准率precision、查全率recall、查准率和查全率的调和平均F-measure、正负类查准率的调和平均F2-measure[30], ROC (Receiver operating characteristic)曲线下面积AUC (Area under and ROC curve)[46-48].
表 6 实验使用的基因数据集描述Table 6 Descriptions of gene datasets using in experiments数据集 样本数 特征数 类别数 Colon 62 2000 2 Prostate 102 12625 2 Myeloma 173 12625 2 Gas2 124 22283 2 SRBCT 83 2308 4 Carcinoma 174 9182 11 由于基因数据集所含特征数成千上万, 为了减少各特征选择算法的运行时间开销, 实验首先采用D-score算法[22]对表6数据集进行特征预选择, 剔除部分不相关和冗余特征, 得到各数据集的候选特征子集, 各算法在候选特征子集上进行特征选择. 表7展示了GDFS+SFFS与特征选择算法DFS+SFFS、Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR及FSSC-SD的5-折交叉验证实验结果, 加粗和下划线表示最优结果. 对比算法的参数设置为: Relief算法的最近邻数为3; LLE Score算法的类内邻域为4, 类外邻域为12; AVC算法的preSelePara参数为默认值.
表 7 各算法在表6基因数据集的5-折交叉验证实验结果Table 7 The 5-fold cross-validation experimental results of all algorithms on datasets from Table 6Data sets 算法 特征数 Accuracy AUC recall precision F-measure F2-measure Colon GDFS+SFFS 5.2 0.7590 0.8925 0.9 0.7 0.78 0.4133 DFS+SFFS 5.4 0.7256 0.78 0.8250 0.6856 0.7352 0.2332 Relief 8 0.7231 0.7575 0.9 0.6291 0.7396 0.16 DRJMIM 13 0.7282 0.7825 0.8750 0.6642 0.7495 0.3250 mRMR 5 0.7602 0.7325 0.85 0.6281 0.7185 0.1578 LLE Score 7 0.7577 0.6563 0.8750 0.6537 0.7431 0.2057 AVC 2 0.7256 0.7297 0.86 0.6439 0.7256 0.2126 SVM-RFE 5 0.7577 0.7588 0.75 0.6273 0.6775 0.3260 VMInaive 2 0.7423 1 1 0.6462 0.7848 0 AMID 8 0.7436 0.95 0.95 0.6328 0.7581 0 AMID-DWSFS 2 0.8397 0.9875 0.9750 0.6688 0.7895 0.1436 CFR 3 0.7603 0.95 1 0.6462 0.7848 0 FSSC-SD 2 0.7269 0.9750 0.9750 0.6401 0.7721 0 Prostate GDFS+SFFS 6.4 0.9305 0.9029 0.8836 0.8836 0.8829 0.8818 DFS+SFFS 6.6 0.9105 0.9349 0.8816 0.8818 0.8529 0.8497 Relief 11 0.93 0.8525 0.8255 0.7824 0.7981 0.79 DRJMIM 9 0.94 0.8629 0.7891 0.8747 0.8216 0.83 mRMR 12 0.9414 0.7895 0.7327 0.7816 0.7520 0.7597 LLE Score 26 0.9119 0.6796 0.7291 0.6582 0.6847 0.6616 AVC 12 0.9514 0.8144 0.7655 0.7598 0.7592 0.7573 SVM-RFE 22 0.92 0.8453 0.6927 0.8474 0.7567 0.7824 VMInaive 9 0.9419 0.8605 0.7655 0.7418 0.7481 0.7580 AMID 27 0.9314 0.7929 0.7655 0.7936 0.7690 0.7797 AMID-DWSFS 4 0.9514 0.7251 0.7127 0.7171 0.7011 0.7098 CFR 7 0.9410 0.7840 0.88 0.7430 0.7922 0.7942 FSSC-SD 23 0.9024 0.7796 0.8018 0.8205 0.7892 0.8130 Myeloma GDFS+SFFS 9.6 0.7974 0.6805 0.8971 0.8230 0.8558 0.5463 DFS+SFFS 9.8 0.7744 0.6296 0.8971 0.8047 0.8474 0.3121 Relief 23 0.8616 0.6453 0.8693 0.8225 0.8415 0.4631 DRJMIM 36 0.8559 0.6210 0.8392 0.7881 0.8124 0.2682 mRMR 12 0.8436 0.6332 0.8095 0.8046 0.8067 0.3539 LLE Score 64 0.8492 0.6169 0.9127 0.7909 0.8461 0.2313 AVC 22 0.8329 0.5820 0.8974 0.8098 0.8501 0.3809 SVM-RFE 20 0.8330 0.6270 0.8971 0.7935 0.8416 0.3846 VMInaive 19 0.8383 0.5639 0.8847 0.7902 0.8331 0.2691 AMID 11 0.8325 0.6743 0.8979 0.8282 0.8603 0.5254 AMID-DWSFS 38 0.8381 0.6233 0.8381 0.8197 0.8249 0.5224 CFR 14 0.8504 0.5931 0.9124 0.8014 0.8523 0.3010 FSSC-SD 15 0.8381 0.6662 0.8754 0.8173 0.8438 0.4992 Gas2 GDFS+SFFS 7.4 0.9840 0.9704 0.9051 0.9846 0.9412 0.9474 DFS+SFFS 8.4 0.9429 0.9465 0.9064 0.9212 0.9203 0.9018 Relief 4 0.9763 0.9520 0.8577 0.9316 0.8911 0.9005 DRJMIM 19 0.9750 0.9004 0.8192 0.8848 0.8449 0.8584 mRMR 5 0.9756 0.9358 0.8551 0.9131 0.8815 0.8895 LLE Score 25 0.9769 0.9312 0.8659 0.8748 0.8449 0.8538 AVC 3 0.9840 0.9073 0.8897 0.9390 0.9122 0.9160 SVM-RFE 18 0.9756 0.9009 0.8205 0.9052 0.8503 0.8716 VMInaive 10 0.9763 0.9425 0.7372 0.9778 0.8311 0.8778 AMID 16 0.9833 0.9305 0.9205 0.8829 0.8968 0.9013 AMID-DWSFS 2 0.9840 0.9247 0.8359 0.9424 0.8839 0.8977 CFR 10 0.9917 0.9080 0.9013 0.8236 0.8432 0.8434 FSSC-SD 16 0.9596 0.9095 0.8538 0.8758 0.8555 0.8642 SRBCT GDFS+SFFS 11.6 0.9372 0.9749 0.9567 0.9684 0.9579 0.9573 DFS+SFFS 11.6 0.9034 0.9130 0.9356 0.9449 0.9452 0.9352 Relief 10 0.9631 0.9479 0.9439 0.9589 0.9467 0.9390 DRJMIM 4 0.9389 0.9363 0.9656 0.9511 0.9555 0.9503 mRMR 8 0.9528 0.9479 0.9283 0.9624 0.9275 0.9294 LLE Score 11 0.9271 0.8941 0.9333 0.9332 0.9247 0.9154 AVC 8 0.9042 0.9355 0.9139 0.9544 0.9223 0.9183 SVM-RFE 13 0.8421 0.9149 0.9128 0.9385 0.9159 0.8240 VMInaive 14 0.9409 0.9181 0.9250 0.9429 0.9269 0.9188 AMID 13 0.9387 0.8999 0.9567 0.9335 0.9407 0.9239 AMID-DWSFS 9 0.9167 0.8151 0.8178 0.8516 0.82 0.7466 CFR 8 0.9314 0.6839 0.8994 0.8570 0.8693 0.7150 FSSC-SD 6 0.8806 0.9096 0.9267 0.9422 0.9284 0.9160 Carcinoma GDFS+SFFS 23.4 0.7622 0.9037 0.7872 0.7879 0.7839 0.5570 DFS+SFFS 19.4 0.7469 0.8998 0.7808 0.7869 0.7801 0.6261 Relief 42 0.7351 0.8701 0.7687 0.7785 0.7680 0.5392 DRJMIM 13 0.7757 0.8991 0.6742 0.6621 0.6656 0.4557 mRMR 24 0.8079 0.9188 0.7613 0.7505 0.7533 0.5089 LLE Score 76 0.6682 0.8452 0.6689 0.6702 0.6663 0.4109 AVC 77 0.7227 0.8746 0.7872 0.7790 0.7796 0.5068 SVM-RFE 30 0.7213 0.87 0.7027 0.6933 0.6929 0.4065 VMInaive 33 0.7443 0.8784 0.7487 0.7527 0.7441 0.4731 AMID 42 0.7307 0.8878 0.7295 0.7165 0.7194 0.4841 AMID-DWSFS 38 0.7412 0.6231 0.7558 0.7447 0.7457 0.4255 CFR 33 0.7054 0.6216 0.7514 0.74 0.7410 0.5315 FSSC-SD 21 0.7306 0.8716 0.7039 0.7016 0.6992 0.4344 表7各算法选择的特征子集的ELM分类器的Accuracy、AUC、recall、precision、F-measure和F2-measure实验结果显示, 提出的GDFS+SFFS算法所选特征子集的分类能力除了在Prostate数据集的AUC、在Gas2的recall、在Carcinoma的F2-measure略低于DFS+SFFS算法外, 在该3个数据集的其他5个评价指标, 以及在其他3个基因数据集的6个评价指标Accuracy、AUC、recall、precision、F-measure和F2-measure均优于原始DFS+SFFS算法. 从特征子集规模来看, 提出的GDFS+SFFS算法除了在Carcinoma数据集的特征子集规模略高于(即选择的特征数稍多于) DFS+SFFS算法外, 在其他数据集得到的特征子集的规模(特征数)都不高于DFS+SFFS. 因此, 可以说提出的特征子集区分度评价准则GDFS优于原始DFS, 能选择到规模较小且分类能力强的特征子集.
另外, 提出的GDFS+SFFS算法所选特征子集的ELM分类器的precision和F2-emeasure在5/6个数据集是最优的, F-measure在4/6个数据集优于所有对比算法, AUC和recall分别在3/6和2/6个数据集上取得所有对比算法的最优值. 对比算法VMInaive在Colon数据集的AUC、recall和F-measure优于对比算法, AUC和recall的值均为最大值1, 但此时其F2-measure为0, 说明该算法将测试集的全部负类样本均误识为正类样本. 算法CFR在Colon数据集也存在选择的特征子集的ELM分类器的recall指标为最大值1, 但F2-measure为0的问题, 也是将测试集的负类样本全部误识为正类样本造成的. 另外, 表7的整体实验结果来看, GDFS+SFFS算法选择的特征子集的分类性能是所有13个算法中最好的.
以上分析显示: 提出的特征子集评价准则GDFS比原始DFS准则更好, 能选择出规模小且分类能力更好的特征子集; 另外, GDFS选择的特征子集的分类能力优于特征选择算法Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR和FSSC-SD所选特征子集的分类能力.
4.4 统计重要性检验
为了检验提出的GDFS+SFFS特征选择算法与对比特征选择算法Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR、FSSC-SD以及DFS+SFFS是否具有统计意义上的显著性区别, 采用Friedman检验来检验各算法之间的差异[49-51]. 在Friedman检验检测到算法间的显著性不同之后, 利用Nemenyi后续检验来检测算法对的两算法之间是否存在统计意义上的显著性不同. 根据Nemenyi检验方法, 在给定统计显著性水平
$\alpha $ 时, 如果任一算法对的两算法之间的平均序数差小于临界阈值CD, 则以置信度$1{\rm{ - }}\alpha $ 接受零假设“两算法性能相同”, 否则拒绝原(零)假设, 认为两算法性能存在显著性不同. 其中临界阈值$CD = {q_\alpha }\sqrt {\frac{{M\left( {M + 1} \right)}}{{6N}}} $ , 这里的M和N分别表示算法个数和数据集个数,${q_\alpha }$ 可通过查表获取. 各算法所选特征子集的ELM分类器的Accuracy、AUC、recall、precision、F-measure和F2-measure在$\alpha {\rm{ = }} 0.05$ 时的Friedman检验结果如表8所示.表 8 各算法所选特征子集分类能力的Friedman检测结果Table 8 The Friedman's test of the classification capability of feature subsets of all algorithmsAccuracy AUC recall precision F-measure F2-measure ${\chi ^2}$ 23.4094 27.5527 22.1585 29.2936 26.7608 32.5446 df 12 12 12 12 12 12 p 0.0244 0.0064 0.0358 0.0036 0.0084 0.0011 由表8的Friedma检验结果可知, 各算法所选特征子集的ELM分类器的Accuracy、AUC、recall、precision、F-measure和F2-measure指标对应的p值均小于0.05. 因此, 我们可以拒绝零假设“各特征选择算法性能相同”, 则各算法所选特征子集在6个基因数据集上的分类性能存在显著性差异.
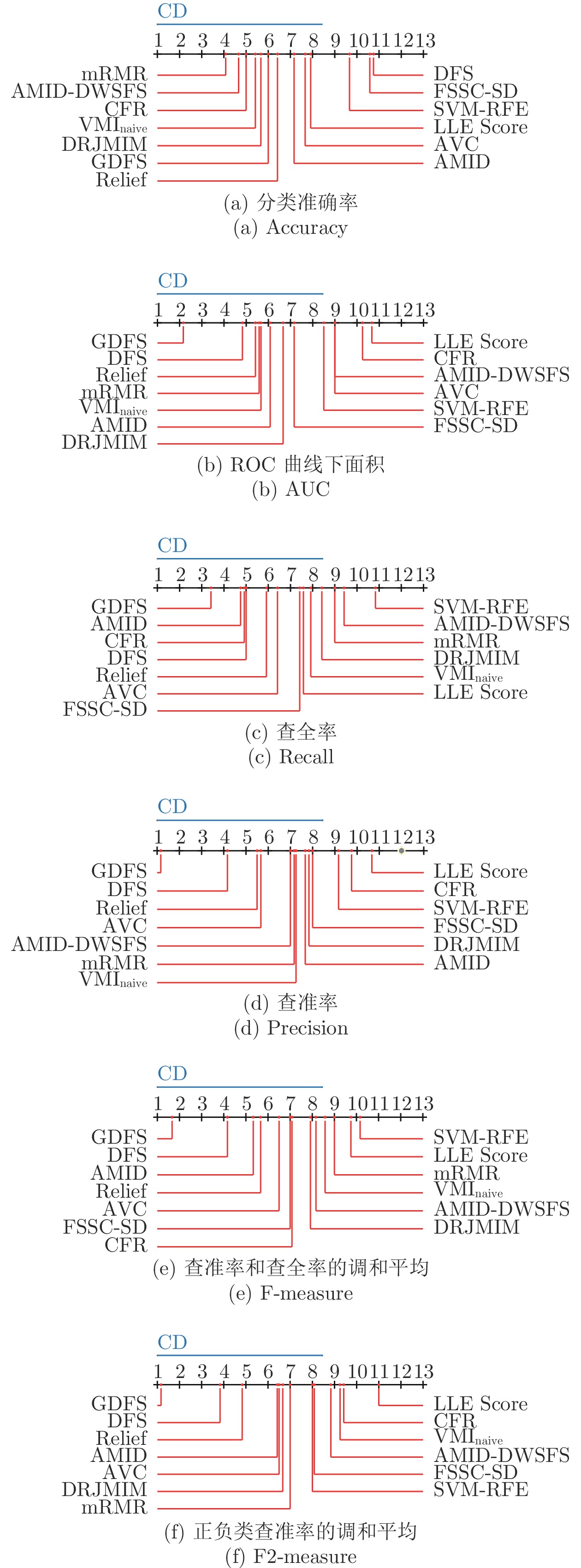
在各算法存在显著性差异的基础上, 采用Nemenyi后续检验来进一步验证各算法对的两算法之间的性能是否显著性不同. 当
$\alpha = 0.05$ , 算法个数为13时, 我们查表可知${q_\alpha }$ = 3.13, 由$CD = $ $ {q_\alpha }\sqrt {\frac{{M\left( {M + 1} \right)}}{{6N}}}$ 计算可得临界阈值CD = 7.4491, 则可信水平为0.95时, 每一对算法采用其选择的特征子集对应ELM分类器的Accuracy、AUC、recall、precision、F-measure和F2-measure指标值的Nemenyi检验结果如图5所示.图5(a)的Nemenyi检验结果显示, GDFS在Accuracy指标上与其他对比算法无显著差异. 众所周知, 基因数据集的不平衡性, 分类准确率已经不适于评价特征子集分类性能[30]. 尽管如此, 图5(a)的检验结果显示, GDFS与其他12种对比算法之间是存在差异的, 与DFS的差异最大, 且优于DFS算法. 图5(b)的Nemenyi检验结果显示, GDFS在AUC指标上与LLE Score和CFR算法存在显著性差异, 且优于LLE Score和CFR算法, 与其他10种对比算法无显著差异, 但存在差异, 且GDFS性能最优. 图5(c)的Nemenyi检验结果显示, GDFS在recall指标上与SVM-RFE存在显著差异, 与其他对比算法无显著差异, 但从实验结果可以看出GDFS与其他11种特征选择算法间存在差异, 且GDFS性能最优, 优于DFS算法. 图5(d)的Nemenyi检验结果可见, GDFS在precision指标上与LLE Score、SVM-RFE和CFR算法存在显著性差异, 且优于LLE Score、SVM-RFE和CFR算法, 与其他9种对比算法无显著差异, 但存在差异, 且优于DFS, 是13种特征选择算法中性能最优的. 图5(e)的Nemenyi检验结果显示, GDFS在F-measure指标上与LLE Score和SVM-RFE算法存在显著性差异, 且优于LLE Score和SVM-RFE算法, 与其他10种对比算法无显著差异, 但存在差异, 且GDFS性能最优, 优于DFS. 图5(f)的Nemenyi检验结果显示, GDFS在F2-measure指标上与LLE Score、CFR、VMInaive和AMID-DWSFS算法存在显著性差异, 且优于LLE Score、VMInaive、AMID-DWSFS和CFR算法, 与其他8种对比算法无显著差异, 但存在差异, 且GDFS性能最优, 优于DFS.
图5各算法的Nemenyi检验结果还显示, 对比算法DFS、Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR和FSSC-SD, 各对算法间不存在统计意义上的显著性差异. 另外, 提出的GDFS优于DFS, 尽管其间没有统计意义上的显著性差异, 但图5的Nemenyi检验结果揭示, 除了recall指标, GDFS与DFS间的等级比较差异值大于2.5, 且recall指标时, GDFS与DFS的等级比较差异值也大于1.5, 这说明尽管GDFS与DFS没有统计意义上的显著性差异, 但其间存在差异. 这一点与表7的实验结果一致.
以上统计重要性分析显示: 提出的GDFS特征子集区分度评价准则优于原始DFS, GDFS+SFFS算法优于12个对比特征选择算法, 能选择到分类性能更好的特征子集. 12个对比算法两两之间不存在显著性差异. 提出的GDFS准则与原始DFS特征子集评价准则选择的特征子集的分类能力有差异, 且GDFS优于DFS, 但不存在统计意义上的显著性差异.
综合以上UCI机器学习数据集和经典基因数据集的5-折交叉验证实验结果得出: 提出的GDFS特征子集区分度评价准则是一种有效的特征子集辨识能力评价准则, UCI机器学习数据集和经典基因数据集的实验测试比较验证了基于该准则的特征选择算法能选择到分类性能更好的特征子集, 达到了保持数据集辨识能力不变情况下进行数据维数压缩的目的.
5. 结论
提出了一种特征子集区分能力评价新准则GDFS, 克服了DFS准则没有考虑特征测量量纲对特征子集区分能力大小影响的缺陷; GDFS结合SFS、SBS、SFFS和SBFS搜索策略, 以ELM为分类器引导特征选择过程, 提出GDFS+SFS、GDFS+SBS、GDFS+SFFS和GDFS+SBFS共4种混合特征选择算法.
UCI机器学习数据集和经典基因数据集的5-折交叉验证实验, 以及与DFS和经典特征选择算法Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR和FSSC-SD的性能比较和统计重要性检验表明, 提出的GDFS特征子集区分度评价准则是一种有效的特征子集辨识能力衡量准则, 其选择的特征子集优于DFS、Relief、DRJMIM、mRMR、LLE Score、AVC、SVM-RFE、VMInaive、AMID、AMID-DWSFS、CFR和FSSC-SD选择的特征子集, 具有更优的分类性能. GDFS准则在提升和保持数据集辨识能力情况下降低了数据的维度.
-
表 1 实验用UCI数据集描述
Table 1 Descriptions of datasets from UCI
数据集 样本个数 特征数 类别数 iris 150 4 3 thyroid-disease 215 5 3 glass 214 9 2 wine 178 13 3 Heart Disease 297 13 3 WDBC 569 30 2 WPBC 194 33 2 dermatology 358 34 6 ionosphere 351 34 2 Handwrite 323 256 2 表 2 GDFS+SFS与DFS+SFS算法的5-折交叉验证实验结果
Table 2 The 5-fold cross-validation experimental results of GDFS+SFS and DFS+SFS algorithms
Data sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.2 3 0.9733 0.9667 thyroid-disease 5 1.4 1.6 0.9163 0.9070 glass 9 2.4 3.2 0.9346 0.9439 wine 13 3.6 3.6 0.9272 0.8925 Heart Disease 13 2.8 3.4 0.5889 0.5654 WDBC 30 3.4 6.2 0.9227 0.9193 WPBC 33 1.8 2 0.7835 0.7732 dermatology 34 4.6 5 0.7151 0.6938 ionosphere 34 4.4 3 0.9029 0.8717 Handwrite 256 7.4 7.2 0.9657 0.9440 平均 43.1 3.4 3.82 0.8630 0.8478 表 5 GDFS+SBFS与DFS+SBFS算法的5-折交叉验证实验结果
Table 5 The 5-fold cross-validation experimental results of GDFS+SBFS and DFS+SBFS algorithms
Data sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.4 2.8 0.98 0.9667 thyroid-disease 5 2.4 2.2 0.9395 0.9209 glass 9 5.4 4 0.8979 0.9490 wine 13 9.2 9.4 0.6519 0.6086 Heart Disease 13 5.4 6.4 0.5757 0.5655 WDBC 30 22.8 24.6 0.8911 0.8893 WPBC 33 24.6 25.4 0.7681 0.7319 dermatology 34 28.2 27.2 0.9444 0.9362 ionosphere 34 28.4 26.2 0.9174 0.9087 Handwrite 256 137.4 148 0.9938 0.9722 平均 43.1 26.62 27.62 0.8560 0.8449 表 3 GDFS+SBS与DFS+SBS算法的5-折交叉验证实验结果
Table 3 The 5-fold cross-validation experimental results of GDFS+SBS and DFS+SBS algorithms
Data sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.6 3.2 0.9867 0.9733 thyroid-disease 5 2.8 3.2 0.9269 0.9070 glass 9 8.2 6.8 0.9580 0.9375 wine 13 12 11.6 0.6855 0.6515 Heart Disease 13 11.8 11.8 0.5490 0.5419 WDBC 30 28 28.8 0.8981 0.8616 WPBC 33 30.8 31.6 0.7785 0.7633 dermatology 34 31 31 0.9443 0.9303 ionosphere 34 31.8 32.2 0.9031 0.8947 Handwrite 256 245 248.6 1 0.9936 平均 43.1 40.4 40.88 0.8630 0.8455 表 4 GDFS+SFFS与DFS+SFFS算法的5-折交叉验证实验结果
Table 4 The 5-fold cross-validation experimental results of GDFS+SFFS and DFS+SFFS algorithms
Data sets #原特征 #选择特征 测试准确率 GDFS DFS GDFS DFS iris 4 2.8 3 0.9867 0.9667 thyroid-disease 5 2.2 2.2 0.9395 0.9349 glass 9 4.2 4.4 0.9629 0.9442 wine 13 4.2 4.4 0.9261 0.9041 Heart Disease 13 4.4 4.8 0.5928 0.5757 WDBC 30 11 11.4 0.9385 0.9074 WPBC 33 5.8 4.4 0.7943 0.7886 dermatology 34 16.8 17.4 0.9522 0.9552 ionosphere 34 9.6 10.2 0.9173 0.9231 Handwrite 256 42.2 40.8 0.9907 0.9846 平均 43.1 10.32 10.3 0.8992 0.8885 表 6 实验使用的基因数据集描述
Table 6 Descriptions of gene datasets using in experiments
数据集 样本数 特征数 类别数 Colon 62 2000 2 Prostate 102 12625 2 Myeloma 173 12625 2 Gas2 124 22283 2 SRBCT 83 2308 4 Carcinoma 174 9182 11 表 7 各算法在表6基因数据集的5-折交叉验证实验结果
Table 7 The 5-fold cross-validation experimental results of all algorithms on datasets from Table 6
Data sets 算法 特征数 Accuracy AUC recall precision F-measure F2-measure Colon GDFS+SFFS 5.2 0.7590 0.8925 0.9 0.7 0.78 0.4133 DFS+SFFS 5.4 0.7256 0.78 0.8250 0.6856 0.7352 0.2332 Relief 8 0.7231 0.7575 0.9 0.6291 0.7396 0.16 DRJMIM 13 0.7282 0.7825 0.8750 0.6642 0.7495 0.3250 mRMR 5 0.7602 0.7325 0.85 0.6281 0.7185 0.1578 LLE Score 7 0.7577 0.6563 0.8750 0.6537 0.7431 0.2057 AVC 2 0.7256 0.7297 0.86 0.6439 0.7256 0.2126 SVM-RFE 5 0.7577 0.7588 0.75 0.6273 0.6775 0.3260 VMInaive 2 0.7423 1 1 0.6462 0.7848 0 AMID 8 0.7436 0.95 0.95 0.6328 0.7581 0 AMID-DWSFS 2 0.8397 0.9875 0.9750 0.6688 0.7895 0.1436 CFR 3 0.7603 0.95 1 0.6462 0.7848 0 FSSC-SD 2 0.7269 0.9750 0.9750 0.6401 0.7721 0 Prostate GDFS+SFFS 6.4 0.9305 0.9029 0.8836 0.8836 0.8829 0.8818 DFS+SFFS 6.6 0.9105 0.9349 0.8816 0.8818 0.8529 0.8497 Relief 11 0.93 0.8525 0.8255 0.7824 0.7981 0.79 DRJMIM 9 0.94 0.8629 0.7891 0.8747 0.8216 0.83 mRMR 12 0.9414 0.7895 0.7327 0.7816 0.7520 0.7597 LLE Score 26 0.9119 0.6796 0.7291 0.6582 0.6847 0.6616 AVC 12 0.9514 0.8144 0.7655 0.7598 0.7592 0.7573 SVM-RFE 22 0.92 0.8453 0.6927 0.8474 0.7567 0.7824 VMInaive 9 0.9419 0.8605 0.7655 0.7418 0.7481 0.7580 AMID 27 0.9314 0.7929 0.7655 0.7936 0.7690 0.7797 AMID-DWSFS 4 0.9514 0.7251 0.7127 0.7171 0.7011 0.7098 CFR 7 0.9410 0.7840 0.88 0.7430 0.7922 0.7942 FSSC-SD 23 0.9024 0.7796 0.8018 0.8205 0.7892 0.8130 Myeloma GDFS+SFFS 9.6 0.7974 0.6805 0.8971 0.8230 0.8558 0.5463 DFS+SFFS 9.8 0.7744 0.6296 0.8971 0.8047 0.8474 0.3121 Relief 23 0.8616 0.6453 0.8693 0.8225 0.8415 0.4631 DRJMIM 36 0.8559 0.6210 0.8392 0.7881 0.8124 0.2682 mRMR 12 0.8436 0.6332 0.8095 0.8046 0.8067 0.3539 LLE Score 64 0.8492 0.6169 0.9127 0.7909 0.8461 0.2313 AVC 22 0.8329 0.5820 0.8974 0.8098 0.8501 0.3809 SVM-RFE 20 0.8330 0.6270 0.8971 0.7935 0.8416 0.3846 VMInaive 19 0.8383 0.5639 0.8847 0.7902 0.8331 0.2691 AMID 11 0.8325 0.6743 0.8979 0.8282 0.8603 0.5254 AMID-DWSFS 38 0.8381 0.6233 0.8381 0.8197 0.8249 0.5224 CFR 14 0.8504 0.5931 0.9124 0.8014 0.8523 0.3010 FSSC-SD 15 0.8381 0.6662 0.8754 0.8173 0.8438 0.4992 Gas2 GDFS+SFFS 7.4 0.9840 0.9704 0.9051 0.9846 0.9412 0.9474 DFS+SFFS 8.4 0.9429 0.9465 0.9064 0.9212 0.9203 0.9018 Relief 4 0.9763 0.9520 0.8577 0.9316 0.8911 0.9005 DRJMIM 19 0.9750 0.9004 0.8192 0.8848 0.8449 0.8584 mRMR 5 0.9756 0.9358 0.8551 0.9131 0.8815 0.8895 LLE Score 25 0.9769 0.9312 0.8659 0.8748 0.8449 0.8538 AVC 3 0.9840 0.9073 0.8897 0.9390 0.9122 0.9160 SVM-RFE 18 0.9756 0.9009 0.8205 0.9052 0.8503 0.8716 VMInaive 10 0.9763 0.9425 0.7372 0.9778 0.8311 0.8778 AMID 16 0.9833 0.9305 0.9205 0.8829 0.8968 0.9013 AMID-DWSFS 2 0.9840 0.9247 0.8359 0.9424 0.8839 0.8977 CFR 10 0.9917 0.9080 0.9013 0.8236 0.8432 0.8434 FSSC-SD 16 0.9596 0.9095 0.8538 0.8758 0.8555 0.8642 SRBCT GDFS+SFFS 11.6 0.9372 0.9749 0.9567 0.9684 0.9579 0.9573 DFS+SFFS 11.6 0.9034 0.9130 0.9356 0.9449 0.9452 0.9352 Relief 10 0.9631 0.9479 0.9439 0.9589 0.9467 0.9390 DRJMIM 4 0.9389 0.9363 0.9656 0.9511 0.9555 0.9503 mRMR 8 0.9528 0.9479 0.9283 0.9624 0.9275 0.9294 LLE Score 11 0.9271 0.8941 0.9333 0.9332 0.9247 0.9154 AVC 8 0.9042 0.9355 0.9139 0.9544 0.9223 0.9183 SVM-RFE 13 0.8421 0.9149 0.9128 0.9385 0.9159 0.8240 VMInaive 14 0.9409 0.9181 0.9250 0.9429 0.9269 0.9188 AMID 13 0.9387 0.8999 0.9567 0.9335 0.9407 0.9239 AMID-DWSFS 9 0.9167 0.8151 0.8178 0.8516 0.82 0.7466 CFR 8 0.9314 0.6839 0.8994 0.8570 0.8693 0.7150 FSSC-SD 6 0.8806 0.9096 0.9267 0.9422 0.9284 0.9160 Carcinoma GDFS+SFFS 23.4 0.7622 0.9037 0.7872 0.7879 0.7839 0.5570 DFS+SFFS 19.4 0.7469 0.8998 0.7808 0.7869 0.7801 0.6261 Relief 42 0.7351 0.8701 0.7687 0.7785 0.7680 0.5392 DRJMIM 13 0.7757 0.8991 0.6742 0.6621 0.6656 0.4557 mRMR 24 0.8079 0.9188 0.7613 0.7505 0.7533 0.5089 LLE Score 76 0.6682 0.8452 0.6689 0.6702 0.6663 0.4109 AVC 77 0.7227 0.8746 0.7872 0.7790 0.7796 0.5068 SVM-RFE 30 0.7213 0.87 0.7027 0.6933 0.6929 0.4065 VMInaive 33 0.7443 0.8784 0.7487 0.7527 0.7441 0.4731 AMID 42 0.7307 0.8878 0.7295 0.7165 0.7194 0.4841 AMID-DWSFS 38 0.7412 0.6231 0.7558 0.7447 0.7457 0.4255 CFR 33 0.7054 0.6216 0.7514 0.74 0.7410 0.5315 FSSC-SD 21 0.7306 0.8716 0.7039 0.7016 0.6992 0.4344 表 8 各算法所选特征子集分类能力的Friedman检测结果
Table 8 The Friedman's test of the classification capability of feature subsets of all algorithms
Accuracy AUC recall precision F-measure F2-measure ${\chi ^2}$ 23.4094 27.5527 22.1585 29.2936 26.7608 32.5446 df 12 12 12 12 12 12 p 0.0244 0.0064 0.0358 0.0036 0.0084 0.0011 -
[1] 陈晓云, 廖梦真. 基于稀疏和近邻保持的极限学习机降维. 自动化学报, 2019, 45(2): 325-333Chen Xiao-Yun, Liao Meng-Zhen. Dimensionality reduction with extreme learning machine based on sparsity and neighborhood preserving. Acta Automatica Sinica, 2019, 45(2): 325-333 [2] Xie J Y, Lei J H, Xie W X, Shi Y, Liu X H. Two-stage hybrid feature selection algorithms for diagnosing erythemato-squamous diseases. Health Information Science and Systems, 2013, 1: Article No. 10 doi: 10.1186/2047-2501-1-10 [3] 谢娟英, 周颖. 一种新聚类评价指标. 陕西师范大学学报(自然科学版), 2015, 43(6): 1-8Xie Juan-Ying, Zhou Ying. A new criterion for clustering algorithm. Journal of Shaanxi Normal University (Natural Science Edition), 2015, 43(6): 1-8 [4] Kou G, Yang P, Peng Y, Xiao F, Chen Y, Alsaadi F E. Evaluation of feature selection methods for text classification with small datasets using multiple criteria decision-making methods. Applied Soft Computing, 2020, 86: Article No. 105836 doi: 10.1016/j.asoc.2019.105836 [5] Xue Y, Xue B, Zhang M J. Self-adaptive particle swarm optimization for large-scale feature selection in classification. ACM Transactions on Knowledge Discovery from Data, 2019, 13(5): Article No. 50 [6] Zhang Y, Gong D W, Gao X Z, Tian T, Sun X Y. Binary differential evolution with self-learning for multi-objective feature selection. Information Sciences, 2020, 507: 67-85. doi: 10.1016/j.ins.2019.08.040 [7] Nguyen B H, Xue B, Zhang M J. A survey on swarm intelligence approaches to feature selection in data mining. Swarm and Evolutionary Computation, 2020, 54: Article No. 100663 doi: 10.1016/j.swevo.2020.100663 [8] Solorio-Fernández S, Carrasco-Ochoa J A, Martínez-Trinidad J F. A review of unsupervised feature selection methods. Artificial Intelligence Review, 2020, 53(2): 907-948 doi: 10.1007/s10462-019-09682-y [9] Karasu S, Altan A, Bekiros S, Ahmad W. A new forecasting model with wrapper-based feature selection approach using multi-objective optimization technique for chaotic crude oil time series.Energy, 2020, 212: Article No. 118750 doi: 10.1016/j.energy.2020.118750 [10] Al-Tashi Q, Abdulkadir S J, Rais H, Mirjalili S, Alhussian H. Approaches to multi-objective feature selection: A systematic literature review. IEEE Access, 2020, 8: 125076-125096 doi: 10.1109/ACCESS.2020.3007291 [11] Deng X L, Li Y Q, Weng J, Zhang J L. Feature selection for text classification: A review. Multimedia Tools and Applications, 2019, 78(3): 3797-3816 doi: 10.1007/s11042-018-6083-5 [12] 贾鹤鸣, 李瑶, 孙康健. 基于遗传乌燕鸥算法的同步优化特征选择. 自动化学报, DOI: 10.16383/j.aas.c200322Jia He-Ming, Li Yao, Sun Kang-Jian. Simultaneous feature selection optimization based on hybrid sooty tern optimization algorithm and genetic algorithm. Acta Automatica Sinica, DOI: 10.16383/j.aas.c200322 [13] Xie J Y, Wang C X. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems With Applications, 2011, 38(5): 5809-5815 doi: 10.1016/j.eswa.2010.10.050 [14] Bolón-Canedo V, Alonso-Betanzos A. Ensembles for feature selection: A review and future trends. Information Fusion, 2019, 52: 1-12 doi: 10.1016/j.inffus.2018.11.008 [15] Kira K, Rendell L A. The feature selection problem: Traditional methods and a new algorithm. In: Proceedings of the 10th National Conference on Artificial Intelligence. San Jos, USA: AAAI Press, 1992. 129−134 [16] Kononenko I. Estimating attributes: Analysis and extensions of RELIEF. In: Proceedings of the 7th European Conference on Machine Learning. Catania, Italy: Springer, 1994. 171−182 [17] Liu H, Setiono R. Feature selection and classification — a probabilistic wrapper approach. In: Proceedings of the 9th International Conference on Industrial and Engineering Applications of Artificial Intelligence and Expert Systems. Fukuoka, Japan: Gordon and Breach Science Publishers, 1997. 419−424 [18] Guyon I, Weston J, Barnhill S. Gene selection for cancer classification using support vector machines. Machine Learning, 2002, 46(1-3): 389-422 [19] Peng H C, Long F H, Ding C. Feature selection based on mutual information criteria of max-dependency, max-relevance, and min-redundancy. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2005, 27(8): 1226-1238 doi: 10.1109/TPAMI.2005.159 [20] Chen Y W, Lin C J. Combining SVMs with various feature selection strategies. Feature Extraction: Foundations and Applications. Berlin, Heidelberg: Springer, 2006. 315−324 [21] 谢娟英, 王春霞, 蒋帅, 张琰. 基于改进的F-score与支持向量机的特征选择方法. 计算机应用, 2010, 30(4): 993-996 doi: 10.3724/SP.J.1087.2010.00993Xie Juan-Ying, Wang Chun-Xia, Jiang Shuai, Zhang Yan. Feature selection method combing improved F-score and support vector machine. Journal of Computer Applications, 2010, 30(4): 993-996 doi: 10.3724/SP.J.1087.2010.00993 [22] 谢娟英, 雷金虎, 谢维信, 高新波. 基于D-score与支持向量机的混合特征选择方法. 计算机应用, 2011, 31(12): 3292-3296Xie Juan-Ying, Lei Jin-Hu, Xie Wei-Xin, Gao Xin-Bo. Hybrid feature selection methods based on D-score and support vector machine. Journal of Computer Applications, 2011, 31(12): 3292-3296 [23] 谢娟英, 谢维信. 基于特征子集区分度与支持向量机的特征选择算法. 计算机学报, 2014, 37(8): 1704-1718Xie Juan-Ying, Xie Wei-Xin. Several feature selection algorithms based on the discernibility of a feature subset and support vector machines. Chinese Journal of Computers, 2014, 37(8): 1704-1718 [24] 李建更, 逄泽楠, 苏磊, 陈思远. 肿瘤基因选择方法LLE Score. 北京工业大学学报, 2015, 41(8): 1145-1150Li Jian-Geng, Pang Ze-Nan, Su Lei, Chen Si-Yuan. Feature selection method LLE score used for tumor gene expressive data. Journal of Beijing University of Technology, 2015, 41(8): 1145-1150 [25] Roweis S T, Saul L K. Nonlinear dimensionality reduction by locally linear embedding. Science, 2000, 290(5500): 2323-2326 doi: 10.1126/science.290.5500.2323 [26] Sun L, Wang J, Wei J M. AVC: Selecting discriminative features on basis of AUC by maximizing variable complementarity. BMC Bioinformatics, 2017, 18(Suppl 3): Article No. 50 [27] 谢娟英, 王明钊, 胡秋锋. 最大化ROC曲线下面积的不平衡基因数据集差异表达基因选择算法. 陕西师范大学学报(自然科学版), 2017, 45(1): 13-22Xie Juan-Ying, Wang Ming-Zhao, Hu Qiu-Feng. The differentially expressed gene selection algorithms for unbalanced gene datasets by maximize the area under ROC. Journal of Shaanxi Normal University (Natural Science Edition), 2017, 45(1): 13-22 [28] Hu L, Gao W F, Zhao K, Zhang P, Wang F. Feature selection considering two types of feature relevancy and feature interdependency. Expert Systems With Applications, 2018, 93: 423-434 doi: 10.1016/j.eswa.2017.10.016 [29] Sun L, Zhang X Y, Qian Y H, Xu J C, Zhang S G. Feature selection using neighborhood entropy-based uncertainty measures for gene expression data classification. Information Sciences, 2019, 502:18-41 doi: 10.1016/j.ins.2019.05.072 [30] 谢娟英, 王明钊, 周颖, 高红超, 许升全. 非平衡基因数据的差异表达基因选择算法研究. 计算机学报, 2019, 42(6): 1232-1251 doi: 10.11897/SP.J.1016.2019.01232Xie Juan-Ying, Wang Ming-Zhao, Zhou Ying, Gao Hong-Chao, Xu Sheng-Quan. Differential expression gene selection algorithms for unbalanced gene datasets. Chinese Journal of Computers, 2019, 42(6): 1232-1251 doi: 10.11897/SP.J.1016.2019.01232 [31] Li J D, Cheng K W, Wang S H, Morstatter F, Trevino R P, Tang J L, et al. Feature selection: A data perspective. ACM Computing Surveys, 2018, 50(6): Article No. 94 [32] 刘春英, 贾俊平. 统计学原理. 北京: 中国商务出版社, 2008.Liu Chun-Ying, Jia Jun-Ping. The Principles of Statistics. Beijing: China Commerce and Trade Press, 2008. [33] Huang G B, Zhu Q Y, Siew C K. Extreme learning machine: Theory and applications. Neurocomputing, 2006, 70(1-3): 489-501 doi: 10.1016/j.neucom.2005.12.126 [34] Frank A, Asuncion A. UCI machine learning repository [Online], available: http://archive.ics.uci.edu/ml, October 13, 2020 [35] Chang C C, Lin C J. LIBSVM: A library for support vector machines. ACM Transactions on Intelligent Systems and Technology, 2011, 2(3): Article No. 27 [36] Hsu C W, Chang C C, Lin C J. A practical guide to support vector classification [Online], available: https://www.ee.columbia.edu/~sfchang/course/spr/papers/svm-practical-guide.pdf, March 11, 2021 [37] Alon U, Barkai N, Notterman D A, Gish K, Ybarra S, Mack D, et al. Broad patterns of gene expression revealed by clustering analysis of tumor and normal colon tissues probed by oligonucleotide arrays. Proceedings of the National Academy of Sciences of the United States of America, 1999, 96(12): 6745-6750 doi: 10.1073/pnas.96.12.6745 [38] Singh D, Febbo P G, Ross K, Jackson D G, Manola J, Ladd C, et al. Gene expression correlates of clinical prostate cancer behavior. Cancer Cell, 2002, 1(2): 203-209 doi: 10.1016/S1535-6108(02)00030-2 [39] Tian E M, Zhan F H, Walker R, Rasmussen E, Ma Y P, Barlogie B, et al. The role of the Wnt-signaling antagonist DKK1 in the development of osteolytic lesions in multiple myeloma. The New England Journal of Medicine, 2003, 349(26): 2483-2494 doi: 10.1056/NEJMoa030847 [40] Wang G S, Hu N, Yang H H, Wang L M, Su H, Wang C Y, et al. Comparison of global gene expression of gastric cardia and noncardia cancers from a high-risk population in China. PLoS One, 2013, 8(5): Article No. e63826 doi: 10.1371/journal.pone.0063826 [41] Li W Q, Hu N, Burton V H, Yang H H, Su H, Conway C M, et al. PLCE1 mRNA and protein expression and survival of patients with esophageal squamous cell carcinoma and gastric adenocarcinoma. Cancer Epidemiology, Biomarkers & Prevention, 2014, 23(8): 1579-1588 [42] Khan J, Wei J S, Ringnér M, Saal L H, Ladanyi M, Westermann F, et al. Classification and diagnostic prediction of cancers using gene expression profiling and artificial neural networks. Nature Medicine, 2001, 7(6): 673-679 doi: 10.1038/89044 [43] Gao S Y, Steeg G V, Galstyan A. Variational information maximization for feature selection. In: Proceedings of the 30th International Conference on Neural Information Processing Systems. Barcelona, Spain: Curran Associates, 2016. 487−495 [44] Gao W F, Hu L, Zhang P, He J L. Feature selection considering the composition of feature relevancy. Pattern Recognition Letters, 2018, 112: 70-74 doi: 10.1016/j.patrec.2018.06.005 [45] 谢娟英, 丁丽娟, 王明钊. 基于谱聚类的无监督特征选择算法. 软件学报, 2020, 31(4): 1009-1024Xie Juan-Ying, Ding Li-Juan, Wang Ming-Zhao. Spectral clustering based unsupervised feature selection algorithms. Journal of Software, 2020, 31(4): 1009-1024 [46] Muschelli III J. ROC and AUC with a binary predictor: A potentially misleading metric. Journal of Classification, 2020, 37(3): 696-708 doi: 10.1007/s00357-019-09345-1 [47] Fawcett T. An introduction to ROC analysis. Pattern Recognition Letters, 2006, 27(8): 861-874 doi: 10.1016/j.patrec.2005.10.010 [48] Bowers A J, Zhou X L. Receiver operating characteristic (ROC) area under the curve (AUC): A diagnostic measure for evaluating the accuracy of predictors of education outcomes. Journal of Education for Students Placed at Risk (JESPAR), 2019, 24(1): 20-46 doi: 10.1080/10824669.2018.1523734 [49] 卢绍文, 温乙鑫. 基于图像与电流特征的电熔镁炉欠烧工况半监督分类方法. 自动化学报, 2021, 47(4): 891-902Lu Shso-Wen, Wen Yi-Xin. Semi-supervised classification of semi-molten working condition of fused magnesium furnace based on image and current features. Acta Automatica Sinica, 2021, 47(4): 891-902 [50] Xie J Y, Gao H C, Xie W X, Liu X H, Grant P W. Robust clustering by detecting density peaks and assigning points based on fuzzy weighted K-nearest neighbors. Information Sciences, 2016, 354: 19-40 doi: 10.1016/j.ins.2016.03.011 [51] 谢娟英, 吴肇中, 郑清泉. 基于信息增益与皮尔森相关系数的2D自适应特征选择算法. 陕西师范大学学报(自然科学版), 2020, 48(6): 69-81Xie Juan-Ying, Wu Zhao-Zhong, Zheng Qing-Quan. An adaptive 2D feature selection algorithm based on information gain and pearson correlation coefficient. Shaanxi Normal University (Natural Science Edition), 2020, 48(6): 69-81 期刊类型引用(2)
1. 孙世政,庞珂,于竞童,陈仁祥. 基于白鲨优化极限学习机的三维力传感器非线性解耦. 光学精密工程. 2023(18): 2664-2674 .  百度学术
百度学术2. 谭敏刚,张潮海,陈斌. 高灵敏度和高区分度电压暂降能量指标研究. 控制理论与应用. 2022(03): 411-420 .  百度学术
百度学术其他类型引用(4)
-




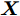
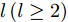
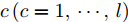
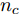

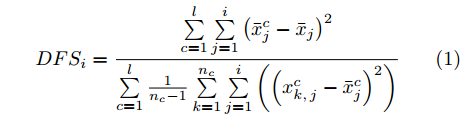
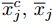

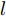
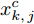
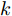
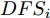
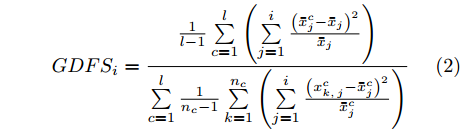
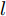

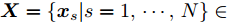
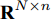
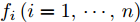
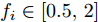
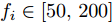
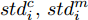
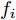
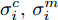
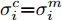
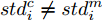
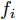
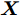
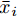
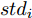
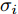
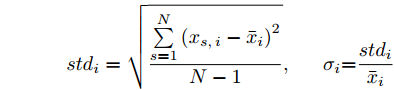
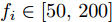
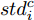
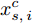
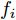
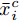
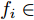
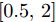
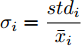
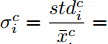
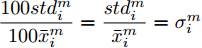
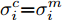
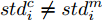
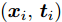
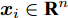
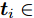
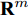
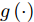
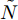
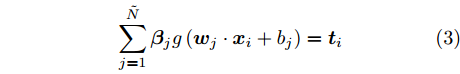
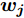
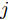
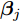
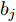
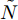
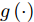
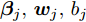
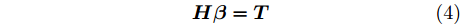
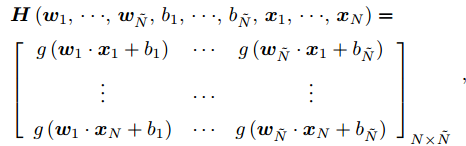
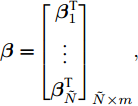
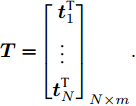
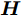
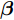
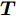
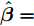
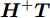
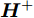
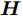
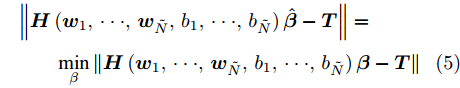
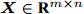
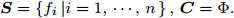

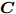
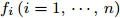

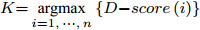
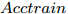
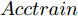
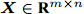
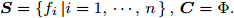

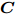
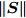
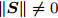
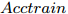
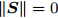
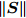
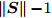
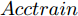
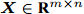
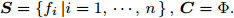

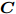
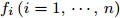

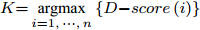
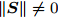
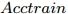

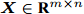
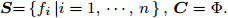

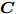
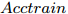
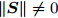
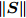
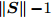

 下载:
下载:

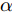
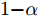
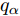
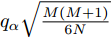
 下载:
下载:
