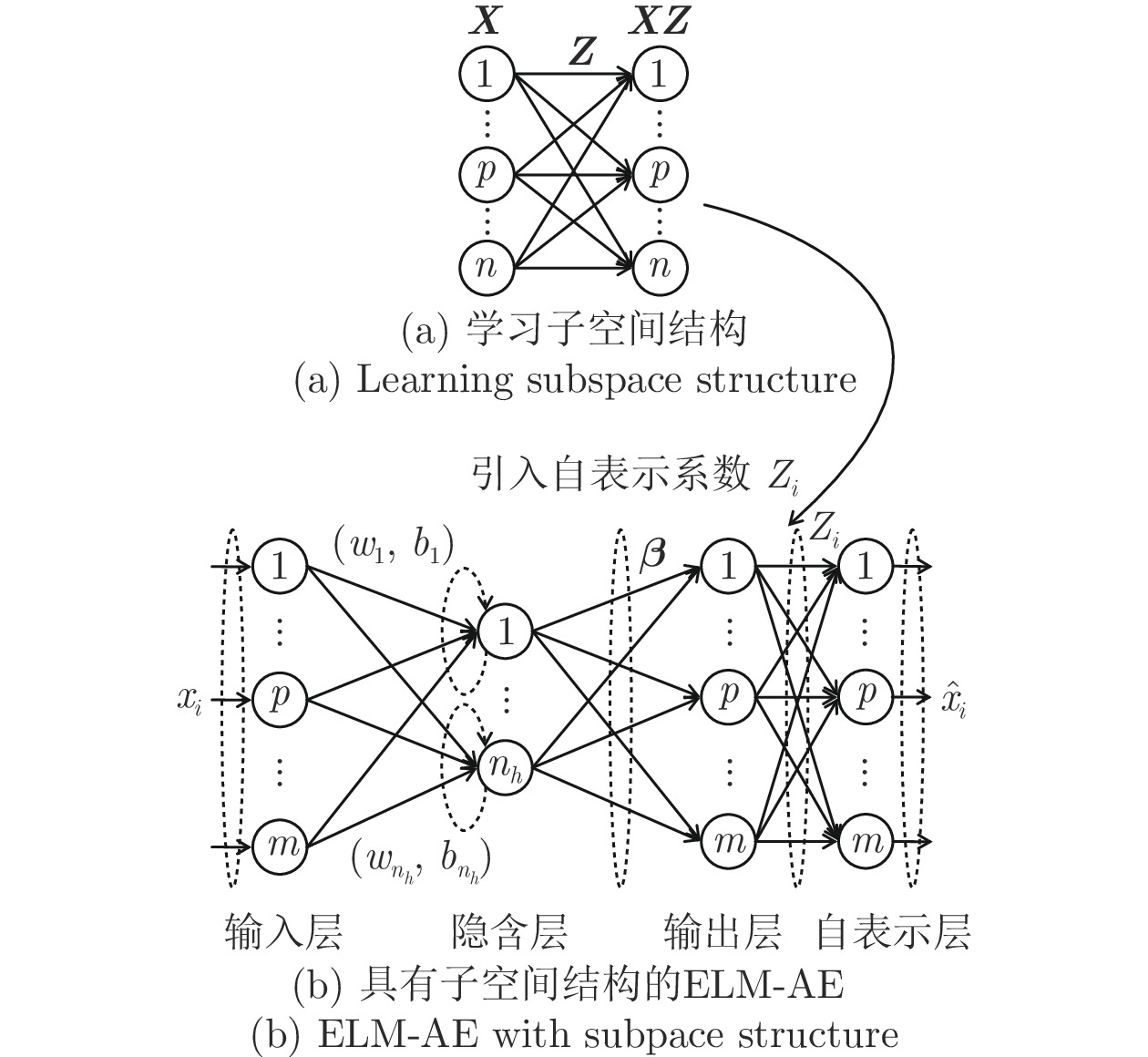
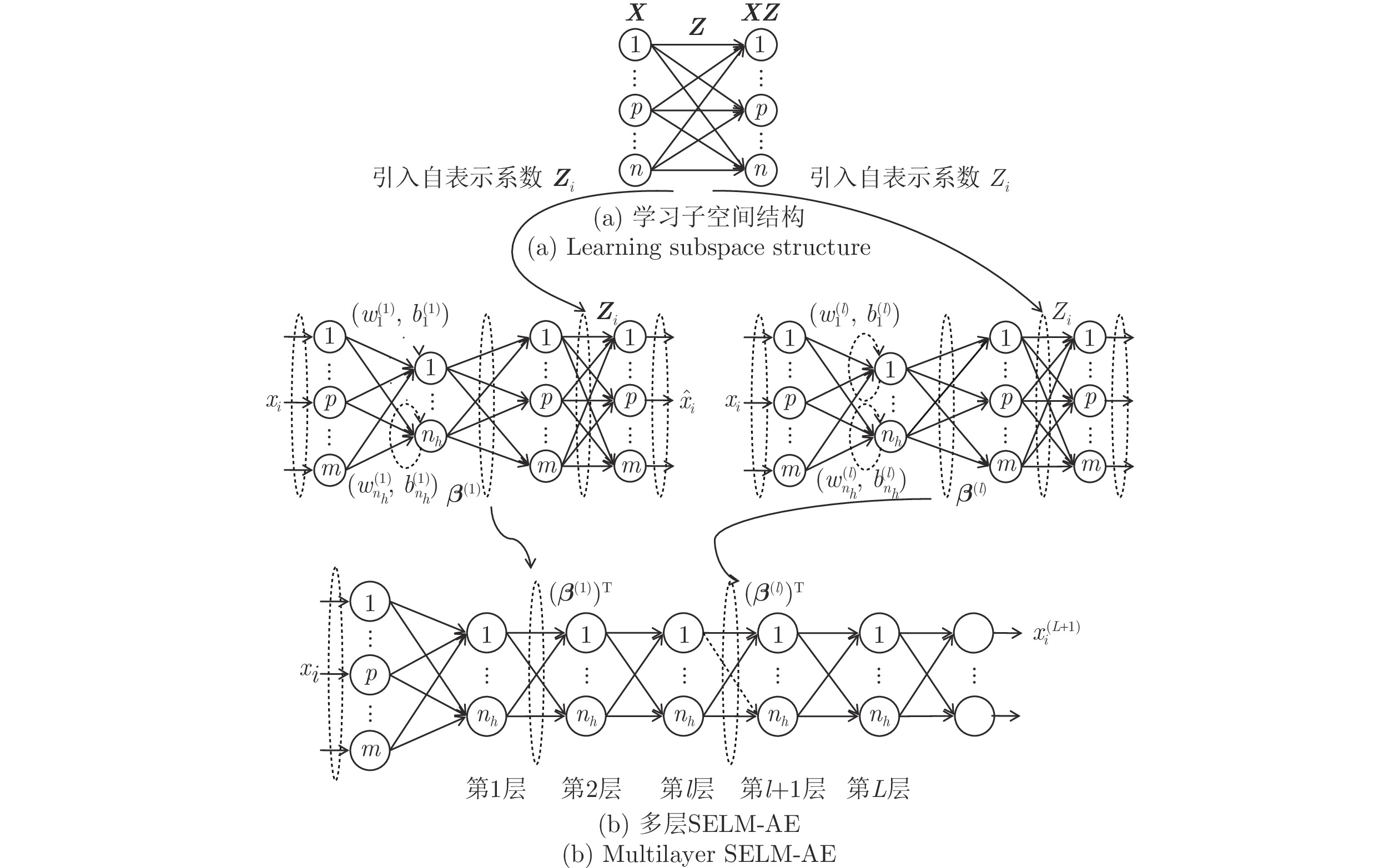
Multi-layer Extreme Learning Machine Autoencoder With Subspace Structure Preserving
-
摘要: 处理高维复杂数据的聚类问题, 通常需先降维后聚类, 但常用的降维方法未考虑数据的同类聚集性和样本间相关关系, 难以保证降维方法与聚类算法相匹配, 从而导致聚类信息损失. 非线性无监督降维方法极限学习机自编码器(Extreme learning machine, ELM-AE)因其学习速度快、泛化性能好, 近年来被广泛应用于降维及去噪. 为使高维数据投影至低维空间后仍能保持原有子空间结构, 提出基于子空间结构保持的多层极限学习机自编码器降维方法(Multilayer extreme learning machine autoencoder based on subspace structure preserving, ML-SELM-AE). 该方法在保持聚类样本多子空间结构的同时, 利用多层极限学习机自编码器捕获样本集的深层特征. 实验结果表明, 该方法在UCI数据、脑电数据和基因表达谱数据上可以有效提高聚类准确率且取得较高的学习效率.Abstract: To deal with the clustering problem of high-dimensional complex data, it is usually reguired to reduce the dimensionality and then cluster, but the common dimensional reduction method does not consider the clustering characteristic of the data and the correlation between the samples, so it is difficult to ensure that the dimensional reduction method matches the clustering algorithm, which leads to the loss of clustering information. The nonlinear unsupervised dimensionality reduction method extreme learning machine autoencoder (ELM-AE) has been widely used in dimensionality reduction and denoising in recent years because of its fast learning speed and good generalization performance. In order to maintain the original subspace structure when high-dimensional data is projected into a low-dimensional space, the dimensional reduction method ML-SELM-AE is proposed. This method captures the deep features of the sample set by using the multi-layer extreme learning machine autoencoder while maintaining multi-subspace structure of clustered samples by self-representation model. Experimental results show that the method can effectively improve the clustering accuracy and achieve higher learning efficiency on UCI data, EEG data and gene expression data.
-
航空母舰搭载投放的舰载机是航母战斗群的核心攻防力量. 航母舰载机的着舰作业直接决定了航母舰载机的出动回收能力, 而舰载机的出动回收能力是影响整个航母编队作战效能的核心要素. 航母舰载机的着舰作业不同于陆上机场着陆, 需要借助拦阻系统进行拉制减速[1]. 现代中、大型航母一般设置有4道拦阻索, 第1道拦阻索位于距舰尾约50 m处, 每道拦阻索之间相隔12 ~ 18 m. 飞行员在着舰前放下尾钩, 着舰后驾驶舰载机滑行, 使尾钩在甲板上拖行. 理想状态下, 舰载机尾钩、主起落架三点同时着舰, 尾钩垂直钩住第2道或第3道拦阻索的中间区域. 然而海面上风、浪、流等环境因素造成航母航空甲板进行复杂不规则运动, 而甲板的运动又不可避免地导致舰载机的理想着舰点成为三维空间中的活动点, 影响舰载机着舰的精度, 增加着舰作业的难度, 威胁舰载机着舰的安全性. 仿真结果表明, 当理想着舰点上升或下沉幅度大于0.8 m时极易造成着舰失败或撞舰事故[2]. 为消除航母甲板运动对舰载机着舰的不利影响, 当舰载机接近触舰时, 应进行甲板运动预估(Deck motion prediction), 可将甲板运动信息提前引入着舰引导律信息中. 航母甲板运动预估是舰载机自动着舰系统的重要功能之一, 也是航母进行舰机安全协同作业的关键技术之一[3]. 根据甲板运动预估的结果, 舰载机能够跟踪甲板运动, 及时进行航迹纠偏, 减小着舰误差, 提高着舰安全性[4].
1. 问题定义
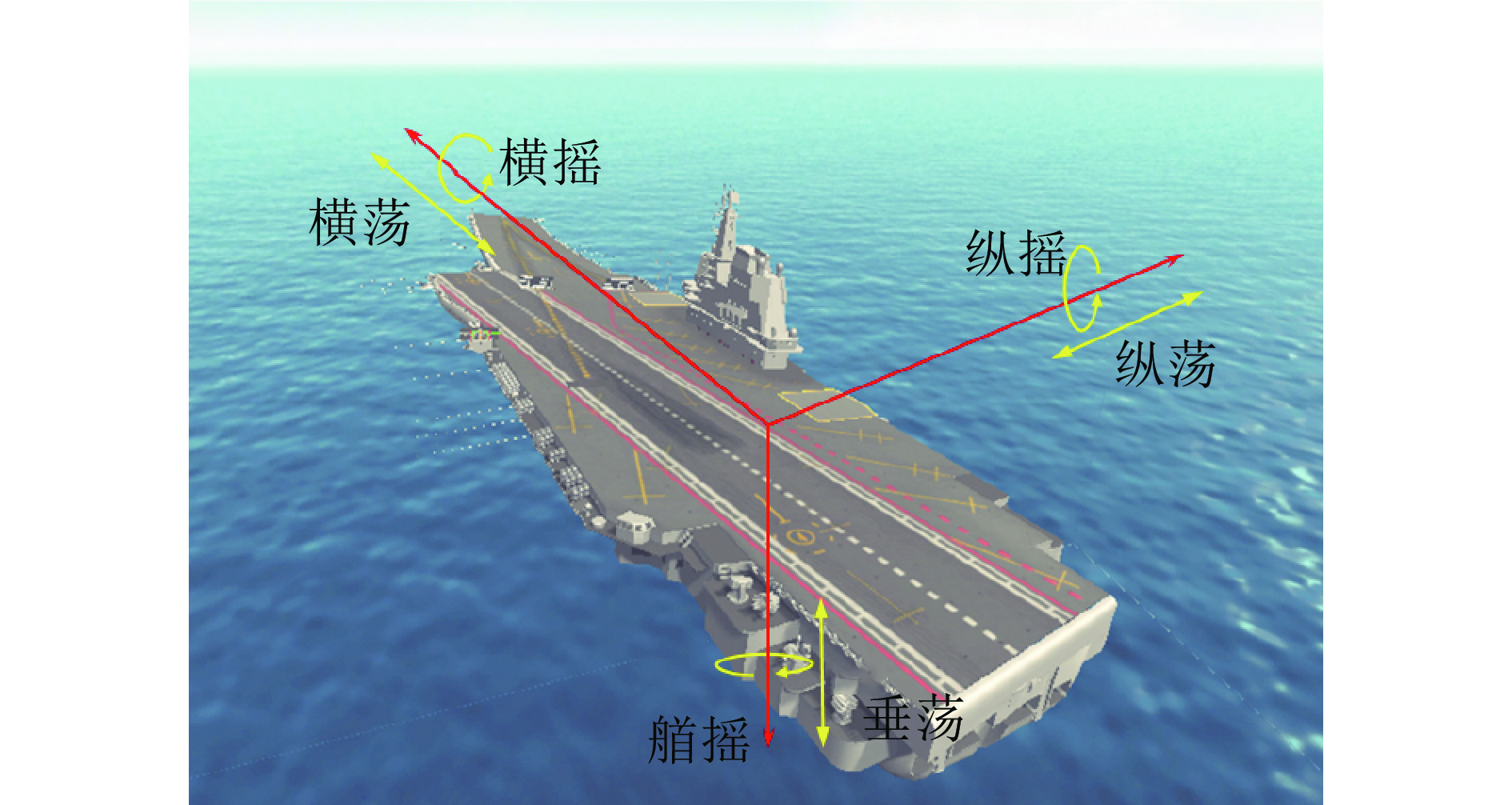
舰船在海上航行和作业过程中, 受到海风、海浪、洋流等因素的影响, 船体会产生平移运动和摇荡运动[5]. 船体的平移运动包括横荡(Sway)、纵荡(Surge)和垂荡(Heave), 摇荡运动包括横摇(Roll)、纵摇(Pitch)和艏摇(Yaw), 共计6个自由度. 如图1所示, 横荡、纵荡和垂荡为沿三个坐标轴的直线运动, 横摇、纵摇和艏摇为绕三个坐标轴的旋转运动. 描述甲板平移运动时通常以“米 (m)”为单位, 描述甲板摇荡运动时通常以“度 (°)”为单位. 甲板横向运动和纵向运动之间的耦合影响很小, 从工程应用的角度研究甲板运动时, 一般在保证模型能够描述运动主要特性的基础上, 把不同状态的运动视为彼此独立的过程. 从机器学习的视角, 可将甲板运动预估问题定义如下:
对于舰船在任意一个自由度上的运动, 假设在$ t $时刻已获得的历史运动状态可表示为 ${\boldsymbol{X}}(t) = \left[ {x(t),x(t - 1),\cdots,x(t - T + 1)} \right]$, 其中, $( t - T + 1 \sim t)$代表历史状态的时间窗口, $ T $为窗口内采样数据点的个数, $ x(t) $为$ t $时刻的描述运动状态的标量值. 从$ t $时刻起, 在接下来$ \left( {t + 1 \sim t + M} \right) $的时间窗口内, 甲板的运动状态可表示为${\boldsymbol{Y}}\left( t \right) = [ y\left( {t + 1} \right), y( t \;+ 2 ), \cdots ,y\left( {t + M} \right) ]$, 其中, $ y(t) $为$ t $时刻的描述运动状态的标量值, $M$为预估时间窗口的大小. 甲板运动预估的目标是找到一个函数$f:{\boldsymbol{X}}\left( t \right) \to {\boldsymbol{Y}}^\prime{\left( t \right) }$, 根据历史运动状态${\boldsymbol{X}}\left( t \right)$预估下一个时间窗口内的运动状态${\boldsymbol{Y}}^\prime{\left( t \right) }$. 损失函数可定义为
$$ L = \frac{1}{M}\sum\limits_{i = 1}^M {\left\| {{\boldsymbol{Y}}\left( {t + i} \right) - {\boldsymbol{Y}}^\prime{{\left( {t + i} \right)}}} \right\|} _2^2 $$ (1) 其中, ${\boldsymbol{Y}}^\prime{\left( {{t_i}} \right)} = \left[ {y^\prime{{\left( {{t_i} + 1} \right)} },y^\prime{{\left( {{t_i} + 2} \right)} }, \cdots ,y^\prime{{\left( {{t_i} + M} \right)} }} \right]$为$ \left( {{t_i} + 1\sim {t_i} + M} \right) $时间内的运动状态预估结果. 函数$f$即为甲板运动预估模型.
2. 相关工作
目前甲板运动预估主要包括基于物理建模的方法和基于机器学习的方法. 在基于物理建模的方法中, 甲板的运动模型包括基于正弦波组合的运动模型、基于功率谱的运动模型和基于Conolly线性理论的运动模型[4]. 基于正弦波组合的运动模型将甲板运动合理假设为具有窄频带的平稳随机过程, 理论上可建模为不同的正弦波的叠加. 基于功率谱的运动模型将一定航速、航向和海情条件下运动的舰船视为一个线性定常系统, 可通过大量仿真实验得到功率谱密度函数, 并据此计算舰船运动成形滤波器的传递函数, 再通过成形滤波器得到舰船的运动状态. 基于Conolly线性理论的运动模型假设海浪具有各态历经性且海浪的波能谱满足 ITTC (International Towing Tank Conference) 单参数谱. 舰船在航行时, 可以把反映扰动力矩的波倾角(波浪表面与水平面间的夹角)信号作为舰船横滚和俯仰运动的输入信号, 把反映扰动力的波高信号作为舰船升沉运动的输入信号, 由能量法和海浪理论得到舰船的运动模型[6]. 基于物理建模的方法理论基础完备, 但对海况海情等条件有较为严格的要求, 通过仿真实验难以完全模拟复杂、多变的航行条件, 因此模型通用性不强, 尤其是在特殊环境下难以保证运动模型的有效性. 与基于物理建模的方法不同, 基于机器学习的方法是数据驱动的方法, 不需要建立舰船运动的理论模型, 而是根据当前及历史运动状态数据对未来的运动状态进行短周期预估. 据公开披露的文献中的报道, 这类方法主要包括: 1)时间序列分析法. 例如张永花等[6]、周鑫等[7]在设计的甲板运动补偿器中利用自回归模型进行甲板运动预估; Xue等[8]在无人机自动着舰系统中采用基于自回归的甲板运动预估方法. 2)基于神经网络的方法. 例如Nicolau等[9]、Liu等[10]、Li等[11]提出的基于神经网络的舰船运动状态预估模型. 3)基于滤波技术的方法. Sidar等[12]早在1983年就验证了卡尔曼滤波理论在舰船升沉运动预估中的有效性. 在后续的相关工作中, 卡尔曼滤波法常用于甲板运动的预估[13-14]; Zhen等[15-16]在其设计的自动着舰系统中采用基于粒子滤波的甲板运动预估方法. 除上述方法外, 还有一些其他方法, 例如伏尔特拉级数模型[17]、基于灰色系统理论(Grey system theory)的方法[18]等. 上述基于机器学习的方法在特定的场景和约束条件下能够取得良好的预估效果, 但存在如下局限性: 1)时间序列分析法适合于较短周期的预估, 预估精度受预估周期的影响较大. 2)真实海况海情往往不能满足卡尔曼滤波法关于模型线性和噪声约束. 3)早期的运动状态预估神经网络架构通常较为简单, 学习能力不强; 较复杂的网络架构又缺乏针对特殊应用场景的有效设计原则, 且训练效率低、调参难度大, 缺乏应对真实环境中噪声和非预知干扰因素的鲁棒性. 针对上述局限性, 本文提出一种面向甲板运动预估的鲁棒神经网络模型, 该模型能实现由数据驱动的自适应动态构建, 采用引入了鲁棒性约束的非梯度快速学习算法, 提高模型的训练效率和鲁棒性.
3. 甲板运动预估模型
本文提出一种面向甲板运动预估的鲁棒学习模型, 通过基本的学习器动态演化, 构建出子网络, 再将子网络作为更复杂的学习器, 演化出规模更大的学习系统. 学习器的训练采用非梯度的伪逆学习策略, 简化了学习控制超参数调优; 学习器的架构设计采用数据驱动的策略, 简化了架构超参数调优; 采用图拉普拉斯正则化方法, 提高了模型对噪声和非预知干扰因素的鲁棒性.
3.1 伪逆学习自编码器
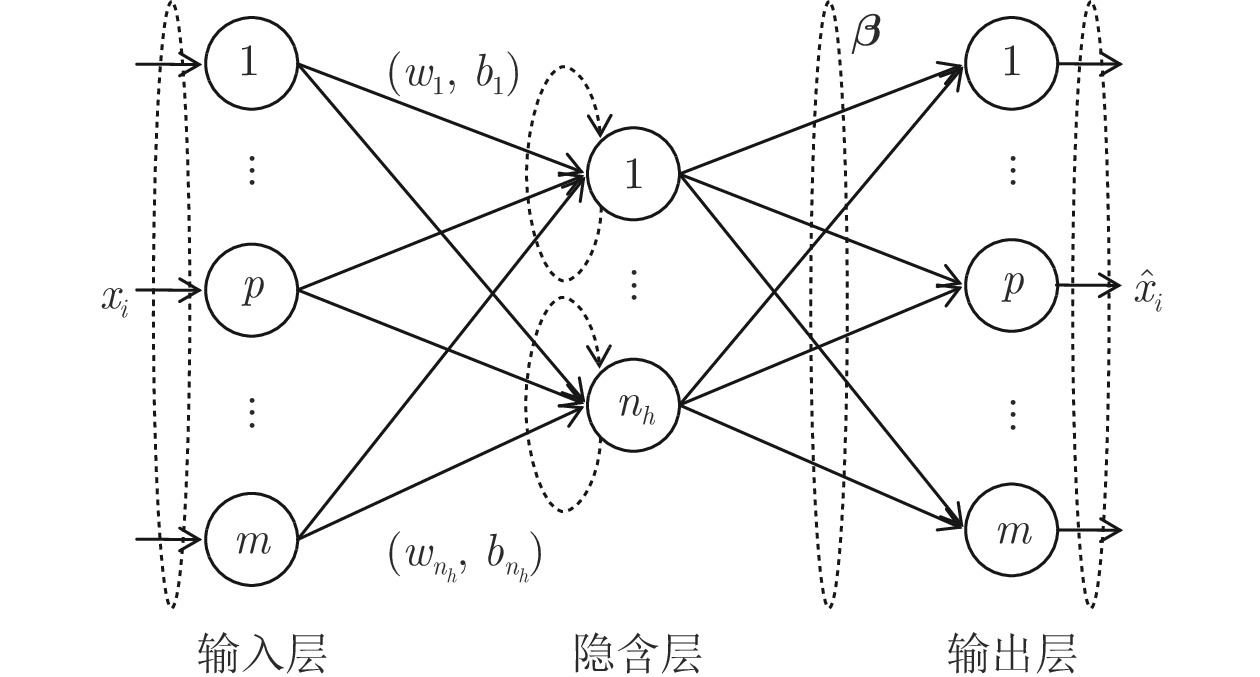
本文所提预估模型的基本学习器具体实现为伪逆学习自编码器(Pseuodoinverse learning based autoencoder, PILAE)[19-21]. 伪逆学习自编码器本质上是一种单隐层前馈神经网络, 其采用的伪逆学习算法[22]是一种训练单隐层前馈神经网络的有监督学习算法, 基本思想是找到一组正交向量基将输入数据映射到隐层特征空间, 并利用非线性激活函数使得隐层神经元的输出向量趋向正交, 然后通过计算伪逆得到网络的输出权重的最优近似解.
假设在一个有监督的学习问题中, 由$ N $个样本构成的训练集记为$D = \left\{ {{{\boldsymbol{x}}^i},{{\boldsymbol{o}}^i}} \right\}_{i = 1}^N$, 其中 ${{\boldsymbol{x}}^i} = ( {x_1}, {x_2},\cdots, {x_d} )^{\text{T}}\in {{\bf{R}}^d}$为第$ i $个$ d $维的训练样本, 与其对应的输出标签为${{\boldsymbol{o}}^i} = {\left( {{o_1},{o_2},\cdots,{o_m}} \right)^{\text{T}}} \in {{\bf{R}}^m}$. 以矩阵$ {\boldsymbol{X}} $表示训练数据, ${\boldsymbol{X}} = \left[ {{{\boldsymbol{x}}^1},{{\boldsymbol{x}}^2},\cdots,{{\boldsymbol{x}}^N}} \right] \in {{\bf{R}}^{d \times N}}$, 以矩阵$ {\boldsymbol{O}} $表示监督信息(自编码器中${\boldsymbol{O = X}}$), ${\boldsymbol{O}} = \left[ {{{\boldsymbol{o}}^1},{{\boldsymbol{o}}^2},\cdots,{{\boldsymbol{o}}^N}} \right] \in {{\bf{R}}^{m \times N}}.$ 对误差函数具有权重衰减正则化形式的单隐层网络, 不失一般性地假设网络输出权重矩阵$ {{\boldsymbol{W}}_o} $符合标准差为$ \sigma $的正态分布, 则训练网络的优化目标可定义为
$$ \begin{split} J\left( {{{\boldsymbol{W}}_o}} \right) =\;& - \lg P\left( {{{\boldsymbol{W}}_o}|{\boldsymbol{X}}} \right)=- ( \lg P\left( {{\boldsymbol{X}}{\text{|}}{{\boldsymbol{W}}_o}} \right) \,+ \\ &\lg P\left( {{{\boldsymbol{W}}_o}} \right) - \lg P\left( {\boldsymbol{X}} \right) ) \end{split}$$ (2) 根据平均场和变分推理理论, 假设数据集${\boldsymbol{X}}\sim {\rm N}\left( {\mu ,1} \right)$, 则有
$$ \lg P({{\boldsymbol{W}}_o}) \approx - \frac{1}{{2{\sigma ^2}}}\sum\limits_i^{} {{{\left( {{\boldsymbol{W}}_o^{\left( i \right)}} \right)}^2}} $$ (3) $$ \lg P\left( {{{\boldsymbol{W}}_o}{\text{|}}{\boldsymbol{X}}} \right) \approx \frac{1}{2}\sum\limits_i^{} {{{\left( {{{\boldsymbol{W}}_o}\partial \left( {{{\boldsymbol{W}}_i}{{\boldsymbol{X}}_i}} \right) - {{\boldsymbol{X}}_i}} \right)}^2}} $$ (4) 联合式(2) ~ (4)可得到优化目标的矩阵形式为
$$ J\left( {\boldsymbol{W}} \right) = \frac{1}{2}\left\| {{{\boldsymbol{W}}_o}\partial \left( {{{\boldsymbol{W}}_i}{\boldsymbol{X}}} \right) - {\boldsymbol{O}}} \right\|_2^2 + \frac{\lambda }{2}\left\| {\boldsymbol{W}} \right\|_2^2 $$ (5) 式中, $ {\boldsymbol{W}} $为网络的连接权重矩阵, 包括输入权重和输出权重, 其中$ {{\boldsymbol{W}}_i} $代表输入权重矩阵, $ {{\boldsymbol{W}}_o} $代表输出权重矩阵, $\partial \left( \cdot \right)$为激活函数, $ \lambda $为权重衰减正则化($ {L_2} $范数)项系数. 伪逆学习算法中使用预设的初始输入权重矩阵$ {{\boldsymbol{W}}_i} $, 在具体实现上, 可通过具有低秩约束的截断奇异值分解(Truncated singular value decomposition, TSVD)得到[19-20]. 根据伪逆学习的基本思想, 目标函数的最优近似解析解作为输出权重, 即
$${{\boldsymbol{W}}_o} = {\boldsymbol{O}}\partial {\left( {{{\boldsymbol{W}}_i}{\boldsymbol{X}}} \right)^{\rm{T}}}{\left( {\partial \left( {{{\boldsymbol{W}}_i}{\boldsymbol{X}}} \right)\partial {{\left( {{{\boldsymbol{W}}_i}{\boldsymbol{X}}} \right)}^{\rm{T}}} + \lambda {\boldsymbol{I}}} \right)^{ - 1}} $$ (6) 最后通过权重捆绑, 将输出权重$ {{\boldsymbol{W}}_o} $的转置作为最终的输入权重, 即${\boldsymbol W}'_i = {\boldsymbol{W}}_o^{\text{T}}$. 训练完成后, 通过前向传播实现特征抽取, 令输入数据$ {\boldsymbol{X}} $在隐层特征空间中的映射为矩阵$ {\boldsymbol{H}} $, 则${\boldsymbol{H}} = \partial ({{{\boldsymbol W}'_i}}{\boldsymbol{X}})$.
3.2 基于图拉普拉斯正则化的鲁棒性增强
为提高学习模型对真实、复杂环境中噪声和非预知干扰因素的鲁棒性, 除权重衰减正则化项之外, 本文在式(5)中引入雅克比正则化项[23]. 由于直接使用雅克比正则化会造成优化求解困难, 因此使用图拉普拉斯正则化对其进行近似[24-26]. 为此, 定义如下惩罚项:
$$ P = T\left( {{\boldsymbol{\hat XL}}{{{\boldsymbol{\hat X}}}^{\text{T}}}} \right) $$ (7) 其中, 函数$T\left( \cdot \right)$返回矩阵的秩, 矩阵${\boldsymbol{\hat X }}={{\boldsymbol{W}}_o}{\boldsymbol{H}}$为重构出的$ {\boldsymbol{X}} $, 矩阵$ {\boldsymbol{L}} = {\boldsymbol{D}} - {\boldsymbol{S}} $为拉普拉斯矩阵, 矩阵$ {\boldsymbol{S}} $为输入数据的相似度矩阵, 矩阵 $ {\boldsymbol{D}} $为按 $ {{\boldsymbol{D}}_{ii}} = \sum\nolimits_j^N {{s_{ij}}} $计算得到的对角阵. 式(5)定义的损失函数修改为
$$ \begin{split} J\left( {\boldsymbol{W}} \right) =\;& \frac{1}{2}\left\| {{{\boldsymbol{W}}_o}\partial \left( {{{\boldsymbol{W}}_i}{\boldsymbol{X}}} \right) - {\boldsymbol{O}}} \right\|_2^2 + \frac{\lambda }{2}\left\| {\boldsymbol{W}} \right\|_2^2 \,+ \\ &\eta T\left( {{{\boldsymbol{W}}_o}{\boldsymbol{HL}}{{\boldsymbol{H}}^{\text{T}}}{\boldsymbol{W}}_o^{\text{T}}} \right) \end{split}$$ (8) 其中, $\eta $为图拉普拉斯正则化项系数. 根据式(8)可求得输出权重的最优近似解为
$$ {{\boldsymbol{W}}_o} = {\boldsymbol{X}}{{\boldsymbol{H}}^{\rm{T}}}{\left( {{\boldsymbol{H}}{{\boldsymbol{H}}^{\rm{T}}} + \lambda {\boldsymbol{I}} + 2\eta {\boldsymbol{HL}}{\boldsymbol{H}^{\rm{T}}}} \right)^{ - 1}} $$ (9) 3.3 多隐层网络结构
使用伪逆学习自编码器作为基本学习器可以构建出多隐层网络. 具体而言, 采用逐层贪婪训练的策略, 逐层训练过的自编码器(去掉解码器)再堆叠为多层网络模型. 多层模型中前一层自编码器的隐层输出作为后一层自编码器的输入, 网络最后一层的输出可作为原始数据的高层抽象特征. 对于一个含有$l\,\,(l > 1)$个隐层的网络模型, 则有
$$ \begin{split} {{\boldsymbol{H}}^{\left( l \right)}} =\;& \partial \left( {{\left( {{\boldsymbol{W}}_o^{\left( {l - 1} \right)}} \right)}^{\text{T}}} \cdots \partial \left( {{\left( {{\boldsymbol{W}}_o^{\left( 2 \right)}} \right)}^{\text{T}}}\,\,\times\right.\right.\\ &\left.\left.\partial \left( {{{\left( {{\boldsymbol{W}}_o^{\left( 1 \right)}} \right)}^{\text{T}}}{\boldsymbol{X}}} \right) \right) \cdots \right)\end{split} $$ (10) 在甲板运动状态预估任务中, 可将输出的特征$ {{\boldsymbol{H}}^{\left( l \right)}} $作为回归器的输入. 回归器以有监督的方式进行训练.
构建多层学习模型时, 需要确定每个隐层的神经元个数与学习模型的层数(即深度)两个架构超参数. 对于隐层神经元个数, 原始伪逆学习算法将其设置为训练样本个数. 但原始伪逆学习算法针对的是小数据集上的精确学习, 而本文中伪逆学习自编码器的目标是表征学习. 隐层神经元的个数过小会造成模型学习能力不足, 重构误差过大; 隐层神经元的个数过大则容易导致过拟合. 为此, 本文根据训练数据的维度和秩, 约减隐层神经元个数的超参数搜索空间. 具体而言, 可将隐层神经元的个数设置为大于训练样本的秩而小于样本维度[19], 这是规避欠拟合与过拟合风险的一种折中策略. 训练集的秩可在计算初始输入权重矩阵${{\boldsymbol{W}}_i}$的过程中得到, 不会增加额外的计算代价. 对于学习模型的层数, 可以采用动态生长的方式自适应地确定, 即先构建一个单隐层的学习模型, 然后逐渐增加层数. 如果添加一个隐层可以提高模型在验证集上的性能则以较大的概率继续添加新的隐层, 否则以较大的概率终止增加新的隐层. 通过上述方法可以较容易地获得一组较优的架构超参数, 在实际问题中可根据实际需要以此为基线再进行人工调优. 与同类工作[27-28]中的模型架构设计过程相比, 上述策略能够约减架构超参数的搜索空间, 简化架构超参数的调优.
本文中多隐层网络用于从输入数据中学习高层特征. 在甲板运动预估问题中, 还需要在第$l$个隐层后添加一个回归器, 以多隐层网络学习到的特征作为输入, 以甲板运动状态的预测结果作为输出. 本文使用一个单隐层神经网络作为回归器. 该回归器同样可采用非梯度的伪逆学习算法以有监督的方式进行训练. 与伪逆学习自编码器的训练过程类似, 将式(9)中${\boldsymbol{X}}$替换为标记矩阵${\boldsymbol{O}} $, 得到回归器输出权重为
$$\begin{split} {\boldsymbol{W}}_o^{\left( r \right)} =\;& {\boldsymbol{{{O}}}}{\left( {{{\boldsymbol{H}}^{\left( r \right)}}} \right)^{\rm{T}}}\left( {{\boldsymbol{H}}^{\left( r \right)}}{{\left( {{{\boldsymbol{H}}^{\left( r \right)}}} \right)}^{\rm{T}}} \,+\right.\\ &\left.\lambda {\boldsymbol{I}} + 2\eta {{\boldsymbol{H}}^{\left( r \right)}}{{\boldsymbol{L}}^{\left( r \right)}}{{\left( {{{\boldsymbol{H}}^{\left( r \right)}}} \right)}^{\rm{T}}} \right)^{ - 1} \end{split}$$ (11) 其中, ${{\boldsymbol{H}}^{\left( r \right)}}$为回归器的隐层神经元输出矩阵, ${{\boldsymbol{L}}^{\left( r \right)}}$为根据${{\boldsymbol{H}}^{\left( r \right)}}$计算得到的拉普拉斯矩阵. 最终的甲板运动预估结果为${\boldsymbol{Y}} = {\boldsymbol{W}}_o^{\left( r \right)}{{\boldsymbol{H}}^{\left( r \right)}}$.
3.4 多网络集成学习系统
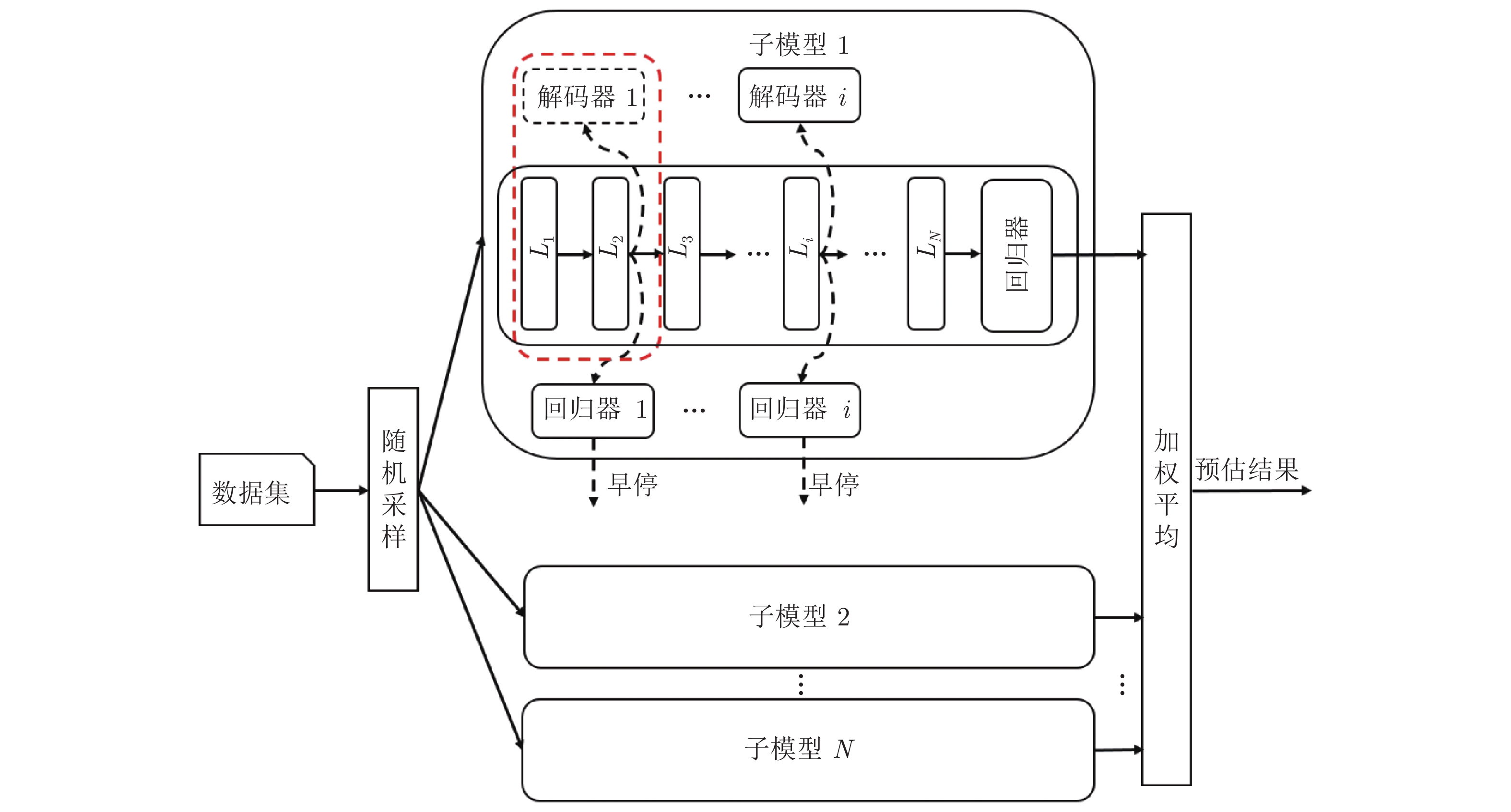
为进一步提升模型的预估性能, 本文由一系列独立训练的多层自编码器网络构建更复杂的集成学习系统, 其架构如图2 所示, 其中的每个子模型都是一个基于自编码器构建的多隐层网络. 图中虚线方框内为一个基本的伪逆学习自编码器, 作为整个学习系统的基本构建单元. 训练子模型时通过bootstrap采样不同的数据子集, 既可以减少训练耗时又可以保证子模型之间的差异性. 所有子模型的预估结果通过加权平均进行集成以纠正单独子模型的学习错误, 提高学习系统的泛化性能. 每个子模型的权重与其在验证集上的预估误差成反比. 构建集成学习系统时采用与构建一个多隐层网络类似的动态生长方式, 即先构建一个只含有一个子模型的学习系统, 然后逐个添加新的子模型. 如果添加新的子模型可以提高学习系统在验证集上的性能, 则以较大的概率继续添加新的子模型, 否则以较大的概率终止训练.
4. 实验验证
4.1 实验设置
甲板的运动一般可作为平稳随机过程处理, 本文在实验中采用工程中广泛使用的基于组合正弦波的甲板运动模型获取仿真数据, 再通过加入高斯随机噪声模拟航母甲板的运动过程. 仿真数据以某型航母30节 (kn)的典型航速巡航时, 在中等海况(有义波高1.25 ~ 2.5 m)条件下的场景为例, 选取与着舰安全密切相关的纵摇、横摇、垂荡三个自由度上的运动. 考虑到在着舰引导系统中, 如果能够提前10 ~ 13 s获取甲板运动状态并进行运动补偿, 则能够显著提升舰载机着舰安全性[3-4]. 因此, 实验中将运动状态预估窗口的大小设为15 s, 甲板历史运动状态采集窗口设定为50 s, 即通过过去50 s的甲板运动预估未来15 s的运动状态. 运动状态的采样频率为1 Hz, 仿真数据总时长为
2800 s, 其中前500 s数据作为训练集, 第501 ~1000 s的数据用作验证集, 第1001 ~2800 s的数据作为测试集. 实验中学习模型的权重衰减正则化项系数$\lambda $和图拉普拉斯正则化项系数$\eta $分别设置为0.001和0.0001 , 子模型训练集的采样率为0.7.4.2 实验结果与分析
4.2.1 鲁棒性分析
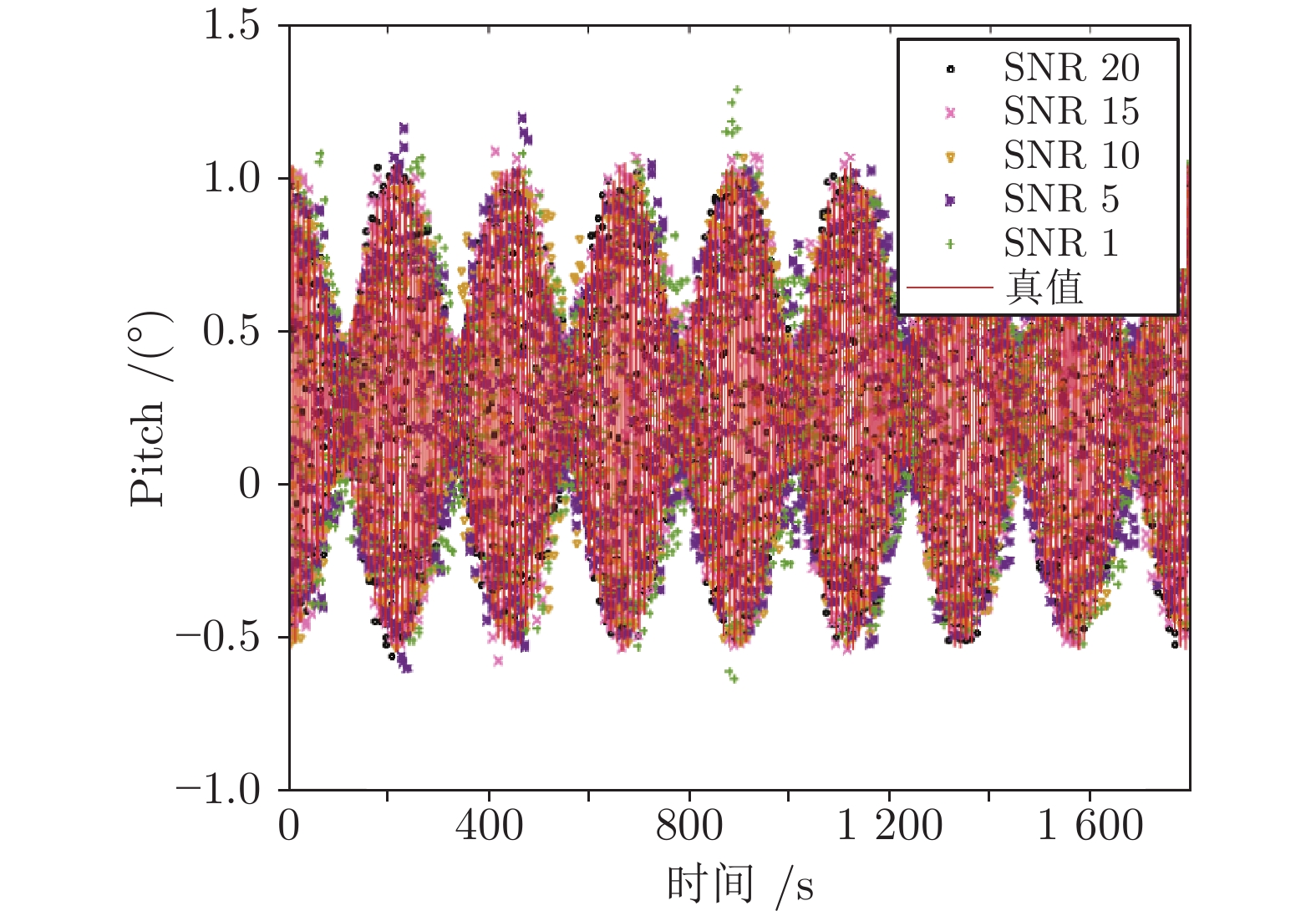
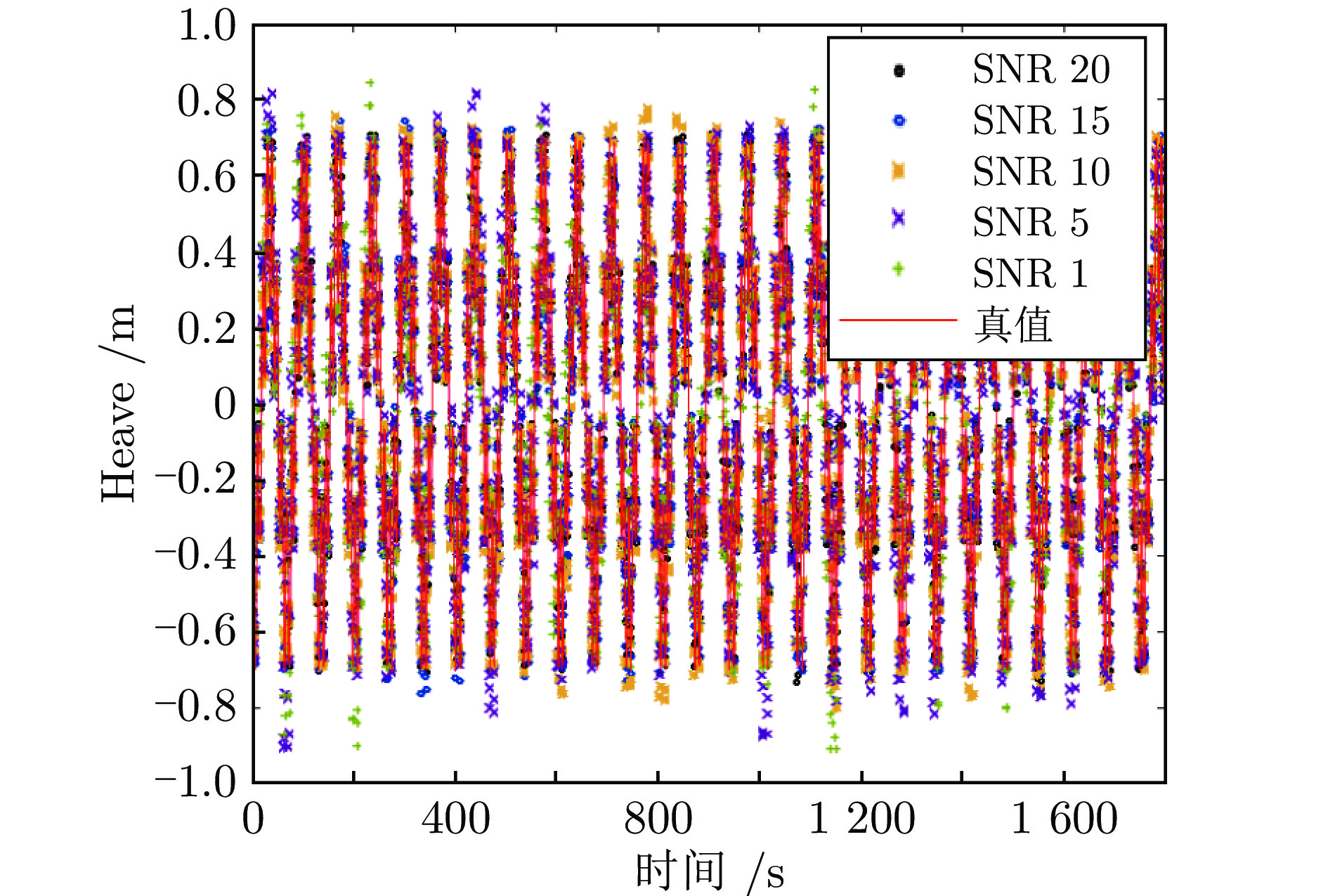
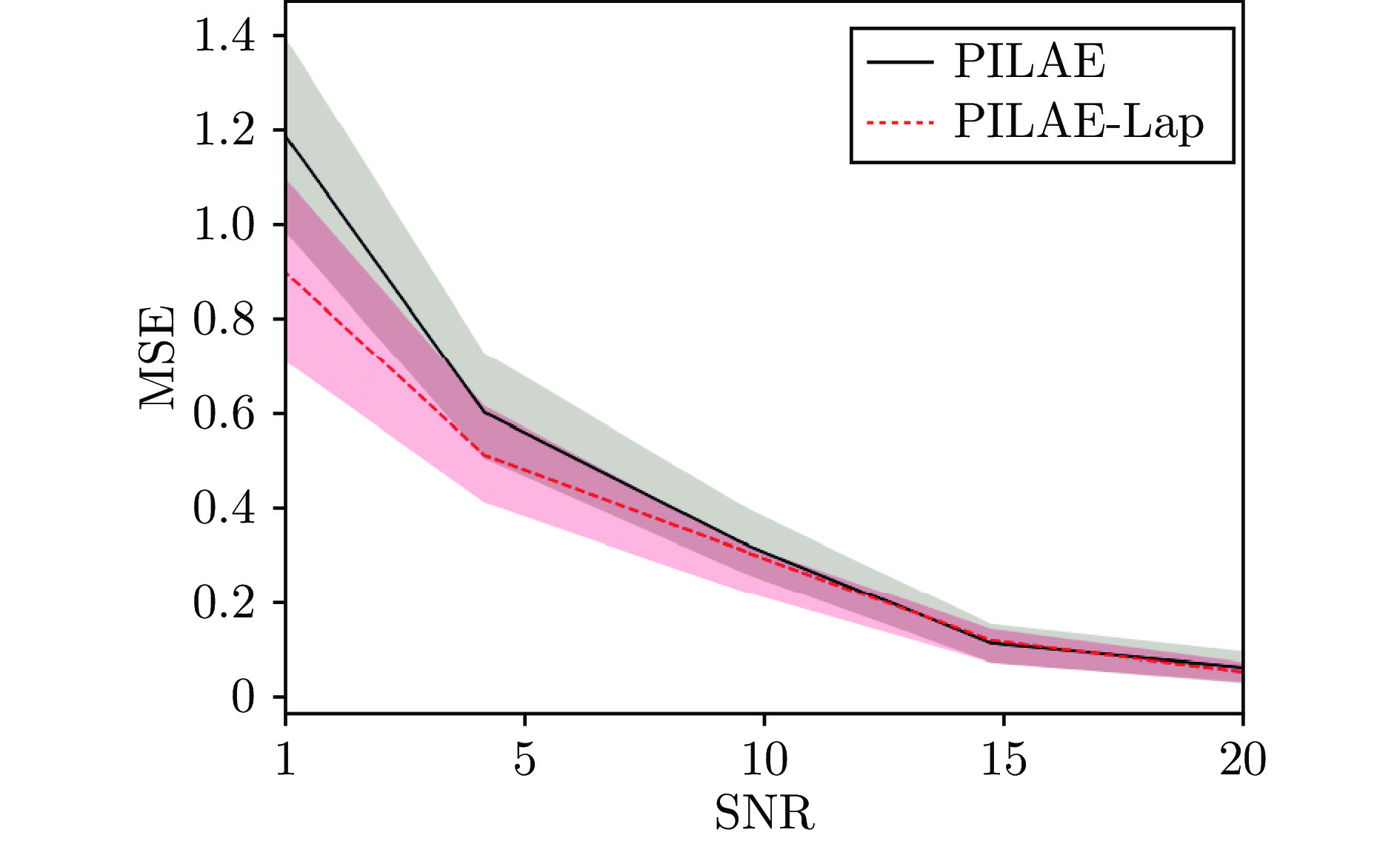
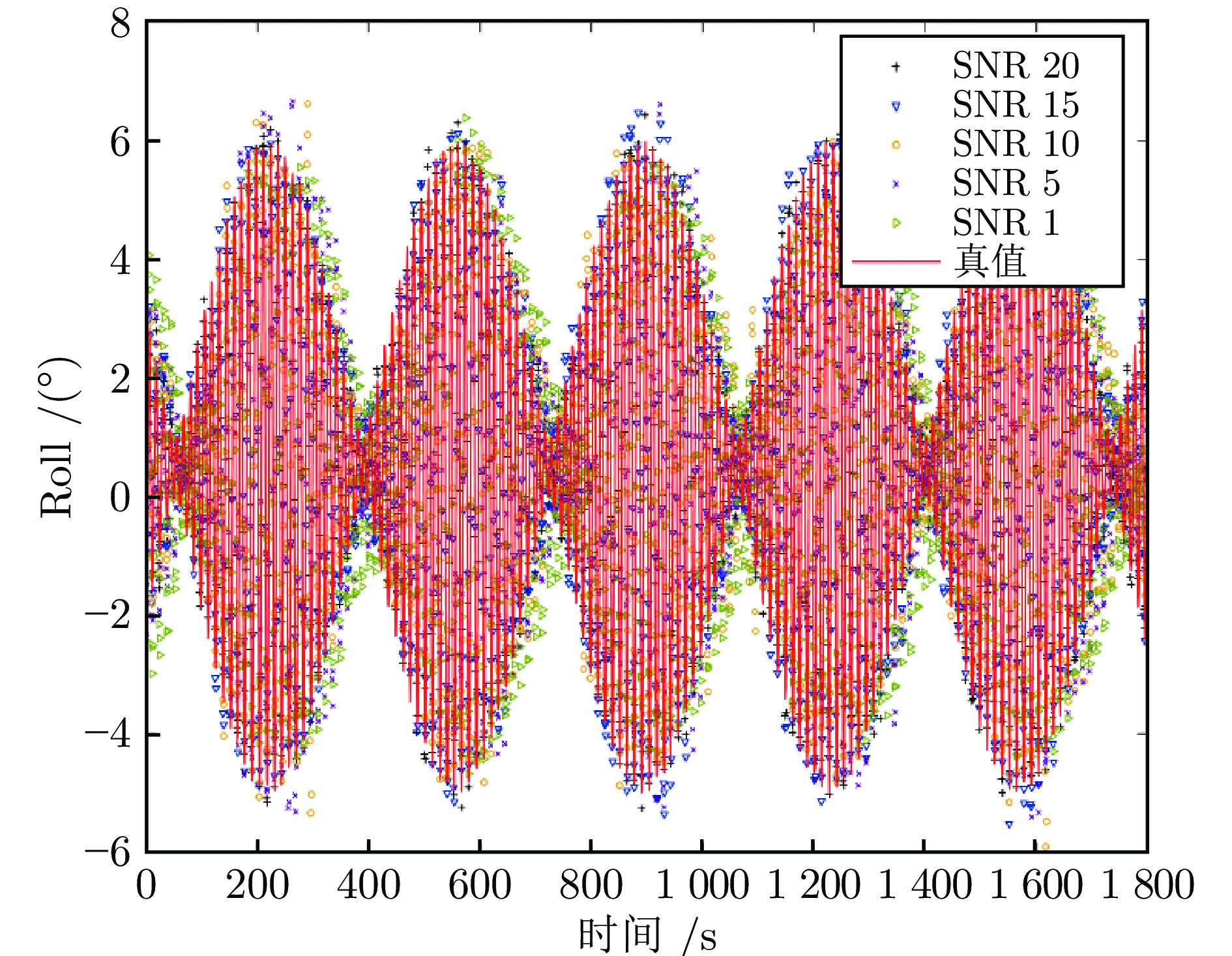
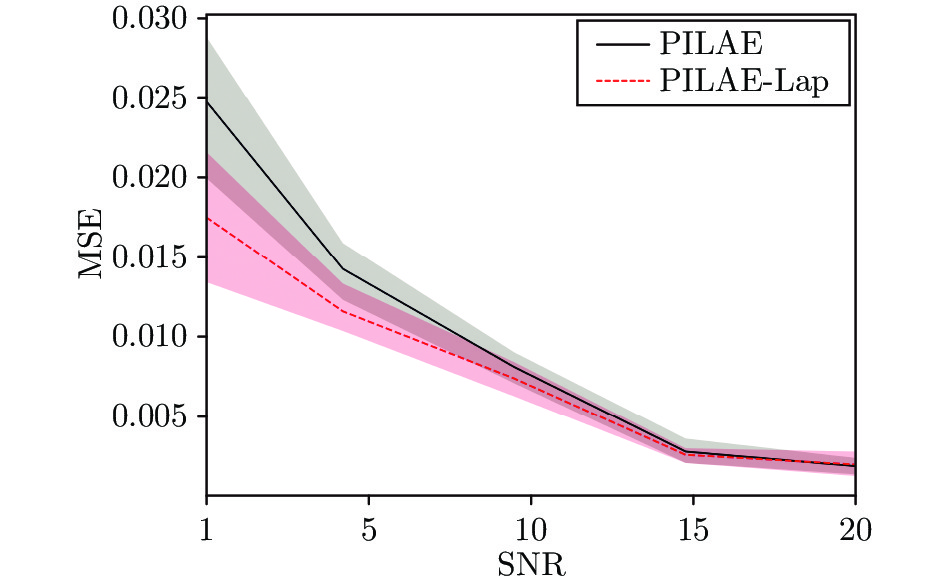
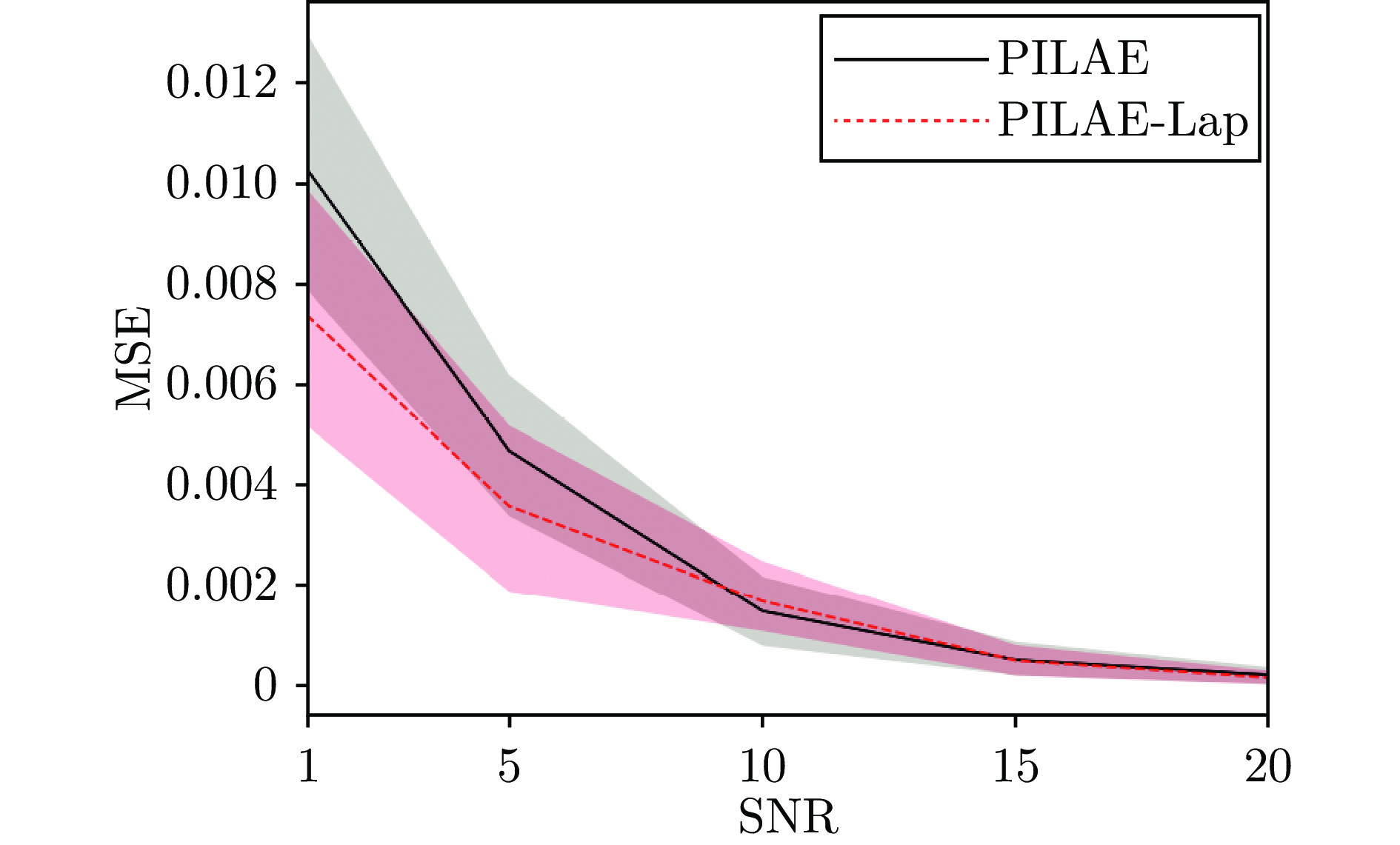
为了验证本文所提方法在不同噪声等级下的性能, 在仿真数据中加入白高斯噪声, 定义了5种不同的噪声等级, 信噪比(Signal to noise ratio, SNR)分别为20, 15, 10, 5, 1. 不同噪声等级下的甲板纵摇、横摇、垂荡运动的预估结果分别如图3 ~ 5所示. 从预估结果可以看出, 本文所提方法在不同的噪声等级下均能够得到较为理想的预估结果, 证明了本文模型在甲板运动预估问题中的有效性. 图6 ~ 8为关于鲁棒性的消融实验结果. 图中实线为PILAE模型在不同噪声等级下预估结果的均方误差(Mean square error, MSE), 虚线代表引入图拉普拉斯正则化项的伪逆学习自编码器(Pseuodoinverse learning based autoencoder with graph Laplace, PILAE-Lap). 从实验结果可以看出, 随着信噪比降低, 模型的预估性能整体呈下降趋势, 但通过引入图拉普拉斯正则化项能够抑制噪声对预估效果的负面影响, 尤其是当信噪比小于10时, 针对鲁棒性进行优化后的模型PILAE-Lap的预估误差明显小于PILAE模型, 证明了引入图拉普拉斯正则化项能够有效地提高甲板预估模型对噪声的鲁棒性.
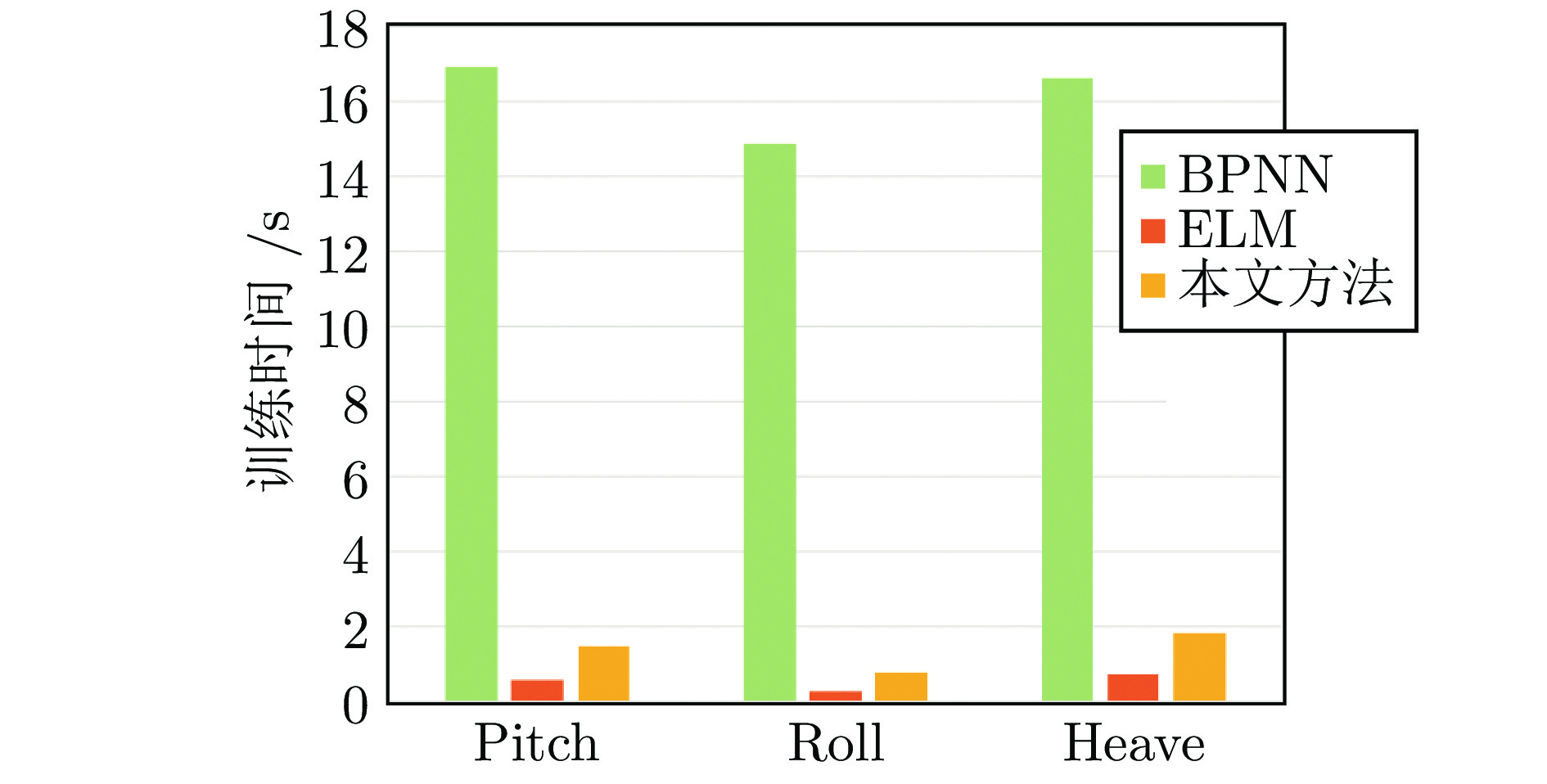
4.2.2 训练效率分析
为了评估本文所提模型的训练效率, 对比了其与基线模型的训练耗时. 实验中的基线方法包括反向传播神经网络(Back propagation neural network, BPNN)和极限学习机(Extreme learning machine, ELM)两种基于神经网络的方法, 其中ELM同样采用了类似的非梯度学习算法. 不同于基线方法, 本文方法能够自动确定网络架构, 因此实验中基线方法采用与本文方法完全相同的网络架构, 以保证对比实验的公平性. 实验结果如图9所示. 从对比实验结果可以看出, 本文模型与ELM的训练耗时与BPNN相比具有明显的优势, 证明了非梯度学习算法的效率优于基于梯度下降的学习算法. 与ELM相比, 本文模型的训练耗时略长, 这是由于本文模型采用具有低秩约束的截断奇异值分解初始化输入层与隐层间的连接权重, 而ELM采用随机连接权重. 虽然ELM训练速度较快, 但随机连接权重的有效性难以保证, 某些情况下会导致泛化性能不佳.
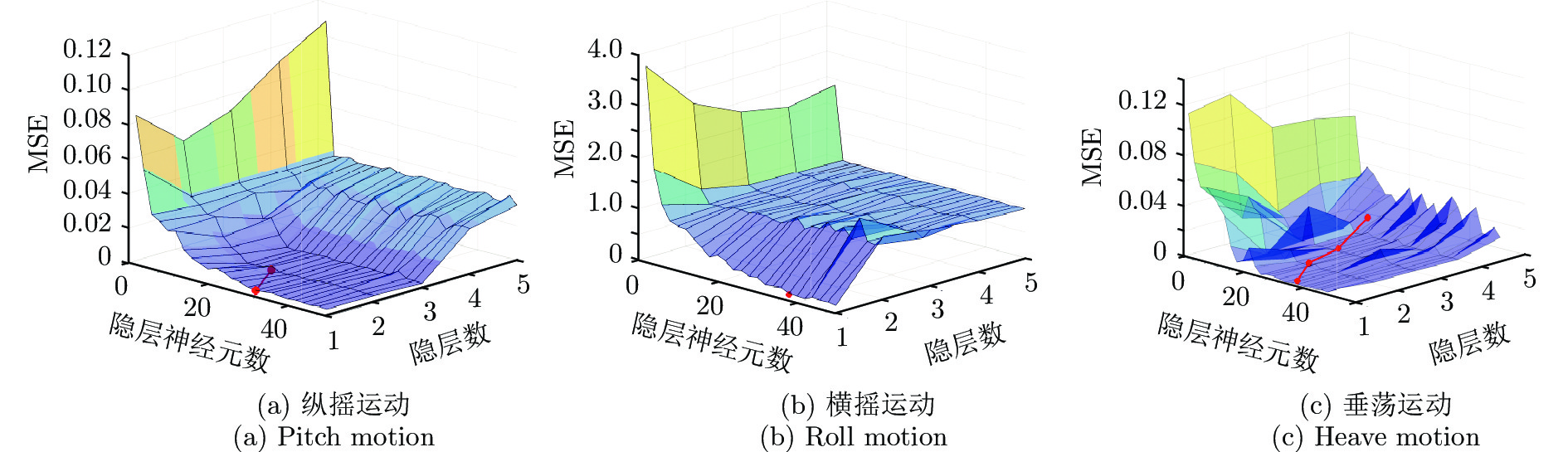
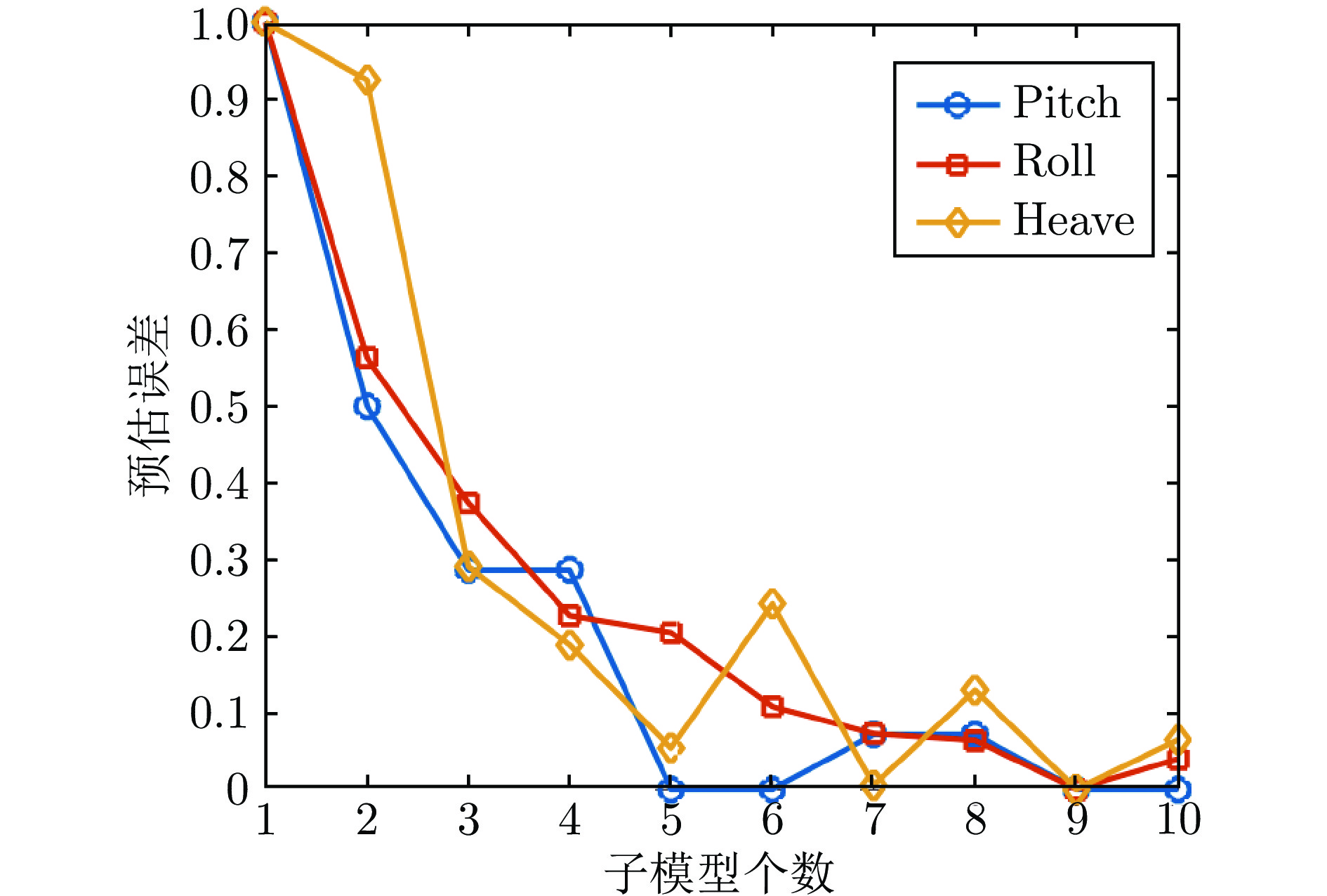
4.2.3 网络架构性能分析
对于单个子模型, 实验中对比了本文所提方法生成的网络架构与手动设计的网络架构的性能. 具体而言, 首先通过网格搜索的策略得到网络架构超参数(隐层神经元个数与网络深度)与预估性能之间的关系, 再将本文所提方法自动确定的超参数映射到超参数空间中. 实验结果如图10所示, 从左到右分别为纵摇、横摇、垂荡的实验结果, 其中折线代表本文方法生成的网络架构. 可以看出, 本文方法得到的架构超参数接近最(较)优值. 对于由多个子模型组成的学习模型, 实验中分析了模型性能与子模型个数的关系. 实验结果如图11所示, 为便于显示, 纵轴为归一化后的预估误差. 可以看出, 采用多个子模型构建出集成学习系统能有效提升最终的预测性能. 以上两组实验结果证明了本文所提模型构建方法的有效性.
4.2.4 与同类工作对比
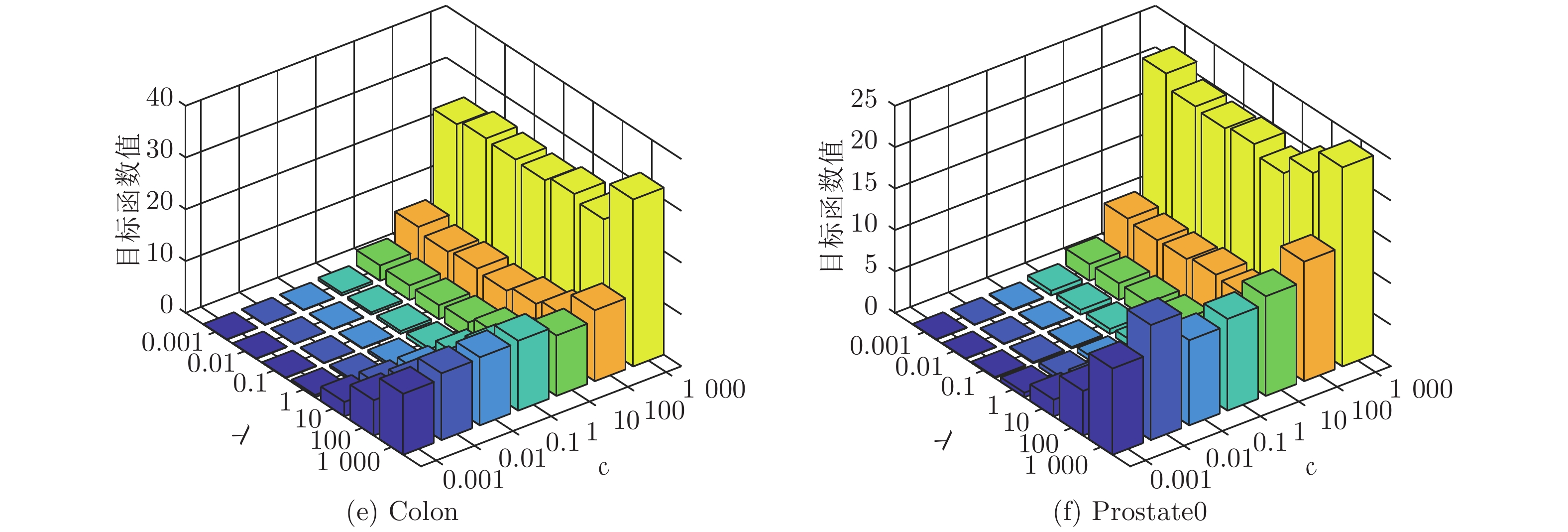
表1为本文方法与公开报道的同类工作[10]在相同实验设置下的对比实验结果. 实验中基线方法包括反向传播神经网络(BPNN)、极限学习机(ELM)、带有粒子群优化的核极限学习机(Kernel extreme learning machine with particle swarm optimization, KELM-PSO)、基于卡尔曼滤波的方法(Kalman filter)和基于自回归的方法(Autoregression). 从实验结果可以看出, 本文方法的甲板纵摇和垂荡运动的预估效果明显优于其他基线算法, 横摇运动预估的均方误差大于BPNN, KELM-PSO和Autoregression, 小于ELM和Kalman filter, 但本文模型在超参数调优和网络架构设计方面具有优势.
表 1 本文所提方法与其他方法的预测均方误差对比Table 1 Comparison of prediction MSE between our proposed method with others方法 Pitch Roll Heave BPNN 0.021 2 0.016 5 0.075 4 ELM 0.019 8 0.116 5 0.076 5 KELM-PSO 0.012 4 0.013 7 0.056 0 Kalman filter 0.022 4 0.573 7 0.026 1 Autoregression 0.006 6 0.016 8 0.020 8 本文方法 0.001 5 0.025 4 0.002 9 注: 加粗字体表示各列最优结果. 5. 算法分析
本文模型采用数据驱动的自适应动态构建策略, 与基于物理模型的方法相比, 不需要建立舰船运动的精确动力学模型, 而是根据当前及历史运动状态数据对未来的运动状态进行短周期预估, 具有更好的通用性. 与其他基于神经网络的方法相比, 本文模型采用的非梯度伪逆学习算法是误差正向传播, 并在传播的过程中根据数据的固有性质动态确定复杂网络的架构, 简化了模型超参数调试, 提高了训练速度, 且不要求激活函数可微. 另外, 与其他采用随机映射的非梯度学习算法相比, 本文方法采用输入数据的伪逆矩阵的截断奇异值分解作为正交投影算子, 能够抑制噪声中的不稳定特征, 再通过引入基于图拉普拉斯的正则化技术, 进一步提高模型鲁棒性.
6. 结束语
本文提出一种面向航空母舰甲板运动预估的鲁棒学习模型, 通过基本的伪逆自编码器逐层动态构建子网络, 再以分治策略通过子网络构建规模更大的学习模型. 模型的训练采用非梯度训练算法, 不仅能提高训练效率, 而且能简化网络架构设计. 通过采用基于图拉普拉斯的正则化方法, 有效提高了模型对噪声的鲁棒性. 通过某型航母在中等海况条件下以典型航速巡航时, 纵摇、横摇及垂荡运动预估的仿真对比实验, 验证了本文所提模型在航空母舰甲板运动预估问题中的有效性及鲁棒性.
-
表 1 数据集描述
Table 1 The data set description
数据集 维数 样本数 类别数 IRIS 4 150 3 Data set IIb 1440 504 2 Data set II 2400 1020 2 DLBCL 5469 77 2 Colon 2 000 62 2 Prostate0 6033 102 2 表 2 传统降维方法的聚类准确率(%) (方差, 维数)
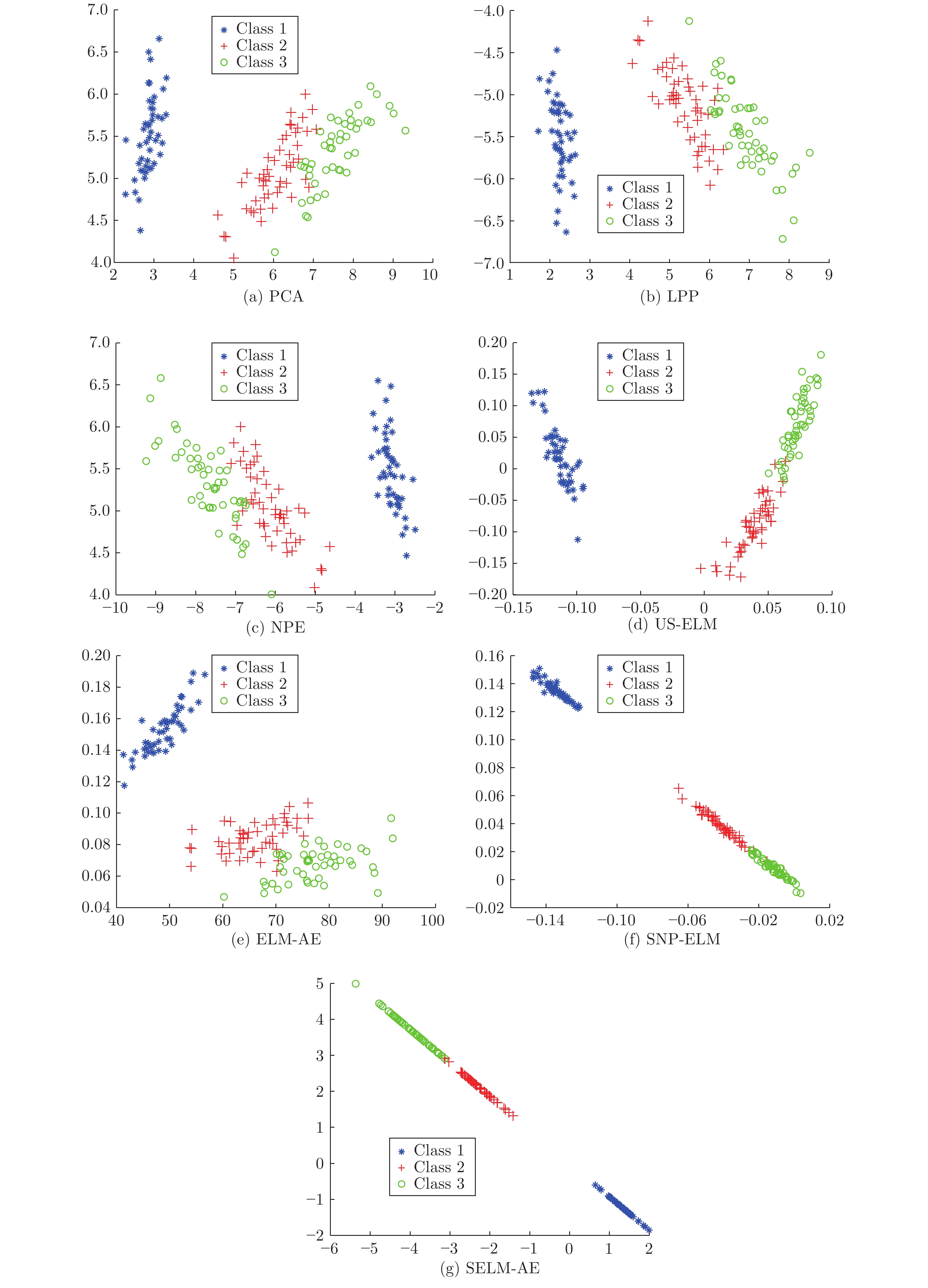
Table 2 Comparison of clustering accuracy of traditional methods (%) (variance, dimension)
数据集 k-means 传统算法 PCA LPP NPE IRIS 89.13 (0.32) 89.07 (0.34, 4) 90.27 (0.84, 2) 88.67 (0.00, 2) Data set IIb 86.47 (2.53) 88.21 (0.61, 4) 88.69 (7.33, 4) 89.58 (6.32, 256) Data set II 72.38 (8.94) 79.31 (4.39, 2) 82.26 (0.13, 512) 82.62 (0.71, 256) DLBCL 68.83 (0.00) 68.83 (0.00, 2) 63.55 (1.86, 8) 69.09 (0.82, 32) Colon 54.84 (0.00) 54.84 (0.00, 2) 54.84 (0.00, 2) 56.45 (0.00, 2) Prostate0 56.86 (0.00) 56.83 (0.00, 2) 56.86 (0.00, 2) 56.86 (0.00, 4) 表 3 ELM降维方法聚类准确率(%) (方差, 维数)(参数)
Table 3 Comparison of clustering accuracy of ELM methods (%) (variance, dimension)(parameters)
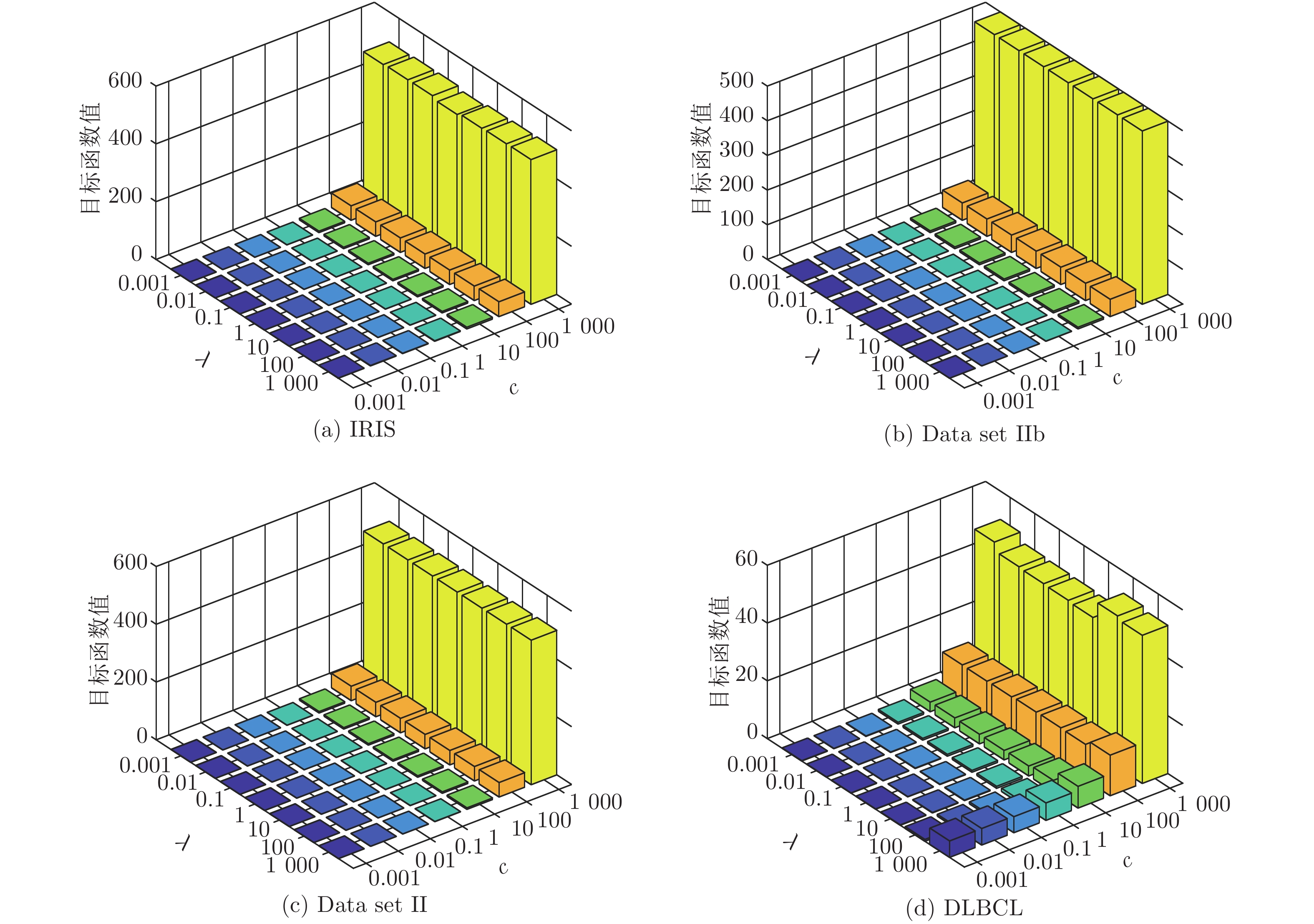
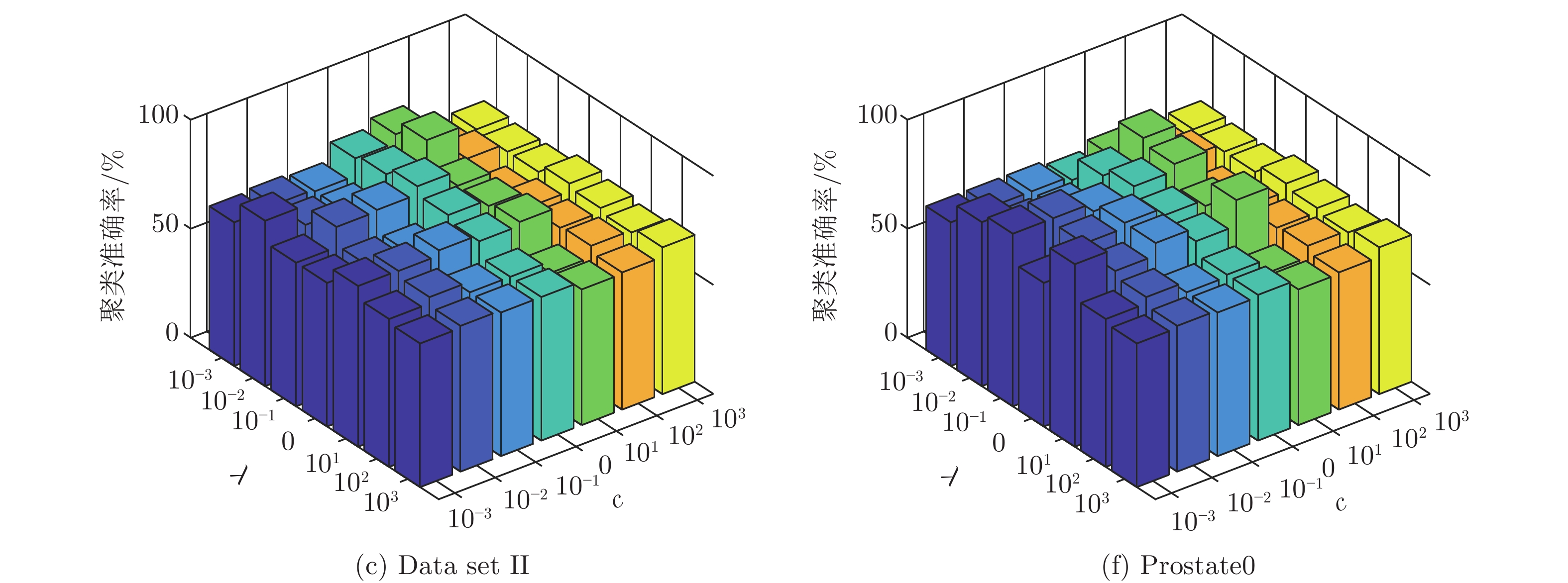
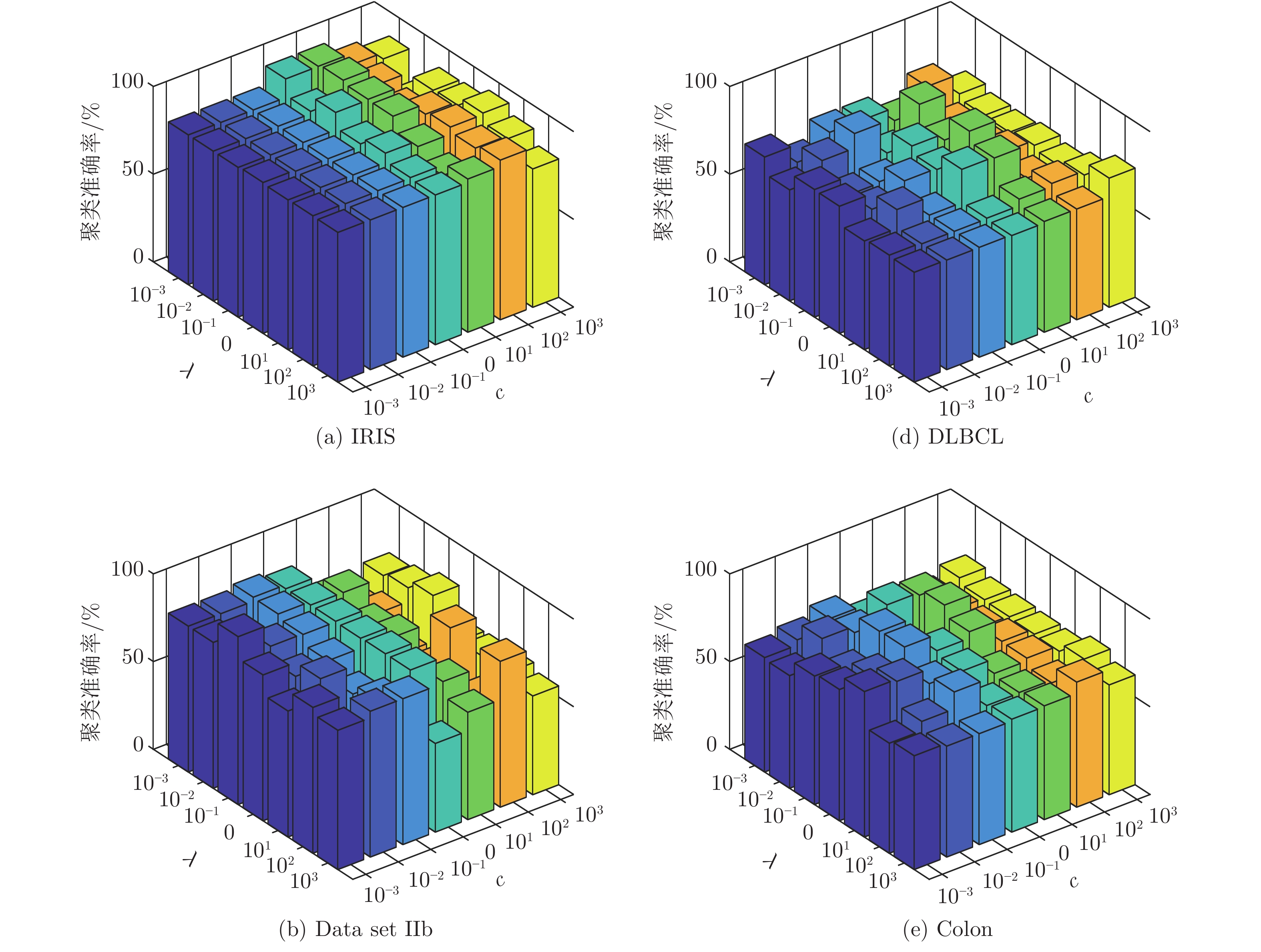
数据集 k-means Unsupervised ELM Subspace + unsupervised ELM US-ELM(λ) ELM-AE(c) ML-ELM-AE (c) SNP-ELM(λ,η,δ) SELM-AE(c, λ) ML-SELM-AE(c, λ) IRIS 89.13
(0.32)93.87
(13.78, 2)
(0.1)93.93
(1.19, 2)
(10)95.20
(1.05, 2)
(0.01)98.46
(0.32, 2)
(10, 0.6, −1)98.00
(0.00, 2)
(10, 0.01)98.40
(0.56, 2)
(10, 0.01)Data set IIb 86.47
(2.53)91.59
(4.25, 4)
(0.1)91.98
(0.25, 4)
(0.1)92.46
(0.08, 16)
(1)92.06
(0.13, 16)
(0.001, 0.8, 0.2)95.29
(0.06, 8)
(0.001, 1)96.63
(0.00, 8)
(0.001, 0.1)Data set II 72.38
(8.94)83.18
(0.32, 256)
(10)82.84
(0.00, 2)
(0.001)83.03
(0.00, 2)
(0.1)83.92
(1.65, 2)
(10, 0.2, −0.2)83.14
(0.00, 2)
(0.01, 1)84.22
(0.00, 2)
(0.001, 10)DLBCL 68.83
(0.00)76.62
(0.00, 32)
(0.001)78.05
(0.73, 2)
(0.001)82.46
(0.68, 2)
(0.001)86.34
(1.78, 8)
(0.001, -0.2, 0.6)83.63
(2.51, 2)
(10, 0.1)86.71
(3.48, 2)
(10, 1)Colon 54.84
(0.00)67.06
(4.19, 32)
(0.001)69.35
(0.00, 2)
(0.001)80.32
(1.02, 2)
(0.001)85.95
(3.69, 8)
(0.001, −0.8, 1)83.87
(0.00, 4)
(10, 0.1)85.97
(0.78, 2)
(10, 0.1)Prostate0 56.86
(0.00)64.09
(5.83, 2)
(0.01)75.98
(0.51, 2)
(0.01)79.61
(1.01, 2)
(0.01)82.92
(2.19, 128)
(0.1, 0.2, 0.8)84.31
(0.00, 2)
(10, 1)85.29
(0.00, 2)
(10, 0.01)表 4 运行时间对比(s)
Table 4 Comparison of running time (s)
数据集 SNP-ELM SELM-AE ML-SELM-AE IRIS 4.58 0.02 0.02 Data set IIb 4.64×103 0.16 0.33 Data set II 8.24×103 0.65 0.76 DLBCL 7.77 0.04 0.06 Colon 3.44×102 0.03 0.11 Prostate0 1.15×102 0.07 0.13 表 5 ML-SELM-AE降维前后数据的聚类准确率(%) (方差)
Table 5 Clustering accuracy before and after ML-SELM-AE dimensionality reduction (%) (variance)
数据集 k-means LSR LRR LatLRR 未降维 已降维 未降维 已降维 未降维 已降维 未降维 已降维 IRIS 89.13 (0.32) 98.40 (0.00) 82.40 (0.69) 97.33 (0.00) 90.87 (0.00) 94.00 (0.83) 81.27 (1.03) 97.33 (0.00) Data set IIb 86.47 (2.53) 93.25 (0.00) 83.13 (0.00) 86.59 (0.19) 83.13 (0.00) 86.11 (0.00) 83.13 (0.00) 86.48 (0.25) Data set II 72.38 (8.94) 84.22 (0.00) 83.24 (0.08) 83.29 (0.05) 83.24 (0.00) 83.24 (0.00) 83.24 (0.00) 83.33 (0.00) DLBCL 68.83 (0.00) 86.71 (3.48) 76.62 (0.00) 81.43 (0.63) 76.62 (0.00) 78.57 (0.68) 74.03 (0.00) 78.18 (3.23) Colon 54.84 (0.00) 85.97 (0.78) 67.74 (0.00) 74.19 (0.00) 63.39 (0.00) 69.35 (0.00) 66.13 (1.67) 75.65 (4.06) Prostate0 56.86 (0.00) 85.29 (0.00) 63.82 (1.37) 70.59 (0.00) 57.84 (0.00) 63.73 (0.00) 55.88 (0.00) 74.51 (0.00) 表 6 三层极限学习机自编码器隐层节点数与聚类准确率(%) (方差)
Table 6 The number of hidden layer nodes and clustering accuracy for three-layer extreme learning machine autoencoder (%) (variance)
数据集 ML-ELM-AE (Multilayer ELM-AE) ML-SELM-AE (Multilayer SELM-AE) 500-100-2 500-100-10 500-100-50 500-100-100 500-100-2 500-100-10 500-100-50 500-100-100 Data set IIb 88.69 (0.00) 91.98 (0.25) 90.89 (0.06) 87.66 (0.20) 95.44 (0.00) 94.92 (0.17) 95.44 (0.00) 94.80 (0.33) Data set II 82.94 (0.00) 82.94 (0.00) 82.94 (0.00) 82.94 (0.00) 83.14 (0.00) 83.04 (0.00) 83.04 (0.00) 83.04 (0.00) DLBCL 74.03 (0.00) 72.99 (3.29) 72.73 (0.00) 69.22 (0.88) 80.52 (0.00) 80.52 (0.00) 78.57 (2.05) 76.62 (0.00) Colon 73.87 (1.67) 59.68 (0.00) 69.52 (7.35) 59.03 (0.83) 78.55 (2.53) 75.97 (7.81) 76.13 (9.46) 70.48 (4.37) Prostate0 66.67 (0.00) 60.78 (0.00) 59.80 (0.00) 62.75 (0.00) 77.16 (0.47) 82.35 (0.00) 78.33 (6.51) 80.39 (0.00) 数据集 2-2-2 10-10-10 50-50-50 100-100-100 2-2-2 10-10-10 50-50-50 100-100-100 Data set IIb 92.46 (0.08) 90.48 (0.00) 90.16 (0.17) 90.40 (0.25) 96.63 (0.00) 95.83 (0.00) 95.44 (0.00) 94.84 (0.00) Data set II 83.04 (0.00) 83.04 (0.00) 83.04 (0.00) 82.94 (0.00) 84.22 (0.00) 83.14 (0.00) 83.04 (0.00) 83.04 (0.00) DLBCL 83.12 (0.00) 77.01 (0.63) 68.83 (0.00) 68.70 (0.41) 86.75 (4.23) 80.52 (0.00) 78.96 (0.82) 76.62 (0.00) Colon 80.64 (0.00) 60.00 (2.50) 70.00 (1.36) 62.90 (0.00) 85.97 (0.78) 68.23 (0.78) 80.65 (0.00) 76.61 (9.48) Prostate0 80.39 (0.00) 57.45 (0.83) 64.41 (0.47) 63.73 (0.00) 85.29 (0.00) 69.61 (0.00) 79.12 (6.67) 84.31 (0.00) -
[1] Hinton G E, Salakhutdinov R R. Reducing the dimensionality of data with neural networks. Science, 2006, 313(5786): 504-507. doi: 10.1126/science.1127647 [2] 田娟秀, 刘国才, 谷珊珊, 鞠忠建, 刘劲光, 顾冬冬. 医学图像分析深度学习方法研究与挑战. 自动化学报, 2018, 44(3): 401-424.Tian Juan-Xiu, Liu Guo-Cai, Gu Shan-Shan, Ju Zhong-Jian, Liu Jin-Guang, Gu Dong-Dong. Deep learning in medical image analysis and its challenges. Acta Automatica Sinica, 2018, 44(3): 401-424. [3] Rik D, Ekta W. Partition selection with sparse autoencoders for content based image classification. Neural Computing and Applications, 2019, 31(3): 675-690. doi: 10.1007/s00521-017-3099-0 [4] Shao H D, Jiang H K, Zhao H W. A novel deep autoencoder feature learning method for rotating machinery fault diagnosis. Mechanical Systems and Signal Processing, 2017, 95: 187-204. doi: 10.1016/j.ymssp.2017.03.034 [5] Chiang H T, Hsieh Y Y, Fu S W, Hung K H, Tsao Y, Chien S Y. Noise reduction in ECG signals using fully convolutional denoising autoencoders. IEEE Access, 2019, 7: 60806-60813. doi: 10.1109/ACCESS.2019.2912036 [6] Yildirim O, Tan R S, Acharya U R. An efficient compression of ECG signals using deep convolutional autoencoders. Cognitive Systems Research, 2018, 52: 198-211. doi: 10.1016/j.cogsys.2018.07.004 [7] Liu W F, Ma T Z, Xie Q S, Tao D P, Cheng J. LMAE: a large margin auto-encoders for classification. Signal Processing, 2017, 141: 137-143. doi: 10.1016/j.sigpro.2017.05.030 [8] Ji P, Zhang T, Li H, Salzmann M, Reid L. Deep subspace clustering networks. In: Proceedings of the 31st Conference on Neural Information Processing Systems. Long Beach, USA, 2017. 23−32 [9] Kasun L L C, Yang Y, Huang G B, Zhang ZH Y. Dimension reduction with extreme learning machine. IEEE Transactions on Image Processing, 2016, 25(8): 3906-3918. doi: 10.1109/TIP.2016.2570569 [10] Huang G B, Zhu Q Y, Siew C K. Extreme learning machine: A new learning scheme of feedforward neural networks. In: Proceedings of Internatinaol Joint Conference on Neural Networks. Budapest, Hungary: IEEE, 2004. 985−990 [11] 许夙晖, 慕晓冬, 柴栋, 罗畅. 基于极限学习机参数迁移的域适应算法. 自动化学报, 2018, 44(2): 311-317.Xu Su-Hui, Mu Xiao-Dong, Chai Dong, Luo Chang. Domain adaption algorithm with ELM parameter transfer. Acta Automatica Sinica, 2018, 44(2): 311-317. [12] Huang G, Song S J, Gupta J N D. Semi-supervised and Unsupervised Extreme Learning Machines. IEEE Transactions on Cybernetics, 2014, 44(12): 2405-2417. doi: 10.1109/TCYB.2014.2307349 [13] 陈晓云, 廖梦真. 基于稀疏和近邻保持的极限学习机降维. 自动化学报, 2019, 45(2): 325-333.Chen Xiao-Yun, Liao Meng-Zhen. Dimensionality reduction with extreme learning machine based on sparsity and neighborhood preserving. Acta Automatica Sinica, 2019, 45(2): 325-333. [14] Lu C Y, Min H, Zhao Z Q. Robust and efficient subspace segmentation via least squares regression. In: Proceedings of the 12th European Conference on Computer Vision. Berlin, Germany: Springer, 2012. 347−360 [15] Ji P, Salzmann M, Li H D. Efficient dense subspace clustering. In: Proceedings of Winter Conference on Applications of Computer Vision. Steamboat Springs, CO, USA: IEEE, 2014.461−468 [16] Sun K, Zhang J S, Z C X, Hu J Y. Generalized extreme learning machine autoencoder and a new deep neural network. Neurocomputing, 2017, 230: 374-381. doi: 10.1016/j.neucom.2016.12.027 [17] Ma J, Yuan Y Y. Dimension reduction of image deep feature using PCA. Journal of Visual Communication and Image Representatio, 2019, 63: 1-8. [18] Wang S J, Xie D Y, Chen F, Gao Q X. Dimensionality reduction by LPP-L21. IET Computer Vision, 2018, 12(5): 659-665. doi: 10.1049/iet-cvi.2017.0302 [19] Kong D D, Chen Y J, Li N, Duan C Q, Lu L X, Chen D X. Tool wear estimation in end milling of titanium alloy using NPE and a novel WOA-SVM model. IEEE Transactions on Instrumentation and Measurement, 2020, 69(7): 5219-5232. doi: 10.1109/TIM.2019.2952476 [20] UCI Machine Learning Repository. [Online], available: http://archive.ics.uci.edu, September 8, 2020 [21] NYS Department of Health. [Online], available: http://www.bbci.de/competition/, September 8, 2020 [22] Gene Expression Model Selector. [Online], available: http://www.gems-system.org, September 8, 2020 [23] Kaper M, Ritter H. Generalizing to new subject in brain-computer interfacing. In: Proceedings of the 26th Annual International Conference of the IEEE Engineering in Medicine and Biology Society. San Francisco, USA: IEEE, 2004. 4363−4366 [24] Chen X Y, Jian C R. Gene expression data clustering based on graph regularized subspace segmentation. Neurocomputing, 2014, 143: 44-50. doi: 10.1016/j.neucom.2014.06.023 [25] Liu G C, Lin Z C, Yu Y. Robust subspace segmentation by low-rank representation. In: the 27th International Conference on Machine Learning. Haifa, Israel, 2010. 663−670 [26] Liu G, Yan S. Latent low-rank representation for subspace segmentation and feature extraction. In: Proceedings of International Conference on Computer Vision. Barcelona, Spain: IEEE, 2011. 1615−1622 -
 AAS-CN-2020-0684数据.zip
AAS-CN-2020-0684数据.zip

-




 下载:
下载:
 下载:
下载: