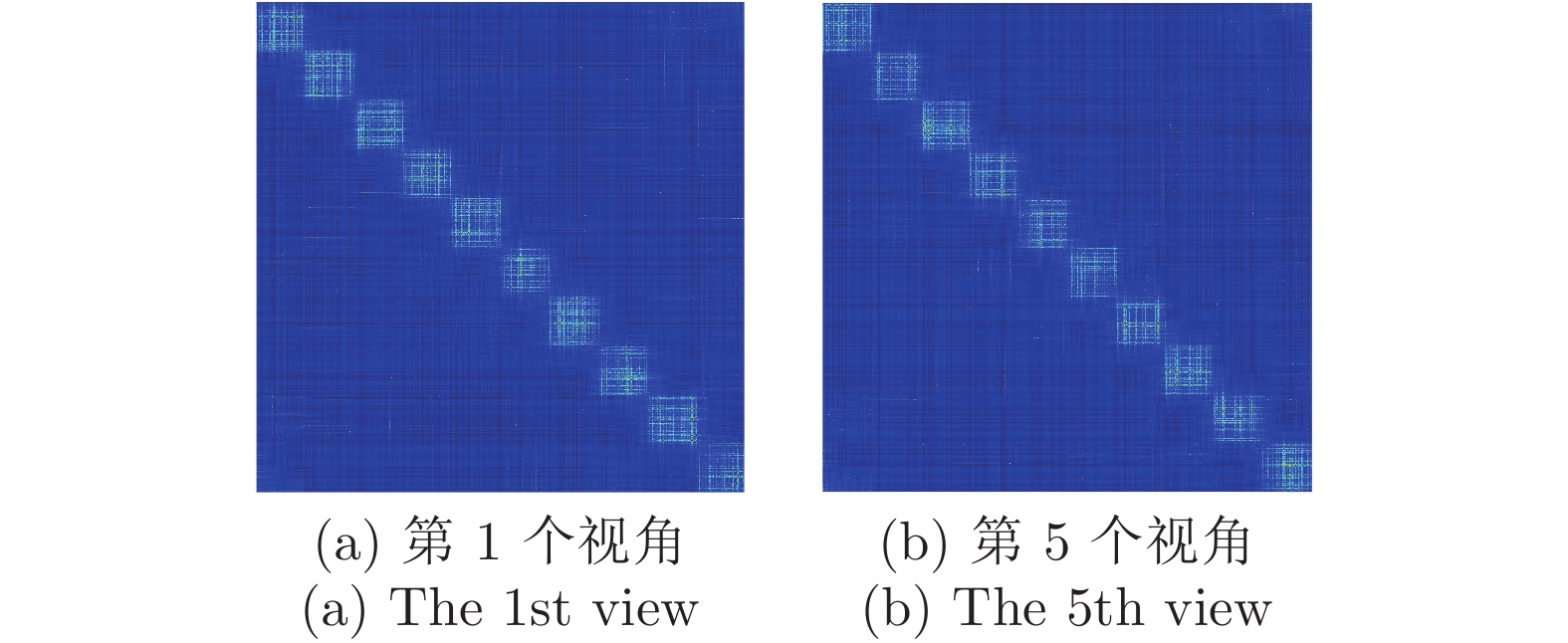
-
摘要: 传统多视角聚类都基于视角完备假设, 要求所有样本的视角信息完整, 不能处理存在部分视角缺失情形下的不完整多视角聚类任务. 为解决该问题, 提出一种基于低秩张量图学习的不完整多视角聚类方法. 为了恢复相似图中缺失视角所对应的样本关联信息, 该方法将低秩张量图约束和视角内在图保持约束融入到多视角谱聚类模型. 通过在一个统一模型中同时挖掘视角间的互补信息和视角内未缺失样例的关联信息, 所提出的方法能够得到表征样例邻接关系的完整相似图和视角间一致的最优聚类指示矩阵. 与12种不完整多视角聚类方法进行实验对比, 实验结果表明所提出的方法在多种视角缺失率下的5个数据集上获得了最好的聚类性能.Abstract: Conventional multi-view clustering methods all assume that the give multi-view data is complete, i.e., all views are fully observed, which are not applicable to the incomplete multi-view clustering case with missing-views. To address this issue, we propose a method, called low-rank tensor graph learning (LASAR). To recover the missing connections corresponding to the missing views in the graph space, the proposed method integrates the low-rank tensor graph constraint and intra-view graph constraint into the multi-view spectral clustering framework. By exploring the inter-view information and intra-view information with respect to the observed views simultaneously, the proposed method can obtain the optimal completed graphs of all views and the optimal clustering indicator matrix shared by all views. Experimental results on five datasets with different missing-view rates show that the proposed method obtains better performance than 12 state-of-the-art incomplete multi-view clustering methods.
-
Key words:
- Multi-view clustering /
- view missing /
- incomplete multi-view clustering /
- graph learning
-
表 1 各方法在不同视角缺失率下的Caltech7和BBCSport数据集上的实验结果
Table 1 Experimental results of different methods on Caltech7 and BBCSport datasets with different missing-view rates
数据集 方法 NMI (%) ACC (%) Purity (%) 0.1 0.3 0.5 0.1 0.3 0.5 0.1 0.3 0.5 Caltech7 MultiNMF+均值 30.16 28.52 28.88 46.39 40.61 37.92 79.80 78.43 78.03 MultiNMF+KNN 36.38 34.27 34.19 50.71 43.98 42.66 80.68 80.88 80.17 CCo_MVSC+零值 35.53 34.18 31.52 38.47 38.16 37.35 78.33 77.89 76.21 CCo_MVSC+KNN 37.38 37.18 37.33 39.06 39.62 37.34 80.25 80.36 81.45 MIC 33.71 27.35 20.44 44.07 38.01 35.80 78.12 73.31 68.26 OMVC 28.13 25.32 18.76 40.88 36.82 33.28 79.21 77.73 74.05 DAIMC 44.61 38.45 36.28 48.29 47.46 44.89 83.32 76.83 75.50 OPIMC 42.98 41.54 35.98 49.24 48.34 44.12 84.89 83.70 80.64 UEAF 39.44 31.07 24.02 50.82 42.71 36.32 81.49 78.26 76.29 IMKKM-IK-MKC 24.09 23.45 22.91 36.54 34.87 36.05 72.98 73.82 72.52 IMVSC_AGL 44.05 42.61 37.35 54.72 54.61 51.78 84.27 83.98 82.31 GM-PMVC 60.26 52.35 47.95 62.65 54.95 47.65 91.26 84.49 83.93 LASAR 66.08 62.80 52.76 70.29 69.02 66.46 92.76 90.27 84.84 BBCSport MultiNMF+均值 23.48 18.25 14.79 48.58 42.75 40.34 47.17 44.66 43.10 MultiNMF+KNN 33.41 29.17 32.66 56.03 54.14 54.66 58.62 55.51 58.27 CCo_MVSC+零值 62.79 57.69 39.55 72.76 70.06 61.38 81.49 78.62 66.72 CCo_MVSC+KNN 59.24 56.25 38.97 69.02 69.83 60.06 80.92 78.56 68.62 MIC 29.90 25.84 24.01 51.21 46.21 46.03 55.00 51.72 52.41 OMVC 30.64 41.57 40.63 53.33 51.38 48.79 56.49 59.20 57.47 DAIMC 56.62 50.17 37.89 68.62 63.45 56.89 76.90 71.72 61.03 OPIMC 35.66 31.56 21.75 54.14 52.93 45.69 58.28 56.72 50.86 UEAF 70.71 68.25 55.13 78.22 77.24 69.31 87.41 87.07 77.07 IMKKM-IK-MKC 72.91 64.42 53.52 77.55 75.66 67.07 88.76 84.03 77.00 IMVSC_AGL 70.46 66.11 54.57 76.41 74.48 69.31 87.41 85.00 78.10 GM-PMVC 70.34 66.26 48.93 74.66 73.48 60.07 85.17 84.48 72.68 LASAR 77.09 70.30 57.04 85.00 77.41 73.28 90.00 88.62 78.79 表 2 各方法在不同视角缺失率下的Handwritten及不同视角配对率下的Animal和Reuters数据集上的实验结果
Table 2 Experimental results of different methods on Handwritten dataset with different missing-view rates, Animal and Reuters datasets with different paired-view rates
数据集 方法 NMI (%) ACC (%) 0.1 0.3 0.5 0.7 0.1 0.3 0.5 0.7 Handwritten MultiNMF+均值 72.05 60.11 41.99 20.88 82.35 71.74 52.03 31.85 MultiNMF+KNN 74.23 74.77 73.81 67.93 83.64 83.89 83.53 78.33 CCo_MVSC+零值 70.90 68.23 62.86 53.14 74.61 73.17 70.15 64.62 CCo_MVSC+KNN 73.03 73.77 73.06 67.66 76.76 76.79 76.20 75.52 MIC 70.84 65.39 52.95 34.71 77.59 73.29 61.27 41.34 OMVC 56.72 44.99 35.16 25.83 65.04 55.00 36.40 29.80 DAIMC 79.78 76.65 68.77 47.10 88.86 86.73 81.92 60.44 OPIMC 77.26 73.74 66.57 51.86 80.20 76.45 69.50 56.66 UEAF 77.74 69.37 55.09 50.56 85.80 76.11 65.39 61.11 IMKKM-IK-MKC 69.43 65.42 59.04 47.36 71.78 69.07 66.08 55.55 IMVSC_AGL 93.68 90.55 86.39 75.44 97.15 95.50 93.19 84.08 GM-PMVC 99.84 99.64 99.12 97.14 99.94 99.87 99.66 98.87 LASAR 99.92 99.81 99.32 99.13 99.97 99.93 99.73 99.68 Animal MultiNMF+均值 48.25 53.51 56.30 61.48 42.04 46.33 48.62 52.92 MultiNMF+KNN 53.68 55.58 58.85 60.68 45.15 47.89 51.51 53.16 CCo_MVSC+零值 52.19 55.31 58.31 61.71 48.12 52.03 54.72 56.73 CCo_MVSC+KNN 49.13 57.96 61.37 63.86 47.71 53.58 56.79 58.41 MIC 48.96 52.79 55.69 59.30 39.44 43.38 45.88 49.15 OMVC 44.66 50.77 53.11 55.38 30.66 42.51 43.98 46.39 DAIMC 49.33 55.03 59.36 62.76 42.76 50.18 53.87 56.42 OPIMC 44.29 52.34 58.51 62.04 37.86 46.33 53.14 53.88 UEAF 54.88 62.10 64.27 68.62 46.76 52.45 58.15 62.71 IMKKM-IK-MKC 51.47 57.21 62.14 66.50 46.05 53.61 58.48 61.85 IMVSC_AGL 56.38 59.65 63.26 66.71 51.61 54.94 57.97 60.73 GM-PMVC 55.57 57.62 61.25 64.55 47.84 50.89 53.99 57.81 LASAR 65.79 69.61 90.26 92.97 58.05 65.67 86.28 88.05 Reuters MultiNMF+均值 19.58 18.82 18.56 19.51 37.68 38.31 37.27 37.58 MultiNMF+KNN 19.91 23.17 22.50 22.21 37.95 38.08 37.47 37.35 CCo_MVSC+零值 17.04 18.66 18.10 20.11 34.66 37.39 37.46 40.01 CCo_MVSC+KNN 17.64 19.33 19.67 20.95 35.42 37.79 39.41 41.06 MIC 18.72 21.30 22.78 24.34 38.47 40.85 41.37 43.05 OMVC 18.05 19.77 21.37 22.97 36.78 37.44 39.45 41.09 DAIMC 26.53 29.98 30.27 31.55 43.32 46.67 47.78 48.89 OPIMC 19.02 22.47 23.82 25.39 40.58 42.19 43.87 44.43 UEAF 24.77 26.85 27.14 28.36 41.45 44.95 46.87 47.92 IMKKM-IK-MKC 24.30 26.75 26.88 28.67 41.13 45.71 46.40 47.29 IMVSC_AGL 25.75 28.41 28.39 29.79 42.38 46.29 47.82 48.91 GM-PMVC 26.22 29.35 30.71 30.89 43.15 46.88 48.19 50.44 LASAR 28.34 32.01 31.27 32.77 45.38 48.14 50.80 52.77 表 3 各方法在5个数据集上的运行时间(s), 其中视角缺失率或配对率为30%
Table 3 Running time (s) of different methods on the above five datasets with a missing-view rate or paired-view rate of 30%
方法 Caltech7 BBCSport Handwritten Animal Reuters 计算复杂度 MultiNMF+均值 60.38 28.62 35.54 13489.18 27385.81 ${\rm{O} } \left( {\tau T\sum\nolimits_{v = 1}^l { {m_v}cn} } \right)$[10] MultiNMF+KNN 59.05 25.46 40.69 13499.88 27408.45 ${\rm{O} } \left( {\tau T\sum\nolimits_{v = 1}^l { {m_v}cn} } \right)$[10] CCo_MVSC+零值 3.01 0.35 5.18 174.16 489.83 ${\rm{O} } \left( {\tau \left( {l{n^3} + {n^3} } \right)} \right)$ CCo_MVSC+KNN 4.36 0.46 7.69 243.77 699.74 ${\rm{O}} \left( {\tau \left( {l{n^3} + {n^3}} \right)} \right)$ MIC 225.31 3.01 149.87 3693.92 22499.46 ${\rm{O}} \left( {\tau T\sum\nolimits_{v = 1}^l {{m_v}cn} } \right)$[10] OMVC 152.54 3.17 27.77 3409.08 18154.71 ${\rm{O}} \left( {\tau T\sum\nolimits_{v = 1}^l {{m_v}cn} } \right)$[10] DAIMC 62.74 81.18 26.72 2191.43 96628.66 ${\rm{O}} \left( {\tau \left( {Tnc{m_{\max }} + lm_{\max }^3} \right)} \right)$[17] OPIMC 0.16 0.05 0.09 3.31 1.05 ${\rm{O}} \left( {\tau lcn{m_{\max }}} \right)$[18] UEAF 4.64 2.36 7.05 1258.93 18599.64 ${\rm{O}} \left( {\tau \left( {2{n^3} + \sum\nolimits_{v = 1}^l {{m_v}{c^2}} } \right)} \right)$[19] IMKKM-IK-MKC 23.68 0.22 41.22 975.38 1762.95 ${\rm{O}} \left( {\tau \left( {{n^3} + l{n^3} + {l^3}} \right)} \right)$[21] IMVSC_AGL 173.58 2.57 81.13 5265.99 28672.45 ${\rm{O}} \left( {\tau \left( {l{n^3} + {n^3} + \sum\nolimits_{v = 1}^l {n_v^3} } \right)} \right)$[20] GM-PMVC 607.40 14.63 602.81 50725.43 1.78×105 ${\rm{O}} \left( {\tau l\left( {{n^3} + kdn + {k^3} + {k^2}d} \right)} \right)$[22] LASAR 73.87 1.09 115.60 2352.88 5677.46 ${\rm{O}} \left( {\tau \left( {l{n^2}\log \left( n \right){\rm{ + }}{l^2}{n^2} + c{n^2}} \right)} \right)$ 注: 本表未考虑K-means聚类算法的计算复杂度. 表中, T表示子循环迭代步数, mmax表示所有视角的最大特征维度, nv表示第$v$个视角的未缺失样例数, d表示多视角数据特征的平均维度, k表示矩阵分解模型中的隐层表征维度. -
[1] Jain A K. Data clustering: 50 years beyond K-means. Pattern Recognition Letters, 2010, 31(8): 651-666 doi: 10.1016/j.patrec.2009.09.011 [2] Yang Y, Wang H. Multi-view clustering: A survey. Big Data Mining and Analytics, 2018, 1(2): 83-107 doi: 10.26599/BDMA.2018.9020003 [3] 张长青. 基于自表达的多视角子空间聚类方法研究 [博士学位论文], 天津大学, 中国, 2015Zhang Chang-Qing. Self-representation Based Multiview Subspace Clustering [Ph.D. dissertation], Tianjin University, China, 2015 [4] Cai X, Nie F P, Huang H. Multi-view K-means clustering on big data. In: Proceedings of the 23rd International Joint Conference on Artificial Intelligence. Beijing, China: AAAI Press, 2013. 2598−2604 [5] 张嘉旭, 王骏, 张春香, 林得富, 周塔, 王士同. 基于低秩约束的熵加权多视角模糊聚类算法. 自动化学报, 2022, 48(7): 1760-1770 doi: 10.16383/j.aas.c190350Zhang Jia-Xu, Wang Jun, Zhang Chun-Xiang, Lin De-Fu, Zhou Ta, Wang Shi-Tong. Entropy-weighting multi-view fuzzy C-means with low rank constraint. Acta Automatica Sinica, 2022, 48(7): 1760-1770 doi: 10.16383/j.aas.c190350 [6] 张祎, 孔祥维, 王振帆, 付海燕, 李明. 基于多视图矩阵分解的聚类分析. 自动化学报, 2018, 44(12): 2160-2169 doi: 10.16383/j.aas.2018.c160636Zhang Yi, Kong Xiang-Wei, Wang Zhen-Fan, Fu Hai-Yan, Li Ming. Matrix factorization for multi-view clustering. Acta Automatica Sinica, 2018, 44(12): 2160-2169 doi: 10.16383/j.aas.2018.c160636 [7] Zhan K, Nie F P, Wang J, Yang Y. Multiview consensus graph clustering. IEEE Transactions on Image Processing, 2019, 28(3): 1261-1270 doi: 10.1109/TIP.2018.2877335 [8] Wen J, Zhang Z, Zhang Z, Fei L K, Wang M. Generalized incomplete multiview clustering with flexible locality structure diffusion. IEEE Transactions on Cybernetics, 2021, 51(1): 101-114 doi: 10.1109/TCYB.2020.2987164 [9] 杨旭, 朱振峰, 徐美香, 张幸幸. 多视角数据缺失补全. 软件学报, 2018, 29(4): 945-956 doi: 10.13328/j.cnki.jos.005416Yang Xu, Zhu Zhen-Feng, Xu Mei-Xiang, Zhang Xing-Xing. Missing view completion for multi-view data. Journal of Software, 2018, 29(4): 945-956 doi: 10.13328/j.cnki.jos.005416 [10] Shao W X, He L F, Lu C T, Yu P S. Online multi-view clustering with incomplete views. In: Proceedings of the IEEE International Conference on Big Data (Big Data). Washington, United States: IEEE, 2016. 1012−1017 [11] Xu C, Guan Z Y, Zhao W, Wu H C, Niu Y F, Ling B L. Adversarial incomplete multi-view clustering. In: Proceedings of the 28th International Joint Conference on Artificial Intelligence. Macao, China: AAAI Press, 2019. 3933−3939 [12] Wen J, Zhang Z, Xu Y, Zhong Z F. Incomplete multi-view clustering via graph regularized matrix factorization. In: Proceedings of the European Conference on Computer Vision Workshop. Munich, Germany: Springer, 2018. 593−608 [13] Zhao H D, Liu H F, Fu Y. Incomplete multi-modal visual data grouping. In: Proceedings of the 25th International Joint Conference on Artificial Intelligence. New York, United States: AAAI Press, 2016. 2392−2398 [14] Li S Y, Jiang Y, Zhou Z H. Partial multi-view clustering. In: Proceedings of the 28th AAAI Conference on Artificial Intelligence. Québec City, Canada: AAAI Press, 2014. 1968−1974 [15] Trivedi A, Rai P, Daumé H, DuVall S L. Multiview clustering with incomplete views. In: Proceedings of the Annual Conference on Neural Information Processing Systems Workshop on Machine Learning for Social Computing, Hyatt Regency. Vancouver, Canada: MIT Press, 2010. 1−8 [16] Shao W X, He L F, Yu P S. Multiple incomplete views clustering via weighted nonnegative matrix factorization with L2,1 regularization. In: Proceedings of the Joint European Conference on Machine Learning and Knowledge Discovery in Databases. Porto, Portugal: Springer, 2015. 318−334 [17] Hu M L, Chen S C. Doubly aligned incomplete multi-view clustering. In: Proceedings of the 27th International Joint Conference on Artificial Intelligence. Stockholm, Sweden: AAAI Press, 2018. 2262−2268 [18] Hu M L, Chen S C. One-pass incomplete multi-view clustering. In: Proceedings of the 33rd AAAI Conference on Artificial Intelligence. Honolulu, USA: AAAI Press, 2019. 3838−3845 [19] Wen J, Zhang Z, Xu Y, Zhang B, Fei L K, Liu H. Unified embedding alignment with missing views inferring for incomplete multi-view clustering. In: Proceedings of the 33rd AAAI Conference on Artificial Intelligence and the 31st Innovative Applications of Artificial Intelligence Conference and the 9th AAAI Symposium on Educational Advances in Artificial Intelligence. Honolulu, USA: AAAI Press, 2019. Article No. 661 [20] Wen J, Xu Y, Liu H. Incomplete multiview spectral clustering with adaptive graph learning. IEEE Transactions on Cybernetics, 2020, 50(4): 1418-1429 doi: 10.1109/TCYB.2018.2884715 [21] Liu X, Zhu X, Li M, Wang L, Zhu E, Liu T, et al. Multiple kernel k-means with incomplete kernels. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2020, 42(5): 1191-1204 [22] 赵博宇, 张长青, 陈蕾, 刘新旺, 李泽超, 胡清华. 生成式不完整多视图数据聚类. 自动化学报, 2021, 47(8): 1867-1875 doi: 10.16383/j.aas.c200121Zhao Bo-Yu, Zhang Chang-Qing, Chen Lei, Liu Xin-Wang, Li Ze-Chao, Hu Qing-Hua. Generative model for partial multi-view clustering. Acta Automatica Sinica, 2021, 47(8): 1867-1875 doi: 10.16383/j.aas.c200121 [23] Wu J L, Lin Z C, Zha H B. Essential tensor learning for multi-view spectral clustering. IEEE Transactions on Image Processing, 2019, 28(12): 5910-5922 doi: 10.1109/TIP.2019.2916740 [24] Nie F P, Wang X Q, Jordan M I, Huang H. The constrained Laplacian rank algorithm for graph-based clustering. In: Proceedings of the 30th AAAI Conference on Artificial Intelligence. Phoenix, USA: AAAI Press, 2016. 1969−1976 [25] Wright T G, Trefethen L N. Large-scale computation of pseudospectra using ARPACK and eigs. SIAM Journal on Scientific Computing, 2001, 23(2): 591-605 doi: 10.1137/S106482750037322X [26] Wang H, Zong L L, Liu B, Yang Y, Zhou W. Spectral perturbation meets incomplete multi-view data. In: Proceedings of the 28th International Joint Conference on Artificial Intelligence. Macao, China: AAAI Press, 2019. 3677−3683 [27] Li F F, Fergus R, Perona P. Learning generative visual models from few training examples: An incremental Bayesian approach tested on 101 object categories. In: Proceedings of the Conference on Computer Vision and Pattern Recognition Workshop. Washington, United States: IEEE, 2004. Article No. 178 [28] Li Y Q, Nie F P, Huang H, Huang J Z. Large-scale multi-view spectral clustering via bipartite graph. In: Proceedings of the 29th AAAI Conference on Artificial Intelligence. Austin, United States: AAAI Press, 2015. 2750−2756 [29] Greene D, Cunningham P. Practical solutions to the problem of diagonal dominance in kernel document clustering. In: Proceedings of the 23rd International Conference on Machine Learning. Pittsburgh, United States: ACM, 2006. 377−384 [30] Duin, Robert. Multiple Features. UCI Machine Learning Repository. [Online], available: https://doi.org/10.24432/C5HC70, Match 2, 2021 [31] Lampert C H, Nickisch H, Harmeling S. Learning to detect unseen object classes by between-class attribute transfer. In: Proceedings of the IEEE Conference on Computer Vision and Pattern Recognition. Miami, United States: IEEE, 2009. 951−958 [32] Zhang C Q, Han Z B, Cui Y J, Fu H Z, Zhou J T, Hu Q H. CPM-nets: Cross partial multi-view networks. In: Proceedings of the 33rd International Conference on Neural Information Processing Systems. Vancouver, Canada: Curran Associates Inc, 2019. Article No. 51 [33] Krizhevsky A, Sutskever I, Hinton G E. ImageNet classification with deep convolutional neural networks. In: Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, United States: MIT Press, 2012. 1097−1105 [34] Simonyan K, Zisserman A. Very deep convolutional networks for large-scale image recognition. In: Proceedings of the 3rd International Conference on Learning Representations. San Diego, USA: 2014. 1−14 [35] Amini M R, Usunier N, Goutte C. Learning from multiple partially observed views-an application to multilingual text categorization. In: Proceedings of the 22nd International Conference on Neural Information Processing Systems. Vancouver, Canada: Curran Associates Inc, 2009. 28−36 [36] Liu J L, Wang C, Gao J, Han J W. Multi-view clustering via joint nonnegative matrix factorization. In: Proceedings of the 13th SIAM International Conference on Data Mining. Austin, United States: SIAM, 2013. 252−260 [37] Kumar A, Rai P, Daumé H. Co-regularized multi-view spectral clustering. In: Proceedings of the 24th International Conference on Neural Information Processing Systems. Granada, Spain: Curran Associates Inc, 2011. 1413−1421 [38] Bhadra S, Kaski S, Rousu J. Multi-view kernel completion. Machine Language, 2017, 106(5): 713-739 [39] Troyanskaya O, Cantor M, Sherlock G, Brown P, Hastie T, Tibshirani R, et al. Missing value estimation methods for DNA microarrays. Bioinformatics, 2001, 17(6): 520-525 doi: 10.1093/bioinformatics/17.6.520 -





 下载:
下载: