-
摘要:
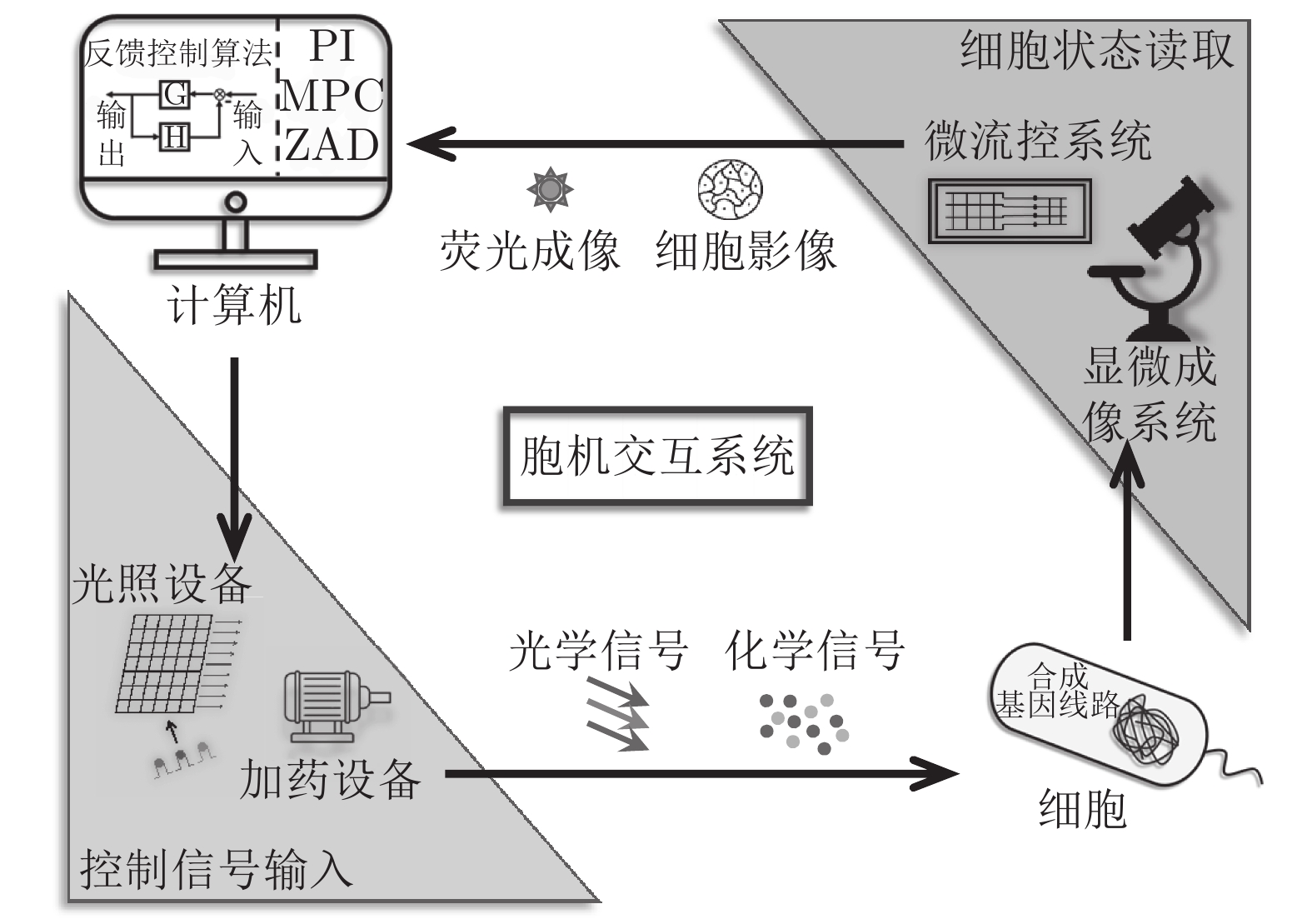
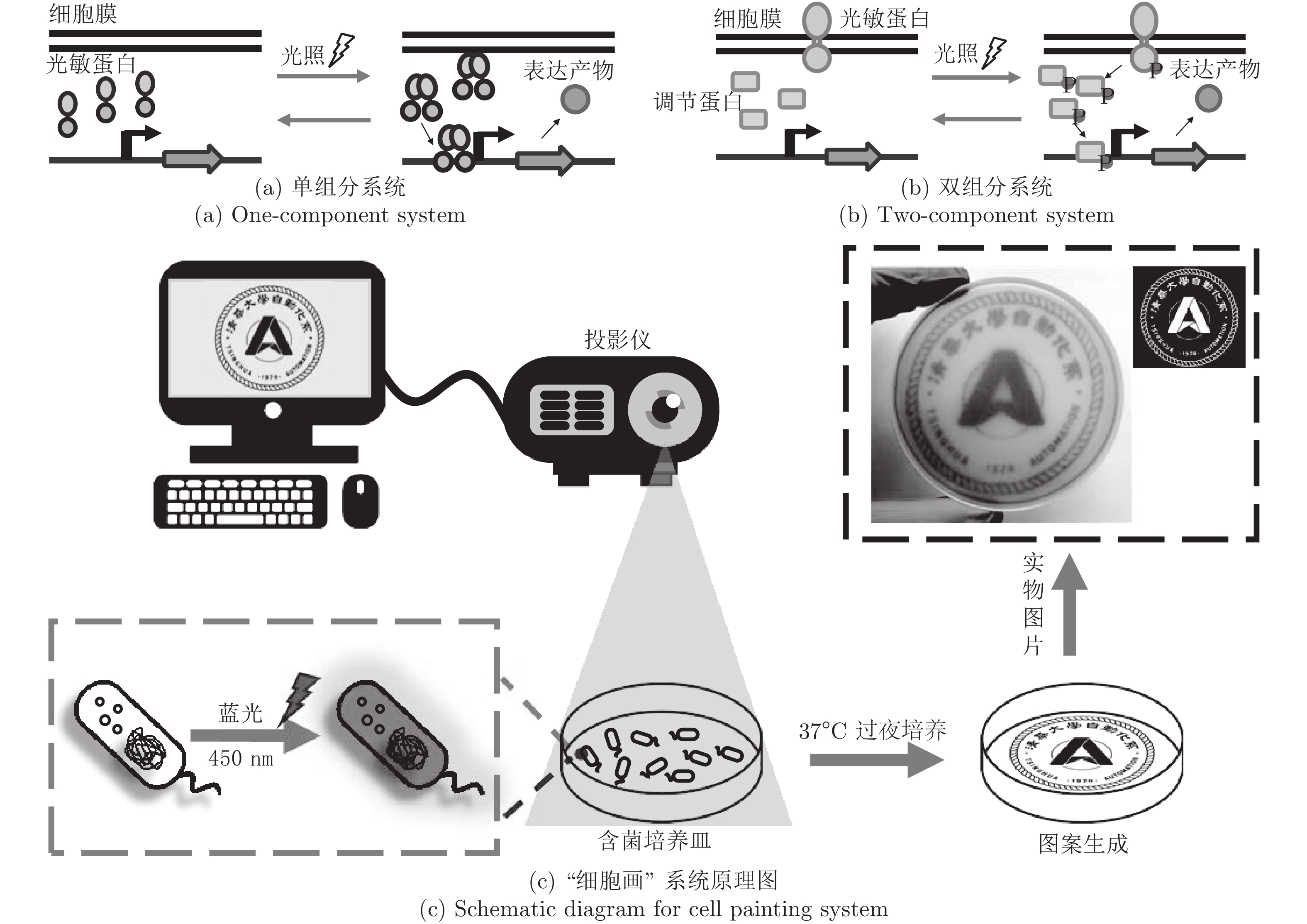
Wiener在控制论(Cybernetics)中强调了两大类控制对象: 机器与动物. 半个世纪以来, 机器控制领域已形成一套较为完备且先进的控制理论, 而在生物控制方面, 由于生物系统的特殊性和复杂性, 对生命的基本组成单位—细胞的控制仍然进展缓慢. 近年来, 随着合成生物学技术的发展, 基于细胞—计算机交互的胞外控制手段开始引起研究者们的关注, 为细胞控制带来了前所未有的机遇. 胞机交互的方式能够适应生物系统的特殊性, 发挥计算机控制的优势, 实现细胞的自动化实时控制, 为人类研究细胞内部基因调控机制与其他各项生命活动提供了大量的数据与方法支持. 本文根据目前基于胞机交互的细胞控制工作, 归纳与总结了胞机交互中常用的生物学工具以及控制算法, 分析了细胞控制的特殊性与难点, 指出研究实现细胞智能控制的可行性与重要性.
Abstract:Wiener emphasized two major categories of control objects in cybernetics: machines and animals. In the past half century, a relatively complete and advanced control theory has been formed in the field of machine control. However, due to the particularity and complexity of biological systems, the control of cell, the basic unit of life, is still progressing slowly. In recent years, with the development of synthetic biology tools, in silico control methods based on cell-computer interfacing has drawn researchers' attention, bringing unprecedented opportunities for cell control. These methods can adapt to the particularity of biological system, take advantages of in silico control, realize automatic real-time cell control and provide a large amount of data and methods for human research on endogenous gene regulation mechanism or other activities. Based on current progress, this review summarized the biological tools and control algorithms used in cell-machine interfacing, illustrated the particularities and difficulties and pointed out the feasibility and importance to achieve intelligent cell control.
-
Key words:
- Cell-computer interfacing /
- feedback control /
- synthetic biology /
- artificial biosystem /
- optogenetics
-
表 1 细胞控制常用控制算法优缺点比较
Table 1 Pros and cons of several control algorithms in cell control
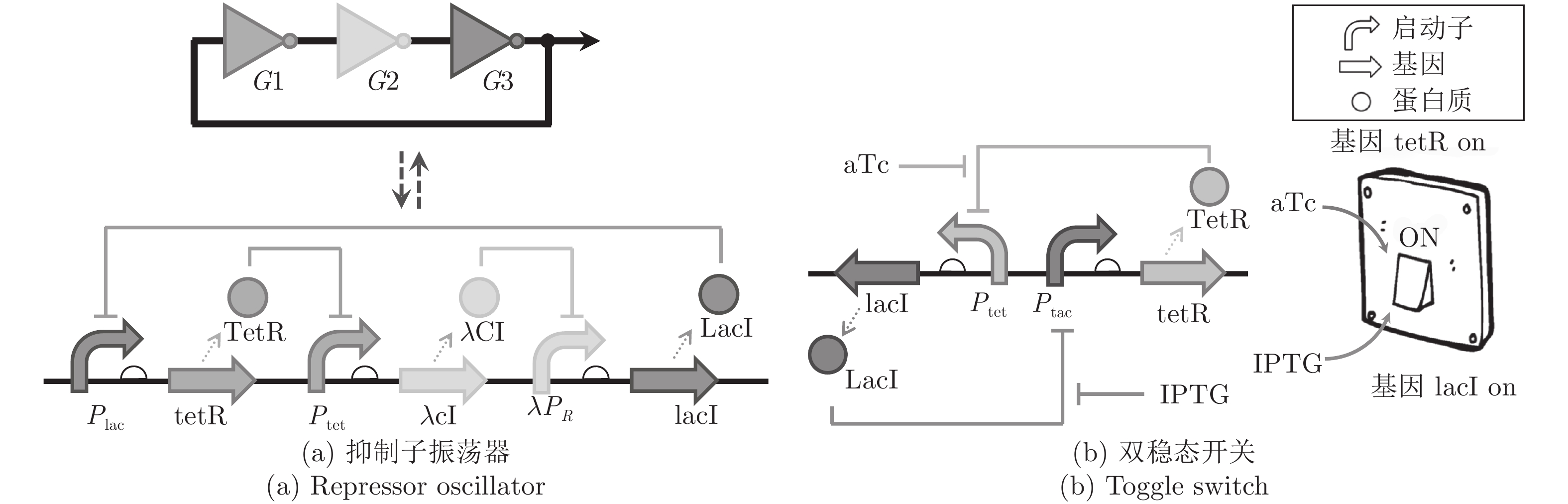
控制算法 是否需要精确建模 优点 缺点 PID控制 否 稳定性好, 计算简便, 不依赖模型 在快速变化的、长时延的系统上效果较差 模型预测控制 是 适用于时变的、有时延的系统, 能够预测未来状态 计算复杂度高, 易受噪声影响, 建模过程繁琐 起停式控制 否 结构最简单方便 控制动作不连续, 容易造成系统振荡 ZAD控制 是 适用于时变的、有时延的系统, 减少了输入开关数量 在快速变化系统中的表现略逊于模型预测控制[20] -
[1] Wiener N. Cybernetics: Or Control and Communication in the Animal and the Machine. Cambridge: MIT Press, 1965. [2] Rao C V, Wolf D M, Arkin A P. Control, exploitation and tolerance of intracellular noise. Nature, 2002, 420(6912): 231−237 doi: 10.1038/nature01258 [3] Kærn M, Elston T C, Blake W J, Collins J J. Stochasticity in gene expression: From theories to phenotypes. Nature Reviews Genetics, 2005, 6(6): 451−464 doi: 10.1038/nrg1615 [4] Elowitz M B, Leibler S. A synthetic oscillatory network of transcriptional regulators. Nature, 2000, 403(6767): 335−338 doi: 10.1038/35002125 [5] Gardner T S, Cantor C R, Collins J J. Construction of a genetic toggle switch in Escherichia coli. Nature, 2000, 403(6767): 339−342 doi: 10.1038/35002131 [6] Ceroni F, Boo A, Furini S, Gorochowski T E, Borkowski O, Ladak Y N, et al. Burden-driven feedback control of gene expression. Nature Methods, 2018, 15(5): 387−393 doi: 10.1038/nmeth.4635 [7] Briat C, Gupta A, Khammash M. Antithetic integral feedback ensures robust perfect adaptation in noisy biomolecular networks. Cell Systems, 2016, 2(1): 15−26 doi: 10.1016/j.cels.2016.01.004 [8] Briat C, Gupta A, Khammash M. Antithetic proportional-integral feedback for reduced variance and improved control performance of stochastic reaction networks. Journal of The Royal Society Interface, 2018, 15(143): 20180079 doi: 10.1098/rsif.2018.0079 [9] Aoki S K, Lillacci G, Gupta A, Baumschlager A, Schweingruber D, Khammash M. A universal biomolecular integral feedback controller for robust perfect adaptation. Nature, 2019, 570(7762): 533−537 doi: 10.1038/s41586-019-1321-1 [10] Del Vecchio D, Abdallah H, Qian Y L, Collins J J. A blueprint for a synthetic genetic feedback controller to reprogram cell fate. Cell Systems, 2017, 4(1): 109−120 doi: 10.1016/j.cels.2016.12.001 [11] Shopera T, Henson W R, Ng A, Lee Y J, Ng K, Moon T S. Robust, tunable genetic memory from protein sequestration combined with positive feedback. Nucleic Acids Research, 2015, 43(18): 9086−9094 doi: 10.1093/nar/gkv936 [12] Gao X J, Chong L S, Kim M S, Elowitz M B. Programmable protein circuits in living cells. Science, 2018, 361(6408): 1252−1258 doi: 10.1126/science.aat5062 [13] Fuqua C, Parsek M R, Greenberg E P. Regulation of gene expression by cell-to-cell communication: Acyl-homoserine lactone quorum sensing. Annual Review of Genetics, 2001, 35(1): 439−468 doi: 10.1146/annurev.genet.35.102401.090913 [14] Dunlop M J, Keasling J D, Mukhopadhyay A. A model for improving microbial biofuel production using a synthetic feedback loop. Systems and Synthetic Biology, 2010, 4(2): 95−104 doi: 10.1007/s11693-010-9052-5 [15] Del Vecchio D, Dy A J, Qian Y L. Control theory meets synthetic biology. Journal of the Royal Society Interface, 2016, 13(120): 20160380 doi: 10.1098/rsif.2016.0380 [16] Scott T D, Sweeney K, McClean M N. Biological signal generators: Integrating synthetic biology tools and in silico control. Current Opinion in Systems Biology, 2019, 14: 58−65 doi: 10.1016/j.coisb.2019.02.007 [17] Lugagne J B, Dunlop M J. Cell-machine interfaces for characterizing gene regulatory network dynamics. Current Opinion in Systems Biology, 2019, 14: 1−8 doi: 10.1016/j.coisb.2019.01.001 [18] Menolascina F, Fiore G, Orabona E, De Stefano L, Ferry M, Hasty J, et al. In-vivo real-time control of protein expression from endogenous and synthetic gene networks. PLoS Computational Biology, 2014, 10(5): e1003625 doi: 10.1371/journal.pcbi.1003625 [19] Lugagne J B, Carrillo S S, Kirch M, Köhler A, Batt G, Hersen P. Balancing a genetic toggle switch by real-time feedback control and periodic forcing. Nature Communications, 2017, 8(1): 1671 doi: 10.1038/s41467-017-01498-0 [20] Fiore G, Perrino G, Di Bernardo M, Di Bernardo D. In vivo real-time control of gene expression: A comparative analysis of feedback control strategies in yeast. ACS Synthetic Biology, 2016, 5(2): 154−162 doi: 10.1021/acssynbio.5b00135 [21] Postiglione L, Napolitano S, Pedone E, Rocca D L, Aulicino F, Santorelli M, et al. Regulation of gene expression and signaling pathway activity in mammalian cells by automated microfluidics feedback control. ACS Synthetic Biology, 2018, 7(11): 2558−2565 doi: 10.1021/acssynbio.8b00235 [22] Perrino G, Fiore D, Napolitano S, Di Bernardo M, Di Bernardo D. Towards feedback control of the cell-cycle across a population of yeast cells. In: Proceedings of the 18th European Control Conference. Naples, Italy: IEEE, 2019. 2644−2650 [23] Guarino A, Fiore D, Di Bernardo M. In-silico feedback control of a MIMO synthetic toggle switch via pulse-width modulation. In: Proceedings of the 18th European Control Conference. Naples, Italy: IEEE, 2019. 680−685 [24] Fracassi C, Postiglione L, Fiore G, Di Bernardo D. Automatic control of gene expression in mammalian cells. ACS Synthetic Biology, 2016, 5(4): 296−302 doi: 10.1021/acssynbio.5b00141 [25] Uhlendorf J, Miermont A, Delaveau T, Charvin G, Fages F, Bottani S, et al. Long-term model predictive control of gene expression at the population and single-cell levels. Proceedings of the National Academy of Sciences of the United States of America, 2012, 109(35): 14271−14276 doi: 10.1073/pnas.1206810109 [26] Kurisawa M, Yokoyama M, Okano T. Gene expression control by temperature with thermo-responsive polymeric gene carriers. Journal of Controlled Release, 2000, 69(1): 127−137 doi: 10.1016/S0168-3659(00)00297-2 [27] Milias-Argeitis A, Rullan M, Aoki S K, Buchmann P, Khammash M. Automated optogenetic feedback control for precise and robust regulation of gene expression and cell growth. Nature Communications, 2016, 7: 12546 doi: 10.1038/ncomms12546 [28] Toettcher J E, Gong D, Lim W A, Weiner O D. Light-based feedback for controlling intracellular signaling dynamics. Nature Methods, 2011, 8(10): 837−839 doi: 10.1038/nmeth.1700 [29] Briat C, Khammash M. In-Silico proportional-integral moment control of stochastic gene expression. arXiv: 1810.12293, 2018. [30] Harrigan P, Madhani H D, El-Samad H. Real-time genetic compensation defines the dynamic demands of feedback control. Cell, 2018, 175(3): 877−886 doi: 10.1016/j.cell.2018.09.044 [31] Milias-Argeitis A, Summers S, Stewart-Ornstein J, Zuleta I, Pincus D, El-Samad H, et al. In silico feedback for in vivo regulation of a gene expression circuit. Nature Biotechnology, 2011, 29(12): 1114−1116 [32] Chait R, Ruess J, Bergmiller T, Tkačik G, Guet C C. Shaping bacterial population behavior through computer-interfaced control of individual cells. Nature Communications, 2017, 8(1): 1535 doi: 10.1038/s41467-017-01683-1 [33] Melendez J, Patel M, Oakes B L, Xu P, Morton P, McClean M N. Real-time optogenetic control of intracellular protein concentration in microbial cell cultures. Integrative Biology, 2014, 6(3): 366−372 doi: 10.1039/c3ib40102b [34] Olson E J, Hartsough L A, Landry B P, Shroff R, Tabor J J. Characterizing bacterial gene circuit dynamics with optically programmed gene expression signals. Nature Methods, 2014, 11(4): 449−455 doi: 10.1038/nmeth.2884 [35] Olson E J, Tzouanas C N, Tabor J J. A photoconversion model for full spectral programming and multiplexing of optogenetic systems. Molecular Systems Biology, 2017, 13(4): 926 doi: 10.15252/msb.20167456 [36] Rullan M, Benzinger D, Schmidt G W, Milias-Argeitis A, Khammash M. An optogenetic platform for real-time, single-cell interrogation of stochastic transcriptional regulation. Molecular Cell, 2018, 70(4): 745−756 doi: 10.1016/j.molcel.2018.04.012 [37] Brophy J A N, Voigt C A. Principles of genetic circuit design. Nature Methods, 2014, 11(5): 508−520 doi: 10.1038/nmeth.2926 [38] Khalil A S, Collins J J. Synthetic biology: Applications come of age. Nature Reviews Genetics, 2010, 11(5): 367−379 doi: 10.1038/nrg2775 [39] Zheng H, Ho P Y, Jiang M L, Tang B, Liu W R, Li D J, et al. Interrogating the Escherichia coli cell cycle by cell dimension perturbations. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(52): 15000−15005 doi: 10.1073/pnas.1617932114 [40] Meshik X, O'Neill P R, Gautam N. Physical plasma membrane perturbation using subcellular optogenetics drives integrin-activated cell migration. ACS Synthetic Biology, 2019, 8(3): 498−510 doi: 10.1021/acssynbio.8b00356 [41] Scholz O, Thiel A, Hillen W, Niederweis M. Quantitative analysis of gene expression with an improved green fluorescent protein. European Journal of Biochemistry, 2000, 267(6): 1565−1570 doi: 10.1046/j.1432-1327.2000.01170.x [42] Yi J R, Wu P X, Huang Q Y, Qu H, Liu B, Hoeppner D J, et al. Multi-scale cell instance segmentation with keypoint graph based bounding boxes. In: Proceedings of the 2019 International Conference on Medical Image Computing and Computer-Assisted Intervention. Shenzhen, China: Springer, 2019. 369−377 [43] Zhou Z B, Wang F, Xi W J, Chen H Y, Gao P, He C K. Joint multi-frame detection and segmentation for multi-cell tracking. arXiv: 1906.10886, 2019. [44] Luo W H, Xing J L, Milan A, Zhang X Q, Liu W, Zhao X W, et al. Multiple object tracking: A literature review. arXiv: 1409.7618, 2014. [45] Ciaparrone G, Sánchez F L, Tabik S, Troiano L, Tagliaferri R, Herrera F. Deep learning in video multi-object tracking: A survey. arXiv: 1907.12740, 2019. [46] Ruzicka J. Flow injection analysis. From test tube to integrated microconduits. Analytical Chemistry, 1983, 55(11): 1041A−1053A [47] Wang P, Robert L, Pelletier J, Dang W L, Taddei F, Wright A, et al. Robust growth of Escherichia coli. Current Biology, 2010, 20(12): 1099−1103 doi: 10.1016/j.cub.2010.04.045 [48] Rosenthal A Z, Qi Y T, Hormoz S, Park J, Li S H J, Elowitz M B. Metabolic interactions between dynamic bacterial subpopulations. eLife, 2018, 7: e33099 doi: 10.7554/eLife.33099 [49] Park J, Dies M, Lin Y H, Hormoz S, Smith-Unna S E, Quinodoz S, et al. Molecular time sharing through dynamic pulsing in single cells. Cell Systems, 2018, 6(2): 216−229 doi: 10.1016/j.cels.2018.01.011 [50] Kempner M E, Felder R A. A review of cell culture automation. Journal of the Association for Laboratory Automation, 2002, 7(2): 56−62 doi: 10.1016/S1535-5535-04-00183-2 [51] Frey O, Rudolf F, Schmidt G W, Hierlemann A. Versatile, simple-to-use microfluidic cell-culturing chip for long-term, high-resolution, time-lapse imaging. Analytical Chemistry, 2015, 87(8): 4144−4151 doi: 10.1021/ac504611t [52] Jo M C, Qin L D. Microfluidic platforms for yeast-based aging studies. Small, 2016, 12(42): 5787−5801 doi: 10.1002/smll.201602006 [53] Marshall J, Qiao X, Baumbach J, Xie J Y, Dong L, Bhattacharyya M K. Microfluidic device enabled quantitative time-lapse microscopic-photography for phenotyping vegetative and reproductive phases in Fusarium virguliforme, which is pathogenic to soybean. Scientific Reports, 2017, 7: 44365 doi: 10.1038/srep44365 [54] Din M O, Danino T, Prindle A, Skalak M, Selimkhanov J, Allen K, et al. Synchronized cycles of bacterial lysis for in vivo delivery. Nature, 2016, 536(7614): 81−85 doi: 10.1038/nature18930 [55] Gottesfeld J M, Neely L, Trauger J W, Baird E E, Dervan P B. Regulation of gene expression by small molecules. Nature, 1997, 387(6629): 202−205 doi: 10.1038/387202a0 [56] Dahl R H, Zhang F Z, Alonso-Gutierrez J, Baidoo E, Batth T S, Redding-Johanson A M, et al. Engineering dynamic pathway regulation using stress-response promoters. Nature Biotechnology, 2013, 31(11): 1039−1046 doi: 10.1038/nbt.2689 [57] Qin S Y, Yin H, Yang C L, Dou Y F, Liu Z M, Zhang P, et al. A magnetic protein biocompass. Nature Materials, 2016, 15(2): 217−226 doi: 10.1038/nmat4484 [58] Mannix R J, Kumar S, Cassiola F, Montoya-Zavala M, Feinstein E, Prentiss M, et al. Nanomagnetic actuation of receptor-mediated signal transduction. Nature Nanotechnology, 2007, 3(1): 36−40 [59] Tseng P, Judy J W, Di Carlo D. Magnetic nanoparticle-mediated massively parallel mechanical modulation of single-cell behavior. Nature Methods, 2012, 9(11): 1113−1119 doi: 10.1038/nmeth.2210 [60] Clites B L, Pierce J T. Identifying cellular and molecular mechanisms for magnetosensation. Annual Review of Neuroscience, 2017, 40: 231−250 doi: 10.1146/annurev-neuro-072116-031312 [61] Rahmani A, Nadri S, Kazemi H S, Mortazavi Y, Sojoodi M. Conductive electrospun scaffolds with electrical stimulation for neural differentiation of conjunctiva mesenchymal stem cells. Artificial Organs, 2019, 43(8): 780−790 doi: 10.1111/aor.13425 [62] Frank H Y, Catterall W A. The VGL-chanome: A protein superfamily specialized for electrical signaling and ionic homeostasis. Science Signaling, 2004, 2004(253): re15 [63] Wang X, Chen X J, Yang Y. Spatiotemporal control of gene expression by a light-switchable transgene system. Nature Methods, 2012, 9(3): 266−269 doi: 10.1038/nmeth.1892 [64] Pathak G P, Spiltoir J I, Höglund C, Polstein L R, Heine-Koskinen S, Gersbach C A, et al. Bidirectional approaches for optogenetic regulation of gene expression in mammalian cells using Arabidopsis cryptochrome 2. Nucleic Acids Research, 2017, 45(20): e167 doi: 10.1093/nar/gkx260 [65] Klimas A, Ambrosi C M, Yu J Z, Williams J C, Bien H, Entcheva E. OptoDyCE as an automated system for high-throughput all-optical dynamic cardiac electrophysiology. Nature Communications, 2016, 7: 11542 doi: 10.1038/ncomms11542 [66] Zhang K, Cui B X. Optogenetic control of intracellular signaling pathways. Trends in Biotechnology, 2015, 33(2): 92−100 doi: 10.1016/j.tibtech.2014.11.007 [67] Levskaya A, Chevalier A A, Tabor J J, Simpson Z B, Lavery L A, Levy M, et al. Synthetic biology: Engineering Escherichia coli to see light. Nature, 2005, 438(7067): 441−442 doi: 10.1038/nature04405 [68] Shimizu-Sato S, Huq E, Tepperman J M, Quail P H. A light-switchable gene promoter system. Nature Biotechnology, 2002, 20(10): 1041−1044 doi: 10.1038/nbt734 [69] Müller K, Weber W. Optogenetic tools for mammalian systems. Molecular BioSystems, 2013, 9(4): 596−608 doi: 10.1039/c3mb25590e [70] Repina N A, Rosenbloom A, Mukherjee A, Schaffer D V, Kane R S. At light speed: Advances in optogenetic systems for regulating cell signaling and behavior. Annual Review of Chemical and Biomolecular Engineering, 2017, 8(1): 13−39 doi: 10.1146/annurev-chembioeng-060816-101254 [71] Liu Z D, Zhang J Z, Jin J, Geng Z L, Qi Q S, Liang Q F. Programming bacteria with light-sensors and applications in synthetic biology. Frontiers in Microbiology, 2018, 9: 2692 doi: 10.3389/fmicb.2018.02692 [72] Jayaraman P, Devarajan K, Chua T K, Zhang H Z, Gunawan E, Poh C L. Blue light-mediated transcriptional activation and repression of gene expression in bacteria. Nucleic Acids Research, 2016, 44(14): 6994−7005 doi: 10.1093/nar/gkw548 [73] Ramakrishnan P, Tabor J J. Repurposing synechocystis PCC6803 UirS-UirR as a UV-violet/green photoreversible transcriptional regulatory tool in E.coli. ACS Synthetic Biology, 2016, 5(7): 733−740 doi: 10.1021/acssynbio.6b00068 [74] Möglich A, Ayers R A, Moffat K. Design and signaling mechanism of light-regulated histidine kinases. Journal of Molecular Biology, 2009, 385(5): 1433−1444 doi: 10.1016/j.jmb.2008.12.017 [75] Hirose Y, Shimada T, Narikawa R, Katayama M, Lkeuchi M. Cyanobacteriochrome CcaS is the green light receptor that induces the expression of phycobilisome linker protein. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(28): 9528−9533 doi: 10.1073/pnas.0801826105 [76] Fernandez-Rodriguez J, Moser F, Song M, Voigt C A. Engineering RGB color vision into Escherichia coli. Nature Chemical Biology, 2017, 13(7): 706−708 doi: 10.1038/nchembio.2390 [77] Müller K, Engesser R, Timmer J, Zurbriggen M D, Weber W. Orthogonal optogenetic triple-gene control in mammalian cells. ACS Synthetic Biology, 2014, 3(11): 796−801 doi: 10.1021/sb500305v [78] 杨汶娟, 金爱娟, 李少龙. 零平均动态控制算法的研究. 信息技术, 2014, (5): 79−81 doi: 10.3969/j.issn.1009-2552.2014.05.023Yang Wen-Juan, Jin Ai-Juan, Li Shao-Long. An algorithm of zero average dynamic control. Information Technology, 2014, (5): 79−81 doi: 10.3969/j.issn.1009-2552.2014.05.023 [79] Bowsher C G, Swain P S. Environmental sensing, information transfer, and cellular decision-making. Current Opinion in Biotechnology, 2014, 28: 149−155 doi: 10.1016/j.copbio.2014.04.010 [80] Kothare M V, Campo P J, Morari M, Nett C N. A unified framework for the study of anti-windup designs. Automatica, 1994, 30(12): 1869−1883 doi: 10.1016/0005-1098(94)90048-5 [81] Zhu P X, Fajardo O, Shum J, Schärer Y P Z, Friedrich R W. High-resolution optical control of spatiotemporal neuronal activity patterns in zebrafish using a digital micromirror device. Nature Protocols, 2012, 7(7): 1410−1425 doi: 10.1038/nprot.2012.072 [82] 王沛, 吕金虎. 基因调控网络的控制: 机遇与挑战. 自动化学报, 2013, 39(12): 1969−1979Wang Pei, Lv Jin-Hu. Control of genetic regulatory networks: Opportunities and challenges. Acta Automatica Sinica, 2013, 39(12): 1969−1979 [83] Schrödinger E. What is Life? The Physical Aspect of the Living Cell. Cambridge: Cambridge University Press, 1944. [84] 雷锦志. 系统生物学: 建模, 分析, 模拟. 上海: 上海科学技术出版社, 2010.Lei Jin-Zhi. Systems Biology-Modelling, Analysis, Simulation. Shanghai: Shanghai Science and Technology Press, 2010. [85] Yuan Y, Liu B, Xie P, Zhang M Q, Li Y D, Xie Z, et al. Model-guided quantitative analysis of microRNA-mediated regulation on competing endogenous RNAs using a synthetic gene circuit. Proceedings of the National Academy of Sciences, 2015, 112(10): 3158−3163 doi: 10.1073/pnas.1413896112 [86] Wei L, Yuan Y, Hu T, Li S L, Cheng T R, Lei J Z, et al. Regulation by competition: A hidden layer of gene regulatory network. Quantitative Biology, 2019, 7(2): 110−121 doi: 10.1007/s40484-018-0162-5 [87] 刘向杰, 周孝信, 柴天佑. 模糊控制研究的现状与新发展. 信息与控制, 1999, 28(4): 283−292 doi: 10.3969/j.issn.1002-0411.1999.04.008Liu Xiang-Jie, Zhou Xiao-Xin, Chai Tian-You. Status and development of fuzzy control. Information and Control, 1999, 28(4): 283−292 doi: 10.3969/j.issn.1002-0411.1999.04.008 [88] Yao X M, Guo L. Composite anti-disturbance control for Markovian jump nonlinear systems via disturbance observer. Automatica, 2013, 49(8): 2538−2545 doi: 10.1016/j.automatica.2013.05.002 [89] Neftci E O, Averbeck B B. Reinforcement learning in artificial and biological systems. Nature Machine Intelligence, 2019, 1(3): 133−143 doi: 10.1038/s42256-019-0025-4 [90] Chevalier M, Gómez-Schiavon M, Ng A H, El-Samad H. Design and analysis of a proportional-integral-derivative controller with biological molecules. Cell Systems, 2019, 9(4): 338−353 doi: 10.1016/j.cels.2019.08.010 [91] Qiao L X, Zhao W, Tang C, Nie Q, Zhang L. Network topologies that can achieve dual function of adaptation and noise attenuation. Cell Systems, 2019, 9(3): 271−285 doi: 10.1016/j.cels.2019.08.006 [92] Bashivan P, Kar K, DiCarlo J J. Neural population control via deep image synthesis. Science, 2019, 364(6439): eaav9436 doi: 10.1126/science.aav9436 [93] Ponce C R, Xiao W, Schade P F, Hartmann T S, Kreiman G, Livingstone M S. Evolving images for visual neurons using a deep generative network reveals coding principles and neuronal preferences. Cell, 2019, 177(4): 999−1009 doi: 10.1016/j.cell.2019.04.005 [94] Ye H F, Baba M D E, Peng R W, Fussenegger M. A synthetic optogenetic transcription device enhances blood-glucose homeostasis in mice. Science, 2011, 332(6037): 1565−1568 doi: 10.1126/science.1203535 [95] Shao J W, Xue S, Yu G L, Yu Y H, Yang X P, Bai Y, et al. Smartphone-controlled optogenetically engineered cells enable semiautomatic glucose homeostasis in diabetic mice. Science Translational Medicine, 2017, 9(387): eaal2298 doi: 10.1126/scitranslmed.aal2298 [96] Chen X J, Liu R M, Ma Z C, Xu X P, Zhang H Q, Xu J H, et al. An extraordinary stringent and sensitive light-switchable gene expression system for bacterial cells. Cell Research, 2016, 26(7): 854-857 [97] Liang J H, Hu Y Q, Chen G R, Zhou T S. A universal indicator of critical state transitions in noisy complex networked systems. Scientific Reports, 2017, 7: 42857 doi: 10.1038/srep42857 [98] Zhou T S, Chen L N, Aihara K. Molecular communication through stochastic synchronization induced by extracellular fluctuations. Physical Review Letters, 2005, 95(17): 178103 doi: 10.1103/PhysRevLett.95.178103 [99] Zhang J J, Yuan Z J, Zhou T S. Physical limits of feedback noise-suppression in biological networks. Physical Biology, 2009, 6(4): 046009 doi: 10.1088/1478-3975/6/4/046009 [100] Gu H B, Lv J H, Lin Z L. On PID control for synchronization of complex dynamical network with delayed nodes. Science China Technological Sciences, 2019, 62(8): 1412−1422 doi: 10.1007/s11431-018-9379-8 [101] Deisseroth K. Optogenetics: 10 years of microbial opsins in neuroscience. Nature Neuroscience, 2015, 18(9): 1213−1225 doi: 10.1038/nn.4091 [102] Ma G L, Liu J D, Ke Y P, Liu X, Li M Y, Wang F, et al. Optogenetic control of voltage-gated calcium channels. Angewandte Chemie International Edition, 2018, 57(24): 7019−7022 doi: 10.1002/anie.201713080 [103] Chaffiol A, Caplette R, Jaillard C, Brazhnikova E, Desrosiers M, Dubus E, et al. A new promoter allows optogenetic vision restoration with enhanced sensitivity in macaque retina. Molecular Therapy, 2017, 25(11): 2546−2560 doi: 10.1016/j.ymthe.2017.07.011 [104] Chernet B T, Adams D S, Lobikin M, Levin M. Use of genetically encoded, light-gated ion translocators to control tumorigenesis. Oncotarget, 2016, 7(15): 19575−19588 doi: 10.18632/oncotarget.8036 [105] Chen S, Weitemier A Z, Zeng X, He L M, Wang X Y, Tao Y Q, et al. Near-infrared deep brain stimulation via upconversion nanoparticle-mediated optogenetics. Science, 2018, 359(6376): 679−684 doi: 10.1126/science.aaq1144 -




 下载:
下载: