An Overview of Identification Methods on Human Brain Effective Connectivity Networks Based on Functional Magnetic Resonance Imaging
-
摘要: 人脑效应连接网络刻画了脑区间神经活动的因果效应. 对不同人群的脑效应连接网络进行研究不仅能为神经精神疾病病理机制的理解提供新视角, 而且能为疾病的早期诊断和治疗评价提供新的脑网络影像学标记, 具有十分重要的理论意义和应用价值. 利用计算方法从功能磁共振成像(Functional magnetic resonance imaging, fMRI)数据中识别脑效应连接网络是目前人脑连接组学中一项重要的研究课题. 本文首先概括了从fMRI数据中进行脑效应连接网络识别的主要流程, 说明了其中的主要步骤和方法; 然后, 给出了一种脑效应连接网络识别方法的分类体系, 并对其中一些代表性的识别算法进行了阐述; 最后, 通过对该领域挑战性问题的分析, 预测了脑效应连接网络识别未来的研究方向, 以期对相关研究提供一定的参考.Abstract: The brain effective connectivity networks characterize the causal interactions of neural activity between brain regions. Researches on brain effective connectivity networks of different populations can not only provide a new perspective for understanding the pathological mechanism of neuropsychiatric diseases, but also provide novel brain network imaging markers for the early diagnosis and evaluation for treatment of diseases, thus have very important theoretical and practical value. Using computational approaches to identify brain effective connectivity networks from functional magnetic resonance imaging (fMRI) data is currently an important subject in the human brain connectome. This paper firstly summarizes a workflow of identifying brain effective connectivity networks from fMRI data and illustrates its main processes and methods. Next, a comprehensive category system of identifying brain effective connectivity networks is presented, and several typical identifying algorithms in each category are described. Finally, by analyzing challenging problems in this area, we predict the further research directions in identifying brain effective connectivity networks and hope to present some references for related researches.
-
在深海勘探开发生产中, 海洋柔性立管作为连接海面作业平台与海床井口的关键构件[1].在风、浪、洋流等外部载荷作用下, 海洋立管会产生振动现象, 而长期的振动则是造成柔性立管疲劳破损的主因[2-4].因此, 开展先进的海洋柔性立管振动主动控制系统研究, 对延长立管使用寿命、提高生产效率和保证海洋油气生产安全具有重要的理论和实际意义.
从数学的观点看, 具有振动的海洋柔性立管系统可认为是典型的无限维分布参数系统[5-11].其动力学往往建模为耦合的偏微分–常微分方程, 这使得现有许多对传统刚性系统成熟的方法不能直接应用.对海洋柔性立管振动控制的研究主要包括模态控制和边界控制.模态控制是基于提取的有限维受控子系统进行控制设计, 而忽略掉的高频模态可能导致系统产生控制溢出效应.边界控制能克服上述方法的缺点, 且容易由系统机械能相关的Lyapunov函数得出, 因此边界控制与其他控制技术如PID控制、鲁棒控制、自适应控制、反步控制、输出反馈控制等相结合的方法广泛应用于柔性立管系统的振动控制领域[12-16].上述研究仅仅局限于柔性立管系统的振动控制, 而这些方法将不适用于具有输入非线性特性的柔性立管系统.
在实际的海洋油气生产环境中, 柔性立管系统除了受到风浪扰动和海洋洋流分布式扰动影响外, 其面临的情况可能会比之前研究的问题更加复杂.如系统固有的物理约束和执行器的约束将使得系统产生死区、饱和、磁滞、反向间隙等不光滑的非线性特性[17-20].而这些不光滑的非线性特性将会限制系统的瞬态性能, 更为甚者, 将会致使系统不稳定.因此, 需要将这些不光滑的非线性约束特性考虑在控制设计中.为了解决海洋柔性立管系统的输入非线性约束问题, 一些学者基于立管原始无限维模型探索了不同的边界控制方法[13, 21-24].文献[13]面向具有系统不确定性、输出约束和输入饱和的海洋立管系统, 基于反推技术研发了障碍边界控制策略以抑制振动、补偿系统不确定性以及处理系统的输入输出限制.文献[21]针对具有执行器输入饱和非线性约束和外部海洋扰动的海洋柔性立管系统, 在顶端构建边界控制器以稳定其在平衡位置的小邻域并利用辅助系统补偿执行器饱和的影响.文献[22]设计了鲁棒自适应控制器用以稳定具有参数不确定性和输入受限的海洋柔性立管系统.文献[23]采用光滑的双曲正切函数、Nussbaum函数和辅助系统设计边界控制器以抑制立管振动并限制控制输入在给定范围内, 该方法解决了文献[21−22]中应用符号函数限制控制输入所带来的震颤问题.文献[24]引入辅助函数和变量设计边界控制器来实现立管的振动减弱并消除混合的死区−饱和非线性约束影响.然而, 这些成果仅仅解决了柔性立管系统执行器输入饱和或输入饱和−死区非线性约束问题, 而对于具有输入反向间隙−饱和非线性约束的柔性立管系统, 上述方法将不能适用.
本文针对执行器非光滑反向间隙−饱和约束特性的深海柔性立管系统(如图 1所示), 首先将反向间隙−饱和约束转换成虚拟的输入饱和约束, 其后引入辅助系统并采用Lyapunov理论, 构建边界控制以抑制柔性立管的振动并消除饱和非线性约束的影响.随后, 证明了闭环系统在Lyapunov意义下的一致有界稳定性.最后, 通过数值仿真, 验证了本文所提出控制能处理非光滑反向间隙−饱和约束非线性影响, 也能有效抑制立管系统振动.
1. 问题描述和预备知识
注1.本文作如下简写: $ (\cdot)(x, t) = (\cdot) $, $ (\cdot)' = \dfrac{\partial(\cdot)}{\partial{x}} $, $ \dot{(\cdot)} $ = $ \dfrac{\partial(\cdot)}{\partial{t}} $.
1.1 立管动力学模型
深海柔性立管系统如图 1所示, 其中$ l $为立管的长度, $ y(z, t) $为立管在位置$ z $时刻$ t $的偏移量, $ f(z, t) $为海洋洋流分布式扰动, $ d(t) $为外部环境扰动, $ u(t) $为边界控制输入.
本研究所考虑立管系统动力学描述如下[1]:
$\begin{split} \rho\ddot{y}(z,&t)-\left\{T[z, y'(z,t)]+\right.\\ & \left.3\psi(z)y'^2(z,t)\right\}y''(z,t)-\\ & \ T'[z, y'(z,t), y''(z,t)]y'(z,t)+c\dot{y}(z,t)-\\ & \ \psi'(z)y'^3(z,t)+ EIy''''(z,t)-\\ & \ f(z,t) = 0,\ \ \ 0<z<l \end{split} \hspace{33pt} $
(1) $ \begin{align} y(0,t) = y'(0,t) = y''(l,t) = 0 \end{align} \hspace{78pt} $
(2) $ \begin{split} m\ddot{y}(l,t)+& T[l, y'(l,t)]y'(l,t)+\psi(l)y'^3(l,t)-u(t)+\\ & d_a\dot{y}(l,t) = EIy'''(l,t)+d(t) \end{split} \hspace{5pt}$
(3) 其中, $ \rho $, $ c $和$ EI $分别为立管的单位长度质量、阻尼系数和弯曲刚度, $ d_a $和$ m $为船的质量和阻尼系数, $ T[z, y'(z, t)] $为立管的时空变化张力, 表示为
$ \begin{align} T[z, y'(z,t)] = T_0(z)+\psi(z)y'^2(z,t) \end{align} $
(4) 其中, $ T_0(z) > 0 $为初始张力, $ \psi(z)\ge 0 $为非线性弹性模量.
执行器输入饱和非线性描述为[25]
$ \begin{align} \varphi(t) = sat(\varrho(t)) = \begin{cases} a , \qquad\quad\ \varrho(t)\ge a \\[2mm] \varrho(t) , \qquad -a < \varrho(t) < a \\[2mm] -a, \qquad\ \ \, \varrho(t)\ -a \end{cases} \end{align} $
(5) 其中, $ a > 0 $为饱和界限.
执行器输入反向间隙非线性描述为[24]
$\begin{array}{l} u(t) = D(\varphi (t)) = \\ \qquad\;\;\;\left\{ {\begin{aligned} &{\varphi (t) - b,\;\qquad \dot \varphi }{ > 0\;\text{且}\;u(t) = \varphi (t) - b}\\ &{\varphi (t) + b,\;\qquad \dot \varphi }{ < 0\;\text{且}\;u(t) = \varphi (t) + b}\\ &{u(t\_), \qquad\quad\;\; \text{其他}}&{} \end{aligned}} \right. \end{array}$
(6) 其中, $ b > 0 $为反向间隙参数.
由输入饱和与反向间隙的表达式(5)和式(6)可知, 系统的非线性特征是相当复杂的, 因此很难直接对其处理.根据文献[25], 可知输入饱和与反向间隙可转换并表示为一个虚拟的输入饱和.因此, 为解决虚拟的输入非线性问题, 我们引入$ D $的右逆$ D^+ $为
$ \begin{align} \varrho(t) = D^+(\tau(t)) = \left\{ \begin{aligned} & \tau(t)+b, \; \; \dot{\tau}(t)>0 \\ & \tau(t)-b, \; \; \dot{\tau}(t)<0 \\ & \varrho(t\_), \; \;\;\;\;\; \dot{\tau}(t) = 0 \end{aligned} \right. \end{align} $
(7) 根据上面的分析和文献[25], 我们可得混合的输入饱和−反向间隙非线性特性可描述为
$ \begin{split} u(t) = & D(sat(D^+(\tau(t)))) = \\ &\left\{ \begin{aligned} & \,a-b, \qquad\;\;\, \tau(t)\ge a-b \\ & \, \tau(t), \qquad\quad\; |\tau(t)|<a-b \\ & -a+b, \quad\;\;\tau(t)\le-a+b \end{aligned} \right. \end{split} $
(8) 由式(8)可知, 我们可将系统的输入饱和−反向间隙非线性视为一个输入饱和来处理.
1.2 预备知识
引理1[26].设$ \chi_1(z, t) $, $ \chi_2(z, t)\in {\bf{R}} $, $ \varphi > 0 $, 其中$ (z, t)\in$ $[0, l]\times[0, +\infty) $, 则
$ \begin{align} \chi_1(z,t)\chi_2(z,t)\le \frac{1}{\varphi}\chi^2_1(z,t)+\varphi\chi^2_2(z,t) \end{align} $
(9) 引理2[26].设$ \chi(z, t)\in {\bf{R}} $为定义在$ (z, t)\in[0, l]\times $ $[0, +\infty) $的函数, 且满足$ \chi(0, t) = 0, \forall t\in[0, +\infty) $, 则
$ \begin{align} \chi^2(z,t) \le l\int^l_0\chi^{{\prime}2}(z,t){\rm{d}}z \end{align} $
(10) 假设1.假定存在常数$ {F} $, $ {D}\in {\bf{R}}^+ $, 使得$\mid f(z, t)\mid \leq $ $ {F, } $ $ \forall{(z, t)}\in{[0, l]\times[0, +\infty), } $ $\mid d(t)\mid \leq {D, } $ $ \forall{t}\in[0, +\infty). $这个假设是合理的, 由于$ f(z, t) $和$ d(t) $是有限能量的, 因此是有界的[21-24].
假设2.假定存在正常数$ \underline{T}_0 $, $ \overline{T}_0 $, $ \underline{\psi}_0 $, $ \overline{\psi}_0 $, 使得$ \underline{T}_0\le T_0(z) \le \overline{T}_0 $, $ \underline{\psi}_0\le \psi(z)\leq \overline{\psi}_0 $.
假设3.对于新的输入饱和表达式(8), 假定存在一个正常数$ \varpi $使得$ |\triangle u|\le \varpi $, 其中, $ \triangle u = u(t)-$ $\tau(t) $.
2. 控制器设计
本节将引入辅助函数和辅助系统用于构建边界控制器以抑制立管振动并消除输入非线性影响.
2.1 边界控制器
首先, 设计辅助系统为
$ \begin{split}\! \dot{\nu}(t) =&\ \frac{1}{m}\left(-k_1\nu(t)-\triangle u+T[l, y'(l,t)]y'(l,t)+ \right. \\ & \left. \psi(l)y'^3(l,t)+d_a\dot{y}(l,t)-EIy'''(l, t)\right) \end{split} $
(11) 其中, $ \nu(t) $为辅助系统的状态变量, $ k_1 $为正常数.
为便于分析闭环立管系统的稳定性, 定义如下辅助变量
$\begin{split} \mu(t) =\;& \dot{y}(l,t)-k_2y'''(l, t)+y'(l,t)+\\ &k_3y'^3(l,t)+\nu(t) \end{split}$
(12) 其中, $ k_2, k_3 $为正常数.
对式(12)求导, 代入式(3)和式(11), 可得
$ \begin{aligned} \dot{\mu}(t) =\;& \frac{1}{m}(\tau(t)+d(t)-mk_2\dot{y}'''(l, t)+m\dot{y}'(l,t)+\\ & 3mk_3y'^2(l,t)\dot{y}'(l,t)-k_1\nu(t)) \end{aligned} $
(13) 根据上述分析, 提出控制律$ \tau(t) $为
$ \begin{aligned} \tau(t) = & -k_4\mu(t)+k_1\nu(t)+mk_2\dot{y}'''(l, t)-m\dot{y}'(l,t) -\\ & \ 3mk_3y'^2(l,t)\dot{y}'(l,t)-{\rm{sgn}}(\mu(t)){D} \end{aligned} $
(14) 其中, $ k_4 $为正常数.
注2.所设计的控制器(14)是由可获得的边界信号组成的, 其中$ y'''(l, t) $、$ y'(l, t) $和$ y(l, t) $分别可由剪切力传感器、倾角计和位移传感器获得.此外, 控制器中这些信号的一阶时间微分信号$ \dot{y}'''(l, t), $ $ \dot{y}'(l, t) $和$ \dot{y}(l, t) $分别可对已获得信号进行后向差分算法得到[21-24].
选取如下Lyapunov函数为
$ Y(t) = {{Y}_{e}}(t)+{{Y}_{f}}(t)+{{Y}_{g}}(t) $
(15) 其中,
$ \begin{align} {{Y}_{e}}(t) = \frac{\varsigma}{2}\rho\int_{0}^{l}{{{{\dot{y}}}^{2}}(z,t){\rm{d}}z} +\frac{\varsigma}{2}\int_{0}^{l}T_0(z){{{ {y}^{\prime2}\left( z,t\right) }}{\rm{d}}z}+\\ \frac{\varsigma}{2}\int_{0}^{l}\psi(z){{{ {y}^{\prime4}\left( z,t\right) }}{\rm{d}}z}+\frac{\varsigma}{2}EI\int_{0}^{l}y^{\prime\prime 2}(z,t){\rm{d}}z \end{align} $
(16) $ \begin{align} {{Y}_{g}}(t) = \frac{\varsigma m}{2}\nu^2(t)+\frac{\varsigma m}{2}\mu^2(t) \end{align} \hspace{78pt}$
(17) $ \begin{align} {{Y}_{f}}(t) = \lambda\rho\int_{0}^{l} z \phi(z){\dot{y} (z,t){y}'(z,t){\rm{d}}z} \end{align} $
(18) 其中, $ \varsigma, \lambda > 0 $.
引理3.选取的Lyapunov函数(16)是一个正定的函数:
$ \begin{split} 0\le\; & \delta_1[Y_e(t)+Y_f(t)]\le Y(t)\le \\ &\delta_2[Y_e(t)+Y_f(t)] \end{split} $
(19) 其中, $ \delta_1 > 0, \; \delta_2 > 1 $.
证明.根据引理1, 式(18)可放缩为
$ \begin{split} \mid Y_g(t)\mid\ \le\ & \frac{\lambda\rho \overline{\phi}l}{2}\int^l_0[\dot{y}^2(z,t)+\\ &\ y^{{\prime}2}(z,t)]{\rm{d}}z \le \delta_0{Y_e(t)} \end{split} $
(20) 其中
$ \begin{align} \delta_0 = \frac{\lambda \rho \overline{\phi}l}{\min\left({\varsigma}\rho, {\varsigma}\underline{T_0}\right)} \end{align} $
(21) 通过恰当地选取$ \varsigma $和$ \beta $得出
$ \begin{align} \delta_1 = 1-\delta_0>0, \;\delta_2 = 1+\beta_0>1 \end{align} $
(22) 式(22)表明$ 0 < \delta < 1 $, 应用式(21)可得
$ \begin{align} {\varsigma}>\frac{\lambda \rho \overline{\phi}l}{\min\left(\rho, \underline{T_0}\right)} \end{align} $
(23) 重排式(20), 有
$ \begin{align} -{\delta}Y_e(t)\le Y_g(t)\le {\delta}Y_e(t) \end{align} $
(24) 将式(22)代入式(24)得出
$ \begin{align} 0\le \delta_1 Y_e(t)\le Y_e(t)+Y_g(t)\leq \delta_2 Y_e(t) \end{align} $
(25) 结合式(15), 有
$ \begin{aligned} 0\le\;& \delta_1[Y_e(t)+Y_f(t)]\le Y(t)\leq\\ &\delta_2[Y_e(t)+Y_f(t)] \end{aligned} $
(26) 其中, $ \delta_1 > 0, \; \delta_2 > 1 $.
引理4.选取Lyapunov函数(16)的导数是有上界的:
$ \begin{align} \dot{Y}(t)\le -\delta Y(t)+\alpha \end{align} $
(27) 其中, $ \delta, \alpha > 0 $.
证明.对式(16)求导, 可得:
$ \begin{align} \dot{Y}(t) = \dot{Y}_e(t)+\dot{Y}_f(t)+\dot{Y}_g(t) \end{align} $
(28) 将式(16)求导, 代入式(1)并应用引理1, 可得
$ \begin{aligned} \dot{Y}_e(t)\leq \; &\frac{\varsigma T_0(l)}{2}\mu^2(t)-\frac{\varsigma T_0(l)}{2}\nu^2(t)-\frac{\varsigma T_0(l)}{2}\dot{y}^2(l,t)-\\& \frac{\varsigma T_0(l)k^2_2}{2}y'''^2(l,t)-\frac{\varsigma T_0(l)}{2}y'^2(l,t)-\\ & \frac{\varsigma T_0(l)k^2_3}{2}y'^6(l,t)+{\varsigma T_0(l)}{k_2}\nu(t){y}'''(l,t)-\\ & ({\varsigma EI}-{\varsigma T_0(l)}{k_2})y'''(l,t)\dot{y}(l,t)-\\ & \varsigma k_3T_0(l)y'^4(l,t)-{\varsigma}(c-{\sigma_1})\int^l_0\dot{y}^2(z, t){\rm{d}}z+\\ & (2\varsigma\psi(l)-{\varsigma k_3T_0(l)})y'^3(l,t)\dot{y}(l,t)+\\ &{\varsigma k_2k_3T_0(l)}{y}'''(l,t)y'^3(l,t)-{\varsigma T_0(l)}\nu(t)\dot{y}(l,t)+\\ &{\varsigma k_2T_0(l)}y'''(l,t){y}'(l,t)-{\varsigma k_3T_0(l)}y'^3(l,t)\nu(t)-\\ &{\varsigma T_0(l)}\nu(t){y}'(l,t)+\frac{\varsigma}{\sigma_1} \int^l_0f^2(z,t){\rm{d}}z \end{aligned} $
(29) 其中, $ \delta_1 > 0 $.
对$ Y_f(t) $求导, 代入式(11)和式(14), 应用引理1, 可得
$ \begin{split} \dot{Y}_g(t)\le& -\varsigma k_4\mu^2(t)-\varsigma \nu(t)\triangle u-\varsigma k_1\nu^2(t)+\\& \varsigma T_0(l)\nu(t)y'(l,t)-\varsigma EI \nu(t)y'''(l, t)+\\& 2\varsigma \psi(l)\nu(t)y'^3(l,t)+\varsigma d_a \nu(t)\dot{y}(l,t) \end{split} $
(30) 对$ Y_g(t) $求微分, 代入式(4)并利用引理1, 有
$ \begin{aligned} \dot{Y}_f(t)\le & -l\lambda EI\phi(l) y'''(l,t){y}'(l,t)+\frac{\lambda \rho l\phi(l)}{2}\dot{y}^2(l,t)-\\ &\frac{3\lambda EI}{2}\int^l_0(\phi(z)+z\phi'(z)){y}^{{\prime\prime}2}(z, t){\rm{d}}z-\\ &\left[\frac{\lambda \rho}{2}(\phi(z)+z\phi'(z))-\frac{l\lambda c}{\sigma_2}\right]\int^l_0\dot{y}^2(z, t){\rm{d}}z-\\ &\bigg[\frac{\lambda }{2}(\phi(z)T_0(z)+z\phi'(z)T_0(z)-z\phi(z)T_0'(z))-\\ & {\lambda\sigma_2cl\phi^2(z)}-{\lambda\sigma_3l\phi^2(z)}\bigg]\int^l_0{y}^{{\prime}2}(z, t){\rm{d}}z-\\ & \frac{\lambda }{2}\int^l_0[3\phi(z)\psi'(z)+3z\phi'(z)\psi(z)-\\ &z\phi(z)\psi'(z)]{y}^{{\prime}4}(z, t){\rm{d}}z+\frac{3\lambda \phi(l)\psi(l)l}{2}y'^4(l,t)+\\ & \frac{l\lambda}{\sigma_3} \int^l_0f^2(x,t){\rm{d}}x+\frac{\lambda \phi(l)T_0(l) l}{2}y'^2(l,t) \end{aligned} $
(31) 其中, $ \sigma_2, \sigma_3 > 0 $.
将式(29)和式(30)代入式(28), 应用引理1, 可得
$ \begin{aligned} \dot{Y}(t)\le\;& -\varsigma\left( k_1+\frac{ T_0(l)}{2}-\frac{1}{\sigma_4}-\frac{|T_0(l)k_2-EI|}{2\sigma_5}-\right.\\ &\left.\frac{|T_0(l)-d_a|}{2\sigma_6}-\frac{| k_3T_0(l)-2 \psi(l)|\sigma_9}{2}\right)\nu^2(t)-\\ & \frac{3\lambda EI}{2}\int^l_0(\phi(z)+z\phi'(z)){y}^{{\prime\prime}2}(z, t){\rm{d}}z +\\ &{\varsigma}{\sigma_4}\triangle u^2-\varsigma\left( k_4-\frac{ T_0(l)}{2}\right)\mu^2(t)-\left(\frac{\varsigma T_0(l)}{2}-\right.\\ &\left.\frac{{|\varsigma T_0(l)k_2-l\lambda{EI}\phi(l)|}{\sigma_8}}{2}-\frac{\lambda \phi(l)T_0(l) l}{2}\right)\times\\ &y'^2(l,t)-\left(\varsigma k_3T_0(l)-\frac{3\lambda \phi(l)\psi(l)l}{2}\right)y'^4(l,t)-\\ &\left(\frac{\varsigma T_0(l)}{2}-\right.\frac{{\varsigma|T_0(l)-d_a|}{\sigma_6}}{2}-\\ &\left.\frac{{\varsigma|T_0(l)k_2-EI|}{\sigma_7}}{2}-\frac{{\varsigma|k_3T_0(l)-2\psi(l)|}{\sigma_{10}}}{2}-\right.\\ &\left.\frac{\lambda \rho l\phi(l)}{2}\right)\dot{y}^2(l,t)-\varsigma\left(\frac{ T_0(l)k^2_3}{2}-\right.\\ &\left.\frac{| k_3T_0(l)-2 \psi(l)|}{2\sigma_9}-\frac{ k_2k_3T_0(l)}{2\sigma_{11}}-\right.\\ &\left.\frac{|k_3T_0(l)-2\psi(l)|}{2\sigma_{10}}\right)y'^6(l,t)-\left(\frac{\varsigma T_0(l)}{2}-\right.\\ &\left.\frac{{\varsigma|T_0(l)k_2-EI|}{\sigma_5}}{2}-\frac{{\varsigma|T_0(l)k_2-EI|}}{2{\sigma_7}}-\right.\\ &\left.\frac{{|\varsigma T_0(l)k_2-l\lambda{EI}\phi(l)|}}{2{\sigma_8}}-\frac{\varsigma k_2k_3T_0(l)\sigma_{11}}{2}\right)\times\\ &\left.y'''^2(l,t)-\left[\frac{\lambda }{2}(\phi(z)T_0(z)+z\phi'(z)T_0(z)-\right.\right.\\ &\left.z\phi(z)T_0'(z))-\right.{\lambda\sigma_2cl\phi^2(z)}-{\lambda\sigma_3l\phi^2(z)}\bigg]\\ &\left.\int^l_0{y}^{{\prime}2}(z, t){\rm{d}}z+\left(\frac{\varsigma}{\sigma_1}+\frac{l\lambda}{\sigma_3}\right)\int^l_0f^2(z,t){\rm{d}}z-\right.\\ &\left.\bigg({\varsigma}c-{\varsigma}{\sigma_1}+\frac{\lambda \rho}{2}(\phi(z)+z\phi'(z))-\frac{l\lambda c}{\sigma_2}\right)\times\\ &\int^l_0\dot{y}^2(z, t){\rm{d}}z-\frac{\lambda }{2}\int^l_0[3\phi(z)\psi'(z)+\\ &3z\phi'(z)\psi(z)-z\phi(z)\psi'(z)]{y}^{{\prime}4}(z, t){\rm{d}}z \end{aligned} $
(32) 其中, $ \sigma_4\sim\sigma_{11} > 0, $选择恰当的参数值$ \varsigma, $ $ \lambda, $ $ k_i, $ $ i = 1, $ $\cdots, 4, \delta_j, j = 1, \cdots, 11, $满足下列条件:
$ \begin{split} \frac{\varsigma T_0(l)}{2}-\;&\frac{{|\varsigma T_0(l)k_2-l\lambda{EI}\phi(l)|}{\sigma_8}}{2}-\\ &\frac{\lambda \phi(l)T_0(l) l}{2}\ge 0 \end{split} \hspace{51pt}$
(33) $ \begin{split} \frac{\varsigma T_0(l)}{2}-\;&\frac{{\varsigma|T_0(l)-d_a|}{\sigma_6}}{2}-\frac{{\varsigma|T_0(l)k_2-EI|}{\sigma_7}}{2}-\\ &\frac{{\varsigma|k_3T_0(l)-2\psi(l)|}{\sigma_{10}}}{2}-\frac{\lambda \rho l\phi(l)}{2}\ge 0 \end{split} \hspace{20pt}$
(34) $ \begin{split} \frac{\varsigma T_0(l)}{2}-\;&\frac{{\varsigma|T_0(l)k_2-EI|}{\sigma_5}}{2}-\frac{{\varsigma|T_0(l)k_2-EI|}}{2{\sigma_7}}-\\ &\frac{{|\varsigma T_0(l)k_2-l\lambda{EI}\phi(l)|}}{2{\sigma_8}}-\frac{\varsigma k_2k_3T_0(l)\sigma_{11}}{2}\ge 0 \end{split} $
(35) $ \begin{split} \frac{ T_0(l)k^2_3}{2}-\;&\frac{| k_3T_0(l)-2 \psi(l)|}{2\sigma_9}-\\ &\frac{|k_3T_0(l)-2\psi(l)|}{2\sigma_{10}}-\frac{ k_2k_3T_0(l)}{2\sigma_{11}}\ge 0 \end{split} \hspace{11pt}$
(36) $ \begin{align} \varsigma k_3T_0(l)-\frac{3\lambda \phi(l)\psi(l)l}{2}\ge 0 \end{align} \hspace{86pt}$
(37) $ \begin{split} \omega_1 =& \min\{ {\varsigma}c-{\varsigma}{\sigma_1}-\frac{l\lambda c}{\sigma_2}+ \\& \frac{\lambda \rho}{2}(\phi(z)+z\phi'(z))\}>0 \end{split} \hspace{78pt}$
(38) $ \begin{aligned} \omega_2 = &\min\bigg\{\frac{\lambda}{2}(\phi(z)T_0(z)+z\phi'(z)T_0(z)-\\& z\phi(z)T_0'(z))-\lambda\sigma_2cl\phi^2(z)-\lambda\sigma_3l\phi^2(z) \bigg\}>0 \end{aligned} $
(39) $ \begin{split} \omega_3 = &\min\{3\phi(z)\psi'(z)+3z\phi'(z)\psi(z)-\\ &z\phi(z)\psi'(z)\} >0 \end{split} \hspace{32pt}$
(40) $ \begin{align} \omega_4 = \min\{\phi(z)+z\phi'(z)\} >0 \end{align}\hspace{67pt} $
(41) $ \begin{split} \omega_5 =\;& k_1+\frac{ T_0(l)}{2}-\frac{1}{\sigma_4}-\frac{| k_3T_0(l)-2 \psi(l)|\sigma_9}{2}-\\ &\frac{|T_0(l)k_2-EI|}{2\sigma_5}-\frac{|T_0(l)-d_a|}{2\sigma_6}>0 \end{split} $
(42) $ \begin{align} \omega_6 = k_4-\frac{ T_0(l)}{2} >0 \end{align} \hspace{105pt}$
(43) $ \begin{align} \alpha = \left(\frac{\varsigma}{\sigma_1}+\frac{l\lambda}{\sigma_3}\right)lF^2+{\varsigma}{\sigma_4}\varpi^2<+\infty \end{align} \hspace{33pt}$
(44) 结合式(33) ~(44), 可得
$ \begin{aligned} \dot{Y}(t) \le & \ \alpha-\omega_1\int^l_0\dot{y}^2(z, t){\rm{d}}z-\omega_2\int^l_0{y}^{{\prime}2}(z, t){\rm{d}}z-\\ & \frac{\lambda }{2}\omega_3\int^l_0{y}^{{\prime}4}(z, t){\rm{d}}z-\frac{3\lambda EI}{2}\omega_4\int^l_0{y}^{{\prime\prime}2}(z, t){\rm{d}}z-\\& \ \varsigma\omega_5\nu^2(t)-\varsigma\omega_6\mu^2(t)\le\\ & \ \delta_3[Y_e(t)+Y_f(t)]+\alpha \end{aligned} $
(45) 其中, $ \delta_3 = {\min}\left(\dfrac{2\omega_1}{{\varsigma}\rho}, \dfrac{2\omega_2}{{\varsigma}\overline{T}_0}, \dfrac{\lambda\omega_3}{\varsigma\overline{\psi}}, \dfrac{3\lambda\pi_4}{\varsigma}, \dfrac{2\pi_5}{m}, \dfrac{2\pi_6}{m}\right) $.
根据式(26)和式(45), 有
$ \begin{align} \dot{Y}(t)\le -\delta{Y}(t)+\alpha \end{align} $
(46) 其中, $ \delta = \delta_3/\delta_2 $.
定理1.针对执行器非光滑反向间隙−饱和约束特性的深海柔性立管系统, 如果系统初始条件是有界的且所选取参数满足约束条件式(33) ~(44), 在设计控制器(14)、假设1和假设2作用下, 闭环系统是一致有界稳定的.
证明.将式(27)乘以$ {\rm{e}}^{{\vartheta}t} $, 得出:
$ \begin{align} \frac{\partial}{\partial t}\left({Y}(t){\rm{e}}^{\delta t}\right)\le \alpha {\rm{e}}^{\delta t} \end{align} $
(47) 积分上式并变换, 有:
$ \begin{align} {Y}(t)\le Y(0){\rm{e}}^{-\delta t}+\frac{\alpha}{\delta}\left(1-{\rm{e}}^{-\delta t}\right)\le Y(0){\rm{e}}^{-\delta t}+\frac{\alpha}{\delta} \end{align} $
(48) 求助于$ Y_{e}(t) $, 式(19)和引理2, 可得
$ \begin{split} \frac{{\varsigma}\underline{T}_0}{2l}y^2(z,t)\le &\frac{{\varsigma}}{2}\int^l_0T_0(z){y}^{{\prime}2}(z,t){\rm{d}}z\le\\ &{Y_e(t)}\le\frac{1}{\delta_1}Y(t) \end{split} $
(49) 将式(48)代入式(49), 产生
$ \begin{split} \mid y(z,t)\mid \le \sqrt{\frac{2l}{{\varsigma}\delta_1\underline{T}_0}\left[Y(0){\rm{e}}^{-\delta t} +\frac{\alpha}{\delta}\right]}, \\ \forall (z,t) \in[0,l]\times[0,+\infty) \end{split} $
(50) 进一步得出
$ \begin{split} \underset{t\to\infty}{\mathop{\lim }} \,\left| y(z,t) \right| \le\sqrt{\frac{2l\alpha}{\varsigma{\underline{T}_0}{\delta}_{1}\delta}}, \ \ \ \forall z\in[0,l] \end{split} $
(51) 3. 数值仿真
为验证所设计控制器的性能, 本节在MATLAB软件中采用有限差分法[27-30]来近似闭环系统的数值解.柔性立管系统的参数为$ l = 1\; 000\, \rm{m}, $ $ \rho = 500\, \rm{kg/m}, $ $ c = 1.0\, \rm{Ns/m^2}, $ $ T_0(z) = 4.5\times10^5\times(1\; 000+z)\, \rm{N}, $ $\psi(z)=$ $ 1\times10^3 (1\; 000+z), $ $ EI $ = $ 1.5\times10^7\, {\rm N m^2}, ~{m}$ $=9.6\times10^6\, \rm{kg}, $ $ d_a = 1\; 000\, \rm{Ns/m}. $系统的初始条件描述为: $ y(z, 0) =$ $ \dfrac{12z}{l}, ~ \dot{y}(z, 0) = 0 $.
外部环境扰动$ d(t) $为
$ \begin{split} d(t) =\;& [3+0.8\sin(0.7t)+0.8\sin(0.5t)+\\& 0.8\sin(0.9t)]\times10^5 \end{split} $
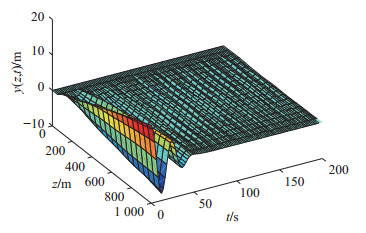
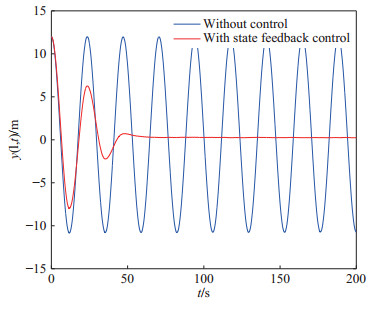
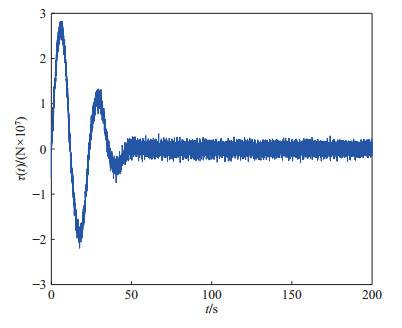
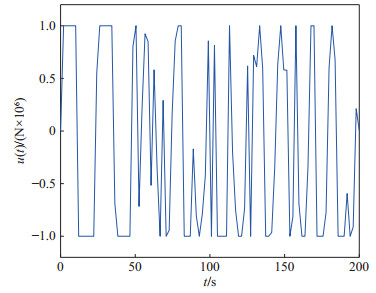
(52) 柔性立管系统在自由振动时, 即$ u(t) = 0 $, 图 2给出了其时空的表示.在所示设计控制器(14)作用下, 选取控制设计参数$ k_1 $ = $ 1\times10^7 $, $ k_2 = {1}/{60}, $ $ k_3= {1}/{225}, $ $ k_4 $ = $ 5\times10^8, $ $ a $ = $ 1\times10^6 $, $ b = 5~\times $ $10^6 $, 立管三维响应显示在图 3中. 图 4则给出了立管中部顶端$ (x = 1\; 000\; {\rm{m}}) $的二维偏移图, 图 5和图 6分别描绘了所设计的控制命令和反向间隙−饱和控制输入.
仿真图 2和图 3表明, 在外部扰动和执行器非光滑反向间隙−饱和约束条件下, 所设计控制器(14)能有效抑制立管振动; 由仿真图 4可得, 立管端点的偏移量稳定在平衡位置附近的小邻域; 仿真图 5和图 6得出, 控制器的输入是非线性的, 执行器非光滑反向间隙−饱和约束特性也相当地明显.根据上述分析, 可得如下结论:由于混合的输入非线性影响, 立管的振动偏移量需要相对长的收敛时间; 本文所构建的控制策略能较好地处理执行器非光滑反向间隙−饱和约束并能有效地抑制立管振动.
4. 结论
本文解决了具有执行器非光滑反向间隙−饱和约束特性的深海柔性立管边界控制问题.首先, 基于Lyapunov理论和边界控制技术, 采用辅助系统和函数在立管顶端构建了边界控制器以实现立管系统的振动抑制和输入非线性的补偿.其后, 应用严格的分析且没有离散化或简化系统的偏微分方程动力学, 证明了受控系统的一致有界性.最后所呈现的仿真结果验证了提出控制能较好地稳定立管系统并有效消除执行器非光滑反向间隙−饱和约束影响.下一步值得探索的研究方向可以为海洋柔性立管系统的有限时间稳定[31]以及基于不确定性和干扰估计[32]的控制设计.
-
表 1 人脑效应连接网络的部分典型识别方法对比
Table 1 The comparisons of several typical identification methods on human brain effective connectivity networks
方法 年份 类别 个体水平/组水平 探索型/验证型 主要优缺点及适用场景 uSEM[55] 2007 基于结构方程模型 个体水平 验证型 适用于组块设计实验, 能够识别同期和滞后的因果效应, 但不能用于事件相关的实验设计 euSEM[56] 2011 基于结构方程模型 个体水平、组水平 验证型、探索型 适用于事件相关设计实验, 不仅识别了同期和滞后的因果效应, 而且描述了外界刺激对脑区神经活动的直接影响和间接调节作用, 在个体水平和组水平上都获得了较好的识别效果 GIMME[58] 2012 基于结构方程模型 个体水平、组水平 验证型、探索型 从高度异质数据中准确地识别了组水平和个体水平的效应连接网络, 且具有良好的灵活性和可扩展性 cs-GIMME[59] 2019 基于结构方程模型 个体水平、组水平 验证型、探索型 与 GIMME 相比, 具有更强地识别组间差异的能力, 但不适于识别大规模脑效应连接网络 spDCM[62] 2014 基于动态因果模型 组水平 验证型 用于从静息态 fMRI 数据中识别高精度的效应连接网络, 对组间差异敏感, 不足在于仅考虑了较小规模的脑区, 且忽略了效应连接的动态性 rDCM[67] 2017 基于动态因果模型 个体水平 验证型 适用于识别较大规模的脑效应连接网络 spare rDCM[68] 2018 基于动态因果模型 个体水平、组水平 验证型 用于准确、高效地识别全脑的效应连接网络, 且对组间差异敏感 文献 [46] 2018 基于动态因果模型 个体水平、组水平 验证型 用于识别动态效应连接网络, 方法具有良好的可扩展性 文献 [71] 2016 基于 OUM 组水平 验证型 适用于识别较大规模的效应连接网络, 但滞后信息可能对因果效应的识别造成混淆 文献 [72] 2018 基于 OUM 组水平 验证型 利用零时滞的功能连接识别效应连接网络, 避免了滞后信息的不良影响, 但方法的鲁棒性较差 AIAEC[44] 2016 基于静态 BN 评分搜索 组水平 探索型 能够准确地识别脑区间的连接方向, 且在大规模脑区上也具有良好的性能, 缺陷是不能计算连接强度 ACOEC[45] 2016 基于静态 BN 评分搜索 组水平 探索型 能够识别连接方向并计算连接强度, 且具有良好的精度和鲁棒性, 但方法的时间复杂度较高 ACOMMEC[79] 2019 基于静态 BN 评分搜索 组水平 探索型 利用多源信息识别脑效应连接网络, 比仅利用 fMRI 数据获得了更高的求解质量和计算效率, 但不能模拟人脑的循环机制 GDBN[80] 2014 基于动态 BN 评分搜索 组水平 探索型 能够识别具有反馈机制的效应连接网络, 但该方法仅采用了一阶马尔科夫链 DBN 模型, 未考虑多个不同时刻的循环交互作用 HO-DBN-DP[81] 2017 基于动态 BN 评分搜索 组水平 探索型 能够高效地识别多个不同时刻的脑区间具有循环和反馈机制的因果效应连接 TB-based score[82] 2018 基于动态 BN 评分搜索 组水平 探索型 基于融合 fMRI 和 DTI 信息的评分函数学习网络结构, 在精度和鲁棒性上均获得了良好性能, 但不能有效地处理数据缺失问题 SVAR[92] 2015 基于格兰杰因果 组水平 探索型 利用因子降维策略获得了较高的计算效率, 适用于识别较大规模的效应连接网络, 不足在于因子模型对一些特殊脑区不适用 文献 [95] 2016 基于格兰杰因果 组水平 探索型 结合了时、频域格兰杰因果的优势, 获得了更高的准确性, 但方法对模型的阶数敏感 MKGC[90] 2017 基于格兰杰因果 组水平 探索型 适用于捕捉脑区间非线性的因果效应, 具有较强的识别出脑区之间连接的能力 lsGC[97] 2019 基于格兰杰因果 组水平 探索型 能高效地识别大规模脑区间的效应连接网络(在90、138 和 246 个节点的大规模脑区上验证了有效性) P-correlation[100] 2017 基于预测相关性 组水平 探索型 在数据非平稳的情况下能够准确地识别每对脑区间的因果效应, 具有灵敏度高、鲁棒性强的特点 GS[101] 2002 基于相空间重构 组水平 探索型 适用于识别每对脑区间的连接方向, 但有时三个指标的方向判断结果不一致 MCA[102] 2018 基于相空间重构 组水平 探索型 用于识别每对脑区间非线性的因果效应, 且在样本量较小时仍具有较高的准确率 文献 [105] 2019 基于概率分布 组水平 探索型 综合考虑了 BOLD 信号多方面的信息, 在每对脑区间方向的识别上具有良好的鲁棒性和准确性 CDD[106] 2019 基于概率分布 组水平 探索型 无需严格假设数据的分布, 能够灵活、准确地度量每对脑区间的非线性因果关系 -
[1] Lehrer J. Neuroscience: Making connections. Nature, 2009, 457(7229): 524−527 doi: 10.1038/457524a [2] 梁夏, 王金辉, 贺永. 人脑连接组研究: 脑结构网络和脑功能网络. 科学通报, 2010, 55(16): 1565−1583 doi: 10.1360/972009-2150Liang Xia, Wang Jin-Hui, He Yong. Human connectome: Structural and functional brain networks. Chinese Science Bulletin, 2010, 55(16): 1565−1583 doi: 10.1360/972009-2150 [3] Friston K J. Functional and effective connectivity: A review. Brain Connectivity, 2011, 1(1): 13−36 doi: 10.1089/brain.2011.0008 [4] Song M, Jiang T Z. A review of functional magnetic resonance imaging for Brainnetome. Neuroscience Bulletin, 2012, 28(4): 389−398 doi: 10.1007/s12264-012-1244-4 [5] Deshpande G, Li Z H, Santhanam P, Coles C D, Lynch M E, Hamann S, et al. Recursive cluster elimination based support vector machine for disease state prediction using resting state functional and effective brain connectivity. PLoS One, 2010, 5(12): e14277 doi: 10.1371/journal.pone.0014277 [6] Li X F, Coyle D, Maguire L, Watson D R, McGinnity T M. Gray matter concentration and effective connectivity changes in Alzheimer' s disease: A longitudinal structural MRI study. Neuroradiology, 2011, 53(10): 733−748 doi: 10.1007/s00234-010-0795-1 [7] Friston K J. Functional and effective connectivity in neuroimaging: A synthesis. Human Brain Mapping, 1994, 2(1−2): 56−78 doi: 10.1002/hbm.460020107 [8] Smith S M, Miller K L, Salimi-Khorshidi G, Webster M, Beckmann C F, Nichols T E, et al. Network modelling methods for fMRI. NeuroImage, 2011, 54(2): 875−891 doi: 10.1016/j.neuroimage.2010.08.063 [9] Farahani F V, Karwowski W, Lighthall N R. Application of graph theory for identifying connectivity patterns in human brain networks: A systematic review. Frontiers in Neuroscience, 2019, 13: 585 doi: 10.3389/fnins.2019.00585 [10] 左西年, 张喆, 贺永, 臧玉峰. 人脑功能连接组: 方法学、发展轨线和行为关联. 科学通报, 2012, 57(35): 3399−3413 doi: 10.1360/972012-702Zuo Xi-Nian, Zhang Zhe, He Yong, Zang Yu-Feng. The human functional connectome: Its methodology, developmental trajectory and behavioral association. Chinese Science Bulletin, 2012, 57(35): 3399−3413 doi: 10.1360/972012-702 [11] Biswal B B, Mennes M, Zuo X N, Gohel S, Kelly C, Smith S M, et al. Toward discovery science of human brain function. Proceedings of the National Academy of Sciences of the United States of America, 2010, 107(10): 4734−4739 doi: 10.1073/pnas.0911855107 [12] Friston K J. Modalities, modes, and models in functional neuroimaging. Science, 2009, 326(5951): 399−403 doi: 10.1126/science.1174521 [13] Turk-Browne N B. Functional interactions as big data in the human brain. Science, 2013, 342(6158): 580−584 doi: 10.1126/science.1238409 [14] Park H J, Friston K. Structural and functional brain networks: From connections to cognition. Science, 2013, 342(6158): 1238411 doi: 10.1126/science.1238411 [15] Hein G, Morishima Y, Leiberg S, Sul S, Fehr E. The brain' s functional network architecture reveals human motives. Science, 2016, 351(6277): 1074−1078 doi: 10.1126/science.aac7992 [16] Tavor I, Jones O P, Mars R B, Smith S M, Behrens T E, Jbabdi S. Task-free MRI predicts individual differences in brain activity during task performance. Science, 2016, 352(6282): 216−220 doi: 10.1126/science.aad8127 [17] Field G D, Gauthier J L, Sher A, Greschner M, Machado T A, Jepson L H, et al. Functional connectivity in the retina at the resolution of photoreceptors. Nature, 2010, 467(7316): 673−677 doi: 10.1038/nature09424 [18] Honey C J, Sporns O, Cammoun L, Gigandet X, Thiran J P, Meuli R, et al. Predicting human resting-state functional connectivity from structural connectivity. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(6): 2035−2040 doi: 10.1073/pnas.0811168106 [19] Smith S M, Fox P T, Miller K L, Glahn D C, Fox M, Mackay C E, et al. Correspondence of the brain' s functional architecture during activation and rest. Proceedings of the National Academy of Sciences of the United States of America, 2009, 106(31): 13040−13045 doi: 10.1073/pnas.0905267106 [20] Riedl V, Utz L, Castrillon G, Grimmer T, Rauschecker J P, Ploner M, et al. Metabolic connectivity mapping reveals effective connectivity in the resting human brain. Proceedings of the National Academy of Sciences of the United States of America, 2016, 113(2): 428−433 doi: 10.1073/pnas.1513752113 [21] Hermundstad A M, Bassett D S, Brown K S, Aminoff E M, Clewett D, Freeman S, et al. Structural foundations of resting-state and task-based functional connectivity in the human brain. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(15): 6169−6174 doi: 10.1073/pnas.1219562110 [22] Buijink A W G, Van Der Stouwe A M M, Broersma M, Sharifi S, Groot P F C, Speelman J D, et al. Motor network disruption in essential tremor: A functional and effective connectivity study. Brain, 2015, 138(10): 2934−2947 doi: 10.1093/brain/awv225 [23] Gibson W S, Jo H J, Testini P, Cho S, Felmlee J P, Welker K M, et al. Functional correlates of the therapeutic and adverse effects evoked by thalamic stimulation for essential tremor. Brain, 2016, 139(8): 2198−2210 doi: 10.1093/brain/aww145 [24] Huang S, Li J, Ye J P, Fleisher A, Chen K W, Wu T, et al. A sparse structure learning algorithm for Gaussian Bayesian network identification from high-dimensional data. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2013, 35(6): 1328−1342 doi: 10.1109/TPAMI.2012.129 [25] Zhou L P, Wang L, Liu L Q, Ogunbona P, Shen D G. Discriminative brain effective connectivity analysis for Alzheimer' s disease: A kernel learning approach upon sparse Gaussian Bayesian network. In: Proceedings of the 2013 IEEE Conference on Computer Vision and Pattern Recognition. Portland, USA: IEEE, 2013. 2243−2250 [26] Zhou L P, Wang L, Liu L Q, Ogunbona P, Shen D G. Learning discriminative Bayesian networks from high-dimensional continuous neuroimaging data. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2016, 38(11): 2269−2283 doi: 10.1109/TPAMI.2015.2511754 [27] Huang S, Li J, Ye J P, Fleisher A, Chen K W, Wu T, et al. Brain effective connectivity modeling for Alzheimer’s disease by sparse Gaussian Bayesian network. In: Proceedings of the 17th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. San Diego, USA: ACM, 2011. 931−939 [28] Logothetis N K, Pauls J, Augath M, Trinath T, Oeltermann A. Neurophysiological investigation of the basis of the fMRI signal. Nature, 2001, 412(6843): 150−157 doi: 10.1038/35084005 [29] 宫殿坤. 人脑认知加工的时空连续性与可塑性研究[博士学位论文], 电子科技大学, 中国, 2015Gong Dian-Kun. Research on the temporal-spatial continuity and plasticity of cognitive processing of human brain [Ph. D. dissertation], University of Electronic Science and Technology of China, China, 2015 [30] Cox R W, Hyde J S. Software tools for analysis and visualization of fMRI data. NMR in Biomedicine, 1997, 10(4-5): 171−178 doi: 10.1002/(SICI)1099-1492(199706/08)10:4/5<171::AID-NBM453>3.0.CO;2-L [31] Avants B B, Tustison N J, Song G, Cook P A, Klein A, Gee J C. A reproducible evaluation of ANTs similarity metric performance in brain image registration. NeuroImage, 2011, 54(3): 2033−2044 doi: 10.1016/j.neuroimage.2010.09.025 [32] Jenkinson M, Beckmann C F, Behrens T E J, Woolrich M W, Smith S M. FSL. NeuroImage, 2012, 62(2): 782−790 doi: 10.1016/j.neuroimage.2011.09.015 [33] Friston K J, Ashburner J T, Kiebel S J, Nichols T E, Penny W D. Statistical Parametric Mapping: The Analysis of Functional Brain Images. Amsterdam: Academic Press, 2007. [34] Song X W, Dong Z Y, Long X Y, Li S F, Zuo X N, Zhu C Z, et al. REST: A toolkit for resting-state functional magnetic resonance imaging data processing. PLoS One, 2011, 6(9): e25031 doi: 10.1371/journal.pone.0025031 [35] Yan C G, Wang X D, Zuo X N, Zang Y F. DPABI: Data processing & analysis for (resting-state) brain imaging. Neuroinformatics, 2016, 14(3): 339−351 doi: 10.1007/s12021-016-9299-4 [36] Esteban O, Markiewicz C J, Blair R W, Moodie C A, Isik A I, Erramuzpe A, et al. fMRIPrep: A robust preprocessing pipeline for functional MRI. Nature Methods, 2019, 16(1): 111−116 doi: 10.1038/s41592-018-0235-4 [37] Gao Q, Duan X J, Chen H F. Evaluation of effective connectivity of motor areas during motor imagery and execution using conditional Granger causality. NeuroImage, 2011, 54(2): 1280−1288 doi: 10.1016/j.neuroimage.2010.08.071 [38] Zuo X N, Kelly C, Adelstein J S, Klein D F, Castellanos F X, Milham M P. Reliable intrinsic connectivity networks: Test-retest evaluation using ICA and dual regression approach. NeuroImage, 2010, 49(3): 2163−2177 doi: 10.1016/j.neuroimage.2009.10.080 [39] Liu C H, JaJa J, Pessoa L. LEICA: Laplacian eigenmaps for group ICA decomposition of fMRI data. NeuroImage, 2018, 169: 363−373 doi: 10.1016/j.neuroimage.2017.12.018 [40] Tzourio-Mazoyer N, Landeau B, Papathanassiou D, Crivello F, Etard O, Delcroix N, et al. Automated anatomical labeling of activations in SPM using a macroscopic anatomical parcellation of the MNI MRI single-subject brain. NeuroImage, 2002, 15(1): 273−289 doi: 10.1006/nimg.2001.0978 [41] Shattuck D W, Mirza M, Adisetiyo V, Hojatkashani C, Salamon G, Narr K L, et al. Construction of a 3D probabilistic atlas of human cortical structures. NeuroImage, 2008, 39(3): 1064−1080 doi: 10.1016/j.neuroimage.2007.09.031 [42] Fan L Z, Li H, Zhuo J J, Zhang Y, Wang J J, Chen L F, et al. The human brainnetome atlas: A new brain atlas based on connectional architecture. Cerebral Cortex, 2016, 26(8): 3508−3526 doi: 10.1093/cercor/bhw157 [43] Glasser M F, Coalson T S, Robinson E C, Hacker C D, Harwell J, Yacoub E, et al. A multi-modal parcellation of human cerebral cortex. Nature, 2016, 536(7615): 171−178 doi: 10.1038/nature18933 [44] Ji J Z, Liu J D, Liang P P, Zhang A D. Learning effective connectivity network structure from fMRI data based on artificial immune algorithm. PLoS One, 2016, 11(4): e0152600 doi: 10.1371/journal.pone.0152600 [45] Liu J D, Ji J Z, Zhang A D, Liang P P. An ant colony optimization algorithm for learning brain effective connectivity network from fMRI data. In: Proceedings of the 2016 IEEE International Conference on Bioinformatics and Biomedicine. Shenzhen, China: IEEE, 2016. 360−367 [46] Park H J, Friston K J, Pae C, Park B, Razi A. Dynamic effective connectivity in resting state fMRI. NeuroImage, 2018, 180: 594−608 doi: 10.1016/j.neuroimage.2017.11.033 [47] Zhou Z Y, Chen Y H, Ding M Z, Wright P, Lu Z H, Liu Y J. Analyzing brain networks with PCA and conditional Granger causality. Human Brain Mapping, 2009, 30(7): 2197−2206 doi: 10.1002/hbm.20661 [48] Sanchez-Romero R, Ramsey J D, Zhang K, Glymour M R K, Huang B W, Glymour C. Estimating feedforward and feedback effective connections from fMRI time series: Assessments of statistical methods. Network Neuroscience, 2019, 3(2): 274−306 doi: 10.1162/netn_a_00061 [49] McLntosh A R, Gonzalez-Lima F. Structural equation modeling and its application to network analysis in functional brain imaging. Human Brain Mapping, 1994, 2(1−2): 2−22 doi: 10.1002/hbm.460020104 [50] Spirtes P, Glymour C, Scheines R. Causation, Prediction, and Search (Second edition). Cambridge: MIT Press, 2000. [51] Ramnani N, Behrens T E J, Penny W, Matthews P M. New approaches for exploring anatomical and functional connectivity in the human brain. Biological Psychiatry, 2004, 56(9): 613−619 doi: 10.1016/j.biopsych.2004.02.004 [52] Penny W D, Stephan K E, Mechelli A, Friston K J. Modelling functional integration: A comparison of structural equation and dynamic causal models. NeuroImage, 2004, 23(Suppl 1): S264−S274 [53] Shimizu S, Hoyer P O, Hyvarinen A, Kerminen A. A linear non-Gaussian acyclic model for causal discovery. Journal of Machine Learning Research, 2006, 7: 2003−2030 [54] Shimizu S, Inazumi T, Sogawa Y, Hyvarinen A, Kerminen A, Washio T, et al. DirectLiNGAM: A direct method for learning a linear non-Gaussian structural equation model. Journal of Machine Learning Research, 2011, 12: 1225−1248 [55] Kim J, Zhu W, Chang L D, Bentler P M, Ernst T. Unified structural equation modeling approach for the analysis of multisubject, multivariate functional MRI data. Human Brain Mapping, 2007, 28(2): 85−93 doi: 10.1002/hbm.20259 [56] Gates K M, Molenaar P C M, Hillary F G, Slobounov S. Extended unified SEM approach for modeling event-related fMRI data. NeuroImage, 2011, 54(2): 1151−1158 doi: 10.1016/j.neuroimage.2010.08.051 [57] Hillary F G, Medaglia J D, Gates K, Molenaar P C, Slocomb J, Peechatka A, et al. Examining working memory task acquisition in a disrupted neural network. Brain, 2011, 134(5): 1555−1570 doi: 10.1093/brain/awr043 [58] Gates K M, Molenaar P C M. Group search algorithm recovers effective connectivity maps for individuals in homogeneous and heterogeneous samples. NeuroImage, 2012, 63(1): 310−319 doi: 10.1016/j.neuroimage.2012.06.026 [59] Henry T R, Feczko E, Cordova M, Earl E, Williams S, Nigg J T, et al. Comparing directed functional connectivity between groups with confirmatory subgrouping GIMME. NeuroImage, 2019, 188: 642−653 doi: 10.1016/j.neuroimage.2018.12.040 [60] 姚垚, 冀俊忠. 基于栈式循环神经网络的血液动力学状态估计方法. 自动化学报, 2020, 46(5): 991−1003Yao Yao, Ji Jun-Zhong. Estimation of hemodynamic states based on stacked recurrent neural network in fMRI time series. Acta Automatica Sinica, 2020, 46(5): 991−1003 [61] Li B J, Daunizeau J, Stephan K E, Penny W, Hu D W, Friston K. Generalised filtering and stochastic DCM for fMRI. NeuroImage, 2011, 58(2): 442−457 doi: 10.1016/j.neuroimage.2011.01.085 [62] Friston K J, Kahan J, Biswal B, Razi A. A DCM for resting state fMRI. NeuroImage, 2014, 94: 396−407 doi: 10.1016/j.neuroimage.2013.12.009 [63] Razi A, Kahan J, Rees G, Friston K J. Construct validation of a DCM for resting state fMRI. NeuroImage, 2015, 106: 1−14 doi: 10.1016/j.neuroimage.2014.11.027 [64] Daunizeau J, David O, Stephan K E. Dynamic causal modelling: A critical review of the biophysical and statistical foundations. NeuroImage, 2011, 58(2): 312−322 doi: 10.1016/j.neuroimage.2009.11.062 [65] Seghier M L, Friston K J. Network discovery with large DCMs. NeuroImage, 2013, 68: 181−191 doi: 10.1016/j.neuroimage.2012.12.005 [66] Razi A, Seghier M L, Zhou Y, McColgan P, Zeidman P, Park H J, et al. Large-scale DCMs for resting-state fMRI. Network Neuroscience, 2017, 1(3): 222−241 doi: 10.1162/NETN_a_00015 [67] Frassle S, Lomakina E I, Razi A, Friston K J, Buhmann J M, Stephan K E. Regression DCM for fMRI. NeuroImage, 2017, 155: 406−421 doi: 10.1016/j.neuroimage.2017.02.090 [68] Frassle S, Lomakina E I, Kasper L, Manjaly Z M, Leff A, Pruessmann K P, et al. A generative model of whole-brain effective connectivity. NeuroImage, 2018, 179: 505−529 doi: 10.1016/j.neuroimage.2018.05.058 [69] Friston K, Zeidman P, Litvak V. Empirical Bayes for DCM: A group inversion scheme. Frontiers in Systems Neuroscience, 2015, 9: 164 [70] Friston K J, Litvak V, Oswal A, Razi A, Stephan K E, Van Wijk B C M, et al. Bayesian model reduction and empirical Bayes for group (DCM) studies. NeuroImage, 2016, 128: 413−431 doi: 10.1016/j.neuroimage.2015.11.015 [71] Gilson M, Moreno-Bote R, Ponce-Alvarez A, Ritter P, Deco G. Estimation of directed effective connectivity from fMRI functional connectivity hints at asymmetries of cortical connectome. PLoS Computational Biology, 2016, 12(3): e1004762 doi: 10.1371/journal.pcbi.1004762 [72] Gilson M, Deco G, Friston K J, Hagmann P, Mantini D, Betti V, et al. Effective connectivity inferred from fMRI transition dynamics during movie viewing points to a balanced reconfiguration of cortical interactions. NeuroImage, 2018, 180: 534−546 doi: 10.1016/j.neuroimage.2017.09.061 [73] Schiefer J, Niederbuhl A, Pernice V, Lennartz C, Hennig J, LeVan P, et al. From correlation to causation: Estimating effective connectivity from zero-lag covariances of brain signals. PLoS Computational Biology, 2018, 14(3): e1006056 doi: 10.1371/journal.pcbi.1006056 [74] Ramsey J D, Hanson S J, Hanson C, Halchenko Y O, Poldrack R A, Glymour C. Six problems for causal inference from fMRI. NeuroImage, 2010, 49(2): 1545−1558 doi: 10.1016/j.neuroimage.2009.08.065 [75] Li R, Chen K W, Zhang N, Fleisher A S, Yao L, Wu X. Effective connectivity analysis of default mode network based on the Bayesian network learning approach. In: Proceedings of the Medical Imaging 2009: Biomedical Applications in Molecular, Structural, and Functional Imaging. Lake Buena Vista (Orlando Area), USA: SPIE, 2009. Article No. 72621W [76] Zheng X B, Rajapakse J C. Learning functional structure from fMR images. NeuroImage, 2006, 31(4): 1601−1613 doi: 10.1016/j.neuroimage.2006.01.031 [77] Rajapakse J C, Zhou J. Learning effective brain connectivity with dynamic Bayesian networks. NeuroImage, 2007, 37(3): 749−760 doi: 10.1016/j.neuroimage.2007.06.003 [78] Burge J, Lane T, Link H, Qiu S B, Clark V P. Discrete dynamic Bayesian network analysis of fMRI data. Human Brain Mapping, 2009, 30(1): 122−137 doi: 10.1002/hbm.20490 [79] 冀俊忠, 刘金铎, 邹爱笑, 杨翠翠. 一种融合多源信息的脑效应连接网络蚁群学习算法. 自动化学报, DOI: 10.16383/j.aas.c180680. 2019Ji Jun-Zhong, Liu Jin-Duo, Zou Ai-Xiao, Yang Cui-Cui. An ant colony optimization algorithm merged with multiple source information for learning brain effective connectivity networks. Acta Automatica Sinica, DOI: 10.16383/j.aas.c180680. 2019. [80] Wu X, Wen X Y, Li J, Yao L. A new dynamic Bayesian network approach for determining effective connectivity from fMRI data. Neural Computing and Applications, 2014, 24(1): 91−97 doi: 10.1007/s00521-013-1465-0 [81] Dang S, Chaudhury S, Lall B, Roy P K. The dynamic programming high-order dynamic Bayesian networks learning for identifying effective connectivity in human brain from fMRI. Journal of Neuroscience Methods, 2017, 285: 33−44 doi: 10.1016/j.jneumeth.2017.05.009 [82] Dang S, Chaudhury S, Lall B, Roy P K. Tractography-based score for learning effective connectivity from multimodal imaging data using dynamic Bayesian networks. IEEE Transactions on Biomedical Engineering, 2018, 65(5): 1057−1068 [83] Harrison L, Penny W D, Friston K J. Multivariate autoregressive modeling of fMRI time series. NeuroImage, 2003, 19(4): 1477−1491 doi: 10.1016/S1053-8119(03)00160-5 [84] Valdes-Sosa P A, Roebroeck A, Daunizeau J, Friston K. Effective connectivity: Influence, causality and biophysical modeling. NeuroImage, 2011, 58(2): 339−361 doi: 10.1016/j.neuroimage.2011.03.058 [85] Liao W, Mantini D, Zhang Z Q, Pan Z Y, Ding J R, Gong Q Y, et al. Evaluating the effective connectivity of resting state networks using conditional Granger causality. Biological Cybernetics, 2010, 102(1): 57−69 doi: 10.1007/s00422-009-0350-5 [86] Guo S X, Seth A K, Kendrick K M, Zou C, Feng J F. Partial Granger causality—eliminating exogenous inputs and latent variables. Journal of Neuroscience Methods, 2008, 172(1): 79−93 doi: 10.1016/j.jneumeth.2008.04.011 [87] Baccala L A, Sameshima K. Partial directed coherence: A new concept in neural structure determination. Biological Cybernetics, 2001, 84(6): 463−474 doi: 10.1007/PL00007990 [88] Eichler M. On the Evaluation of Information flow in multivariate systems by the directed transfer function. Biological Cybernetics, 2006, 94(6): 469−482 doi: 10.1007/s00422-006-0062-z [89] Wu M H, Frye R E, Zouridakis G. A comparison of multivariate causality based measures of effective connectivity. Computers in Biology and Medicine, 2011, 41(12): 1132−1141 doi: 10.1016/j.compbiomed.2011.06.007 [90] Karanikolas G V, Giannakis G B. Identifying directional connections in brain networks via multi-kernel granger models. In: Proceedings of the 2017 IEEE International Conference on Acoustics, Speech and Signal Processing. New Orleans, USA: IEEE, 2017. 6304−6308 [91] Lobier M, Siebenhuhner F, Palva S, Palva J M. Phase transfer entropy: A novel phase-based measure for directed connectivity in networks coupled by oscillatory interactions. NeuroImage, 2014, 85: 853−872 doi: 10.1016/j.neuroimage.2013.08.056 [92] Ting C M, Seghouane A K, Salleh S H, Noor A M. Estimating effective connectivity from fMRI data using factor-based subspace autoregressive models. IEEE Signal Processing Letters, 2015, 22(6): 757−761 doi: 10.1109/LSP.2014.2365634 [93] Samdin S B, Ting C M, Ombao H, Salleh S H. A unified estimation framework for state-related changes in effective brain connectivity. IEEE Transactions on Biomedical Engineering, 2017, 64(4): 844−858 doi: 10.1109/TBME.2016.2580738 [94] Ting C M, Ombao H, Samdin S B, Salleh S H. Estimating dynamic connectivity states in fMRI using regime-switching factor models. IEEE Transactions on Medical Imaging, 2018, 37(4): 1011−1023 doi: 10.1109/TMI.2017.2780185 [95] Wei H L, An J, Shen H, Zeng L L, Qiu S J, Hu D W. Altered effective connectivity among core neurocognitive networks in idiopathic generalized epilepsy: An fMRI evidence. Frontiers in Human Neuroscience, 2016, 10: 447 [96] Meier J, Zhou X, Hillebrand A, Tewarie P, Stam C J, Van Mieghem P. The epidemic spreading model and the direction of information flow in brain networks. NeuroImage, 2017, 152: 639−646 doi: 10.1016/j.neuroimage.2017.02.007 [97] Chockanathan U, DSouza A M, Abidin A Z, Schifitto G, Wismuller A. Automated diagnosis of HIV-associated neurocognitive disorders using large-scale Granger causality analysis of resting-state functional MRI. Computers in Biology and Medicine, 2019, 106: 24−30 doi: 10.1016/j.compbiomed.2019.01.006 [98] Ambrogioni L, Hinne M, Van Gerven M A J, Maris E. GP CaKe: Effective brain connectivity with causal kernels. In: Proceedings of the 31st International Conference on Neural Information Processing Systems. Long Beach, USA: ACM, 2017. 951−960 [99] Farokhzadi M, Hossein-Zadeh G A, Soltanian-Zadeh H. Nonlinear effective connectivity measure based on adaptive Neuro Fuzzy Inference System and Granger Causality. NeuroImage, 2018, 181: 382−394 doi: 10.1016/j.neuroimage.2018.07.024 [100] Xu N, Spreng R N, Doerschuk P C. Initial validation for the estimation of resting-state fMRI effective connectivity by a generalization of the correlation approach. Frontiers in Neuroscience, 2017, 11: 271 doi: 10.3389/fnins.2017.00271 [101] Quian Quiroga R, Kraskov A, Kreuz T, Grassberger P. Performance of different synchronization measures in real data: A case study on electroencephalographic signals. Physical Review E, 2002, 65(4): 41903 doi: 10.1103/PhysRevE.65.041903 [102] Dsouza A M, Abidin A Z, Chockanathan U, Schifitto G, Wismuller A. Mutual connectivity analysis of resting-state functional MRI data with local models. NeuroImage, 2018, 178: 210−223 doi: 10.1016/j.neuroimage.2018.05.038 [103] Patel R S, Bowman D B, Rilling J K. A Bayesian approach to determining connectivity of the human brain. Human Brain Mapping, 2006, 27(3): 267−276 doi: 10.1002/hbm.20182 [104] Hyvärinen A, Smith S M. Pairwise likelihood ratios for estimation of non-Gaussian structural equation models. Journal of Machine Learning Research, 2013, 14(1): 111−152 [105] Bielczyk N Z, Llera A, Buitelaar J K, Glennon J C, Beckmann C F. Increasing robustness of pairwise methods for effective connectivity in Magnetic Resonance Imaging by using fractional moment series of BOLD signal distributions. Network Neuroscience, 2019, 3(4): 1009−1037 doi: 10.1162/netn_a_00099 [106] Lee N, Kim J M. Copula directional dependence for inference and statistical analysis of whole-brain connectivity from fMRI data. Brain and Behavior, 2019, 9(1): e01191 doi: 10.1002/brb3.1191 [107] Chiang S, Guindani M, Yeh H J, Haneef Z, Stern J M, Vannucci M. Bayesian vector autoregressive model for multi-subject effective connectivity inference using multi-modal neuroimaging data. Human Brain Mapping, 2017, 38(3): 1311−1332 doi: 10.1002/hbm.23456 [108] Anwar A R, Muthalib M, Perrey S, Galka A, Granert O, Wolff S, et al. Effective connectivity of cortical sensorimotor networks during finger movement tasks: A simultaneous fNIRS, fMRI, EEG study. Brain Topography, 2016, 29(5): 645−660 doi: 10.1007/s10548-016-0507-1 [109] 纪子龙, 冀俊忠, 刘金铎, 杨翠翠. 基于萤火虫算法的脑效应连接网络学习方法. 哈尔滨工业大学学报, 2019, 51(5): 76−84 doi: 10.11918/j.issn.0367-6234.201811073Ji Zi-Long, Ji Jun-Zhong, Liu Jin-Duo, Yang Cui-Cui. Learning effective connectivity network structure based on firefly algorithm. Journal of Harbin Institute of Technology, 2019, 51(5): 76−84 doi: 10.11918/j.issn.0367-6234.201811073 [110] 纪子龙, 冀俊忠. 基于双萤火虫种群并行搜索的脑效应连接网络学习方法. 浙江大学学报(工学版), 2020, 54(4): 694−703, 777Ji Zi-Long, Ji Jun-Zhong. Learning effective connectivity network structure based on parallel searching of double firefly populations. Journal of Zhejiang University (Engineering Science), 2020, 54(4): 694−703, 777 [111] Goudet O, Kalainathan D, Caillou P, Guyon I, Lopez-Paz D, Sebag M. Causal generative neural networks. arXiv: 1711.08936v2, 2018. [112] Kalainathan D, Goudet O, Guyon I, Lopez-Paz D, Sebag M. SAM: Structural agnostic model, causal discovery and penalized adversarial learning. arXiv: 1803.04929, 2018. [113] Njah H, Jamoussi S, Mahdi W. Deep Bayesian network architecture for Big Data mining. Concurrency and Computation Practice and Experience, 2019, 31(2): e4418 doi: 10.1002/cpe.4418 [114] Dasgupta I, Wang J, Chiappa S, Mitrovic J, Ortega P, Raposo D, et al. Causal reasoning from meta-reinforcement learning. arXiv: 1901.08162v1, 2019. [115] Faes L, Stramaglia S, Marinazzo D. On the interpretability and computational reliability of frequency-domain Granger causality. F1000Research, 2017, 6: 1710 doi: 10.12688/f1000research.12694.1 期刊类型引用(18)
1. 翟漪璇,宋丽梅,贺瑾胜,朱新军. 低重叠率人体点云拼接方法研究. 应用激光. 2024(03): 204-213 .  百度学术
百度学术2. 毕淳锴,张远辉,付铎. 基于多视角热像图序列的物体表面温度场重建. 计量学报. 2024(07): 997-1006 .  百度学术
百度学术3. 王耀南,谢核,邓晶丹,毛建旭,李文龙,张辉. 智能制造测量机器人关键技术研究综述. 机械工程学报. 2024(16): 1-18 .  百度学术
百度学术4. 梁循,李志莹,蒋洪迅. 基于图的点云研究综述. 计算机研究与发展. 2024(11): 2870-2896 .  百度学术
百度学术5. 冯站银. 三维点云语义分割方法综述. 电视技术. 2023(03): 140-143+148 .  百度学术
百度学术6. 李颀,郭梦媛. 基于深度学习的休眠期苹果树点云语义分割. 江苏农业学报. 2023(05): 1189-1198 .  百度学术
百度学术7. 黄淞宣,李新春,刘玉珍. 邻域多维度特征点结合相关熵的点云配准. 激光与红外. 2023(08): 1163-1170 .  百度学术
百度学术8. 单铉洋,孙战里,曾志刚. RFNet:用于三维点云分类的卷积神经网络. 自动化学报. 2023(11): 2350-2359 .  本站查看
本站查看9. 马洁莹,田暄,翟庆,王丞. 基于点到面度量的多视角点云配准方法. 西安交通大学学报. 2022(06): 120-132 .  百度学术
百度学术10. 杨宜林,李积英,王燕,俞永乾. 基于NDT和特征点检测的点云配准算法研究. 激光与光电子学进展. 2022(08): 198-204 .  百度学术
百度学术11. 鲁斌,范晓明. 基于改进自适应k均值聚类的三维点云骨架提取的研究. 自动化学报. 2022(08): 1994-2006 .  本站查看
本站查看12. 陈亚超,樊彦国,樊博文,禹定峰. 基于相对几何不变性的点云粗配准算法研究. 计算机工程与应用. 2022(24): 233-238 .  百度学术
百度学术13. 庄仁诚,陈鹏,傅瑶,黄运华. 列车车轮三维结构光检测中的点云处理研究. 中国测试. 2021(02): 19-25 .  百度学术
百度学术14. 沈小军,于忻乐,王远东,程林,王东升,陈佳. 变电站电力设备红外热像测温数据三维可视化方案. 高电压技术. 2021(02): 387-395 .  百度学术
百度学术15. 杨贵强,李瑞,刘玉君,汪骥,周玉松. 最大相关熵的船体分段扫描数据配准算法. 中国造船. 2021(01): 183-191 .  百度学术
百度学术16. 元沐南,李晓风,李皙茹,许金林. 基于压缩感知的三维足型重建平台. 电子测量技术. 2020(09): 94-98 .  百度学术
百度学术17. 林伟,孙殿柱,李延瑞,沈江华. 复杂型面约束的点云配准序列确定方法. 小型微型计算机系统. 2020(09): 2012-2016 .  百度学术
百度学术18. 黄思捷,梁正友,孙宇,李轩昂. 单Kinect+圆盒的多视角三维点云配准方法研究. 现代计算机. 2020(31): 38-45 .  百度学术
百度学术其他类型引用(17)
-





 下载:
下载:

 下载:
下载: