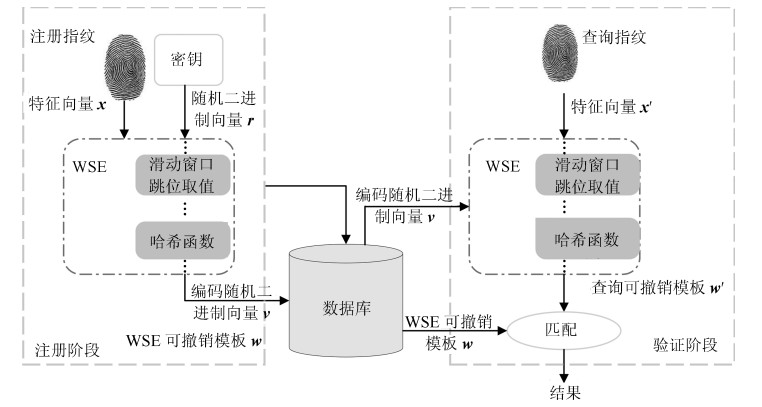
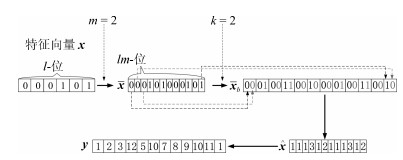
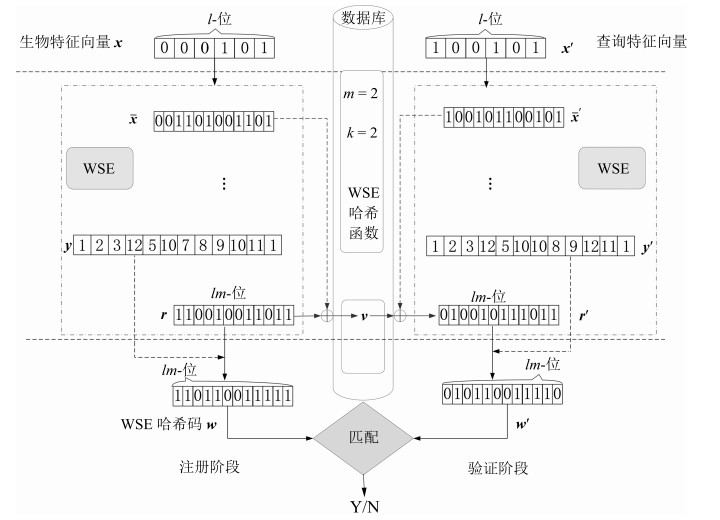
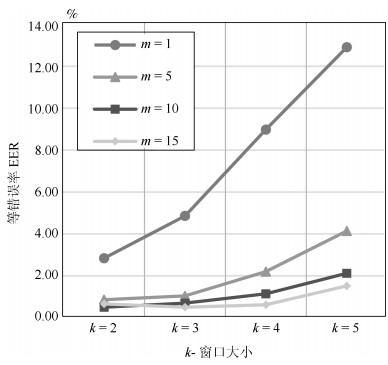
-
摘要: 将令牌化随机数作为外部因子的双因子可撤销生物特征认证方法存在令牌泄露、丢失等安全威胁. 本文提出了一种生物特征作为唯一输入的解决方法, 即单因子的可撤销生物特征认证方法. 首先, 利用扩展的特征向量, 通过预定义的滑动窗口和哈希函数随机化生成二进制种子; 然后替换不同的辅助数据来生成可撤销模板; 最后, 由查询生物特征向量对辅助数据进行解码, 提高了性能和安全性. 在指纹数据库FVC2002和FVC2004的实验结果表明, 该方法不仅满足可撤销生物特征识别的4个设计标准, 同时防御了3种安全攻击.Abstract: Two-factor cncellable biometrics use tokenized random number as an external factor, however, tokenized factor incurs severe security and privacy threats. In this paper, we propose a one-factor cancellable biometrics scheme, which requires sole biometric as input. First, it exploits an expanded feature vector, generating seeds of randomized binary auxiliary data by sliding a pre-defined window and Hash function, which enhances performance and security. Then, the cancellable template hence can be generated by replacing different binary auxiliary data. Finally, the auxiliary data is decoded by using sole query biometric. The experiments have been conducted on FVC2002 and FVC2004 databases, and results show that the scheme does not only fulfill four design criteria of cancellable biometrics but also resist to the threat model that enclosed three security attacks.
-
Key words:
- Biometrics /
- template protection /
- cancellable /
- one-factor /
- two-factor
-
近年来, 人工智能方法经过进一步的发展, 已经得到广泛的应用, 其中在机器人领域的应用也取得了大幅进展[1−3]. 目前的机器人已经具备了较高程度的智能水平 (多模态信息感知与识别能力[4]、复杂环境的最优决策与控制[5−7]等). 然而, 与人脑或者广义程度上的生物神经系统的智能相比, 机器人的智能水平还存在诸多不足[8], 例如缺乏自主性和高级推断能力, 无法针对新颖、复杂、未知的情况或者问题做出像人一样智能的思考与决策等. 针对这一问题, 学术界已经提出一些理论和方法, 其中的类脑智能方法近年来得到了广泛关注[9−10].
自Maass[11]在1997年提出基于脉冲神经元的神经网络之后, 以脉冲神经网络 (Spiking neural network, SNN)为代表的一系列类脑智能方法经过二十余年的发展, 已经展现出独特的优势和广阔的前景, 近年来得到了学术界的广泛关注. Roy等[12]在2019年发表于《自然》的一篇综述文章中指出, SNN利用神经元的时序信息进行信息处理, 与传统的基于实值计算的深度学习网络有本质区别, 前者可以充分利用时空事件数据, 实现稀疏和高效的计算. 类脑智能方法是受大脑或者生物神经运行机制和认知行为机制启发, 以计算建模为手段, 通过软硬件协同实现的机器智能, 具备信息处理机制上类脑、认知行为表现和智能水平上达到或超越人 (或其他仿生对象)的特点. 从机器人系统的角度, 类脑智能的主要应用可以分为软件和硬件两方面, 软件方面主要指机器人实现感知、控制、决策等功能所使用的算法为类脑方法, 这类方法借鉴了生物学上的神经系统的学习机制和表达能力, 能够对传统方法不善于解决的各种问题提出新的解决方案[6, 9−10]; 硬件层面主要指神经形态计算硬件的部署, 即将软件层面的类脑算法采用特殊的计算介质 (例如脉冲神经网络对应的类脑芯片)来实现, 具有计算高效、能耗较低、鲁棒性强的优势[11, 13]. 以上特点有望将目前的机器人系统的智能化程度和综合性能提升到新的高度, 因此具有很高的研究价值.
针对类脑智能方法在机器人系统上的应用, 国内外目前已有少数综述文章, 例如Bing等[14]在2018年的综述中总结了SNN常用的基本模型、学习规则、仿真平台和在机器人控制中的部分应用, 但整体偏向SNN基础理论, 且只关注机器人的控制问题. Qiao等[8]在2022年的综述中总结了基于视觉信息的类脑感知方法、基于情感的类脑决策方法和模仿神经机制的类脑控制方法及其在肌肉骨骼机器人 (Musculoskeletal systems)上的应用, 但对移动机器人和其他类型的仿生机器人关注较少. 2023年, 朱祥维等[15]综述了应用于机器人的导航任务中的类脑方法. 概括而言, 虽然目前关于类脑智能在机器人系统的应用已有不少先进的案例, 但缺少对该领域文献资料较为全面的总结归纳. 本文的主要目的是将目前国内外已有的面向各类机器人系统或者可应用于机器人系统的类脑智能方法研究按照机器人感知、决策和控制三个主要功能进行归纳整理, 对常用的技术路线和方法论进行总结提炼, 对未来可能的研究方向进行思考分析, 最终为后续开展的研究工作提供参考.
1. 类脑智能在感知方面的应用
感知能力是机器人实现与外部自主交互的必备功能, 在机器人系统中的重要性不言而喻. 本节将机器人的感知能力定义为机器人通过传感器获取外部多模态信息的能力, 这些模态主要包括图像信息 (视觉)、声音信息 (听觉)以及与物体接触或碰撞的信息 (触觉)等. 值得一提的是, 部分文献中提出的机器人系统虽然具备传感器和外部信息获取能力, 但这些部分是集成在运动控制系统内的, 本质上服务于某一特定场景或任务的机器人控制问题, 为避免发生混淆, 这些文献将在第2节和第3节中与其他基于类脑智能的决策与控制算法共同讨论, 本节中只涉及对纯感知方向文献的总结.
1)视觉方面. 与传统的卷积神经网络 (Convolutional neural network, CNN)等方法类似, 类脑智能的主要应用场景包括目标检测、图像分类、人脸识别、行为识别等, 但与以CNN为代表的第二代神经网络相比, SNN由于其自身特殊的模型性质 (神经元之间的交互基于脉冲信号, 存在不可导点), 无法直接计算输出对某一参数的梯度信息从而实现误差反向传播, 这一缺点曾经一度制约了SNN在视觉信息加工方面的应用. 替代梯度 (Surrogate gradient)等方法的出现一定程度上解决了这一问题, SNN在视觉上的应用前景也重新得到重视. 例如中国科学院自动化研究所的李国齐研究团队[16−18]针对难以直接训练大规模深度SNN的问题, 提出“阈值依赖批归一化(Spatio-temporal backpropagation (STBP)-threshold-dependent batch normalization (tdBN))”等一系列方法, 可以直接训练非常深的SNN, 并在神经形态硬件上进行有效部署, 同时在多个具有代表性的公共图像数据集上具有可靠的分类性能. 基于这一研究思路, Niu等[19]总结了常见的多层SNN以及SNN与卷积层相结合的混合神经网络在MNIST、NMNIST等代表性数据集上的分类性能, 从结果上看, 目前这一类基于梯度反传的SNN在部分图像处理任务上的性能已经与CNN相当. 具体到在机器人系统的应用中, Zhou等[20]提出一种基于脉冲卷积神经网络 (Spiking CNN, SCNN)的YOLOv2架构, 可以进行实时的物体检测, 该模型在基准数据集上的表现与现有方法相当 (该文认为部分情况下表现更好), 并且具有足以实现实时操作的处理帧率和不足0.25 mJ的总能耗. Li等[21]针对机器人与人交互过程中的手势识别问题, 提出一个基于脉冲双向联想记忆网络的认知机器人模型, 该文提出一种基于脉冲神经网络的手势识别方法, 利用运动轨迹作为手势特征. 实验结果表明, 该文所提出的认知机器人可以在不同场景下与人类进行交互, 并根据手势动作的视觉信息进行相应的回应. Hussaini等[22]针对移动机器人系统的视觉场景识别问题 (Visual place recognition, VPR)提出第一个高性能SNN模型, 并在多个公共数据集上进行测试, 结果表明其模型与目前其他文献中的最优模型具有相当的性能, 并且随着识别场景数量的增加, 模型的识别性能没有出现断崖式下降. 此外, 为了获取SNN在神经形态硬件上低功耗等优势, 同时继承目前CNN在建模和训练上的丰富经验, 也有部分学者提出将训练好的CNN转换为SNN的方法[23].
上述提到的SNN往往采用全连接等传统神经网络的连接形式, 没有选择从网络的拓扑特性上进一步“类脑”. 针对机器人的视觉感知任务, 也有部分研究从生物学上的神经网络特性出发, 提出相应的类脑视觉模型. 例如, Rast等[24]提出一个基于SNN模型作为“视觉注意力模型”并部署在类脑芯片和iCub机器人上. 如图1所示, 该网络由V1、V2等基础视觉特征提取层和FEF、LIP等特征信息加工层组成, 每一层的网络均来自于大脑中实际存在的脑区, 网络从机制上模拟了自下而上的信息传递和自上而下的注意力调节. Qiao等[25−26]针对人脸识别问题, 提出多个受到灵长类动物视觉皮层功能和结构启发的视觉模型, 旨在模拟灵长类动物视觉皮层对特定类别对象的主动、动态的学习和识别过程, 并在公共数据集上进行对比, 结果显示该模型具有训练高效的特点和相比于传统方法更好的性能.
除视觉外, 类脑智能在机器人的听觉、触觉以及空间位置与概念的感知上也有广阔的应用前景. 在听觉方面, 相比于目前主流的声音感知系统, 人耳在感知精度、动态适应性和全息感知能力等方面依然处于绝对的领先地位, 能够对广泛的声音频谱做出高度敏感和精确的响应, 并且根据环境的变化和不同声音源的特征进行动态适应, 实现对声音方向、距离、高度等信息的全息感知. 与之对应的是类脑听觉 (Neuromorphic auditory processing)方法, 该方法模仿人类听觉系统的工作原理, 通过生物学启发的方式实现机器听觉感知. Zou等[4]在其提出的类脑机器人平台中使用SNN来进行语音信号处理与语义理解, 使机器人具备了接收语音命令的能力 (图2); Deng等[27]使用SNN建模人类的听觉通路, 并使用FPGA (Field programmable gate array)实现了SNN模型对应的硬件电路. 该模型在含有噪音的语音分类任务 (10分类)下达到了79.88%的准确率 (该文认为在仿真环境下实现的更大规模的SNN对应的准确率可以达到85.2%), 并在后期的工作中 (见表1) 进一步提出基于FPGA的灵活实现方法, 在降低单芯片能耗并提升数据吞吐量的前提下, 使得整个系统可以根据目标应用场景进行定制化组装; Liu等[28]利用内耳毛细胞的相位锁定机制, 将声波的时间和幅度信息编码为神经脉冲, 提出一个基于SNN的哺乳动物听觉通路的计算模型, 实现了双耳声源定位. 实验结果显示, 该模型能够准确地估计声源的方位, 并且与神经生理数据有高度的一致性. Gao等[29]参考人脑声源定位的双耳时间差理论 (Interaural time difference, ITD), 提出基于忆阻器阵列的类脑声源定位系统, 使用忆阻器元件来模拟脑内神经元的行为, 系统的输入信号为声音信号, 输出为声源定位的结果 (角度), 结果显示该系统具有比以往方法更高的训练准确度 (提升了45.7%), 并且大大降低了能耗 (184倍).
2)在触觉方面. 人类的触觉系统相比于机器人的触觉系统同样具有显著的优势, 例如人类触觉系统提供丰富而复杂的触觉反馈, 并具有强大的学习和适应能力, 借鉴和模拟人类触觉系统的特点可以帮助提高机器人的性能和适应性. Chou等[30]设计了一个名为CARL-SJR的机器人, 建立了一个基于生物学的脉冲神经网络模型, 用于模拟触觉信息在大脑中的处理和学习机制, 使得机器人能够感知人类的轻抚动作并与之交互. Feng等[31]提出一种基于脉冲神经网络的类脑机器人疼痛模型, 通过模拟产生疼痛的神经机制, 使机器人具备类似人类的感知“疼痛”能力, 并成功应用于实际机器人任务中, 在机器人手臂与外界发生干涉或撞击的实验中, 机器人能够感知到“疼痛”并发出警报. 张超凡等[32]针对机器人触觉感知任务中的物体滑动检测, 提出一种基于SNN的解决方案, 模型将传感器的标记点位移编码为脉冲序列, 使用SNN进行分类. 离线测试的结果表明, 该方法具有与CNN方法相当的分类性能, 且在推理速度和能耗方面具有显著优势. 此外, 由于所感知的信号种类的差异 (声光信号与物体实际的接触), 触觉感知相比视觉和听觉模态而言, 具有事件驱动的特性. 部分研究以此为基础, 引入模仿人体皮肤触觉感知神经元的传感器, 并提出基于SNN的触觉感知系统. 例如, Dabbous等[33]使用一个包含160个力传感节点的压阻式传感器阵列, 用于捕获不同的触觉模态, 将该阵列产生的压力数值编码为脉冲序列作为SNN的输入信号, 经运算后输出触觉模态分类结果. 实验结果表明, 该系统在8种触觉模态的分类测试中达到了99.97%的分类准确率. Jiang等[34]利用压电式触觉传感器中反映不同表面粗糙度的特征, 提出一种基于SNN的表面粗糙度分析方法, 将压电传感器的电信号编码为脉冲序列后使用SNN进行分类 (该文将表面粗糙度分为8个类别, 分别为Ra = 50 μm, 25 μm, 12.5 μm, 6.3 μm, 3.2 μm, 1.6 μm, 0.8 μm, 0.4 μm), 结果表明, 系统分类准确率达到了83.3%, 但对于某几个表面粗糙度存在较弱的区分能力. Kang等[35]为了解决已有的脉冲神经元在面对事件驱动的触觉感知数据时表达能力受限的问题, 提出一种新的“位置脉冲神经元”模型, 并基于此神经元进一步建立了位置脉冲响应模型 (Location spiking response model, LSRM)和位置LIF模型 (Location leaky integrate and fire, LLIF), 该文在触觉对象识别数据集和触觉滑动检测数据集上进行了分类测试, 结果显示所提出的模型具有可靠的识别性能, 并且相比于传统的人工神经网络 (Artificial neural network, ANN)具有更低的能耗. Navaraj等[36]提出一种受人皮肤启发的柔性电子皮肤, 能够编码静态和动态的外部触摸刺激, 该文将电子皮肤部署在机械手平台上 (5指), 并训练SNN处理电子皮肤输出的脉冲信号. 实验结果表明, 该机械手在触觉纹理识别任务上的准确率达到99.5%. Han等[37]提出的基于摩擦纳米发电器 (Tribo-electrification nano-generator, TENG)、Lee等[38]提出的基于压电阻传感器和阈值开关的人工触觉神经元的人工机械感受器等研究均采用了类似的研究思路, 在此不做赘述 (图3 ~ 6).
3)在空间位置的感知方面. 针对最受关注的同步建图与定位(Simultaneous localization and mapping, SLAM)这一问题, 已经有不少学者提出基于类脑智能的方法并在硬件上进行了部署, 例如, Kreiser等[41]受到动物大脑中导航类型细胞和果蝇头部方向网络的启发, 提出一种基于神经形态硬件的SNN, 该网络能够自主地匹配机器人的转动速度和学习环境中的地标位置, 并在检测到环境变化时进行更新. Yoon等[42]利用SNN模拟啮齿动物的空间认知能力, 提出了一种基于生物启发的视觉SLAM算法, 并将其部署在神经形态芯片 (NeuroSLAM)上, 系统最高能耗为23.82 mW, 实现了低功耗的SLAM. Tang等[43]同样将其提出的面向SLAM的SNN部署在英特尔的Loihi神经形态处理器上, 实验结果表明其模型具有与经典算法相当的准确性, 同时比在CPU上运行的GMapping算法节省了约100倍的能耗.
除上述提及的方向之外, 类脑智能方法在传感器故障诊断[44]、雷达数据处理[45]等方面也有应用案例, 表1总结了部分使用类脑智能方法实现机器人感知功能的文献. 从以上文献可以看出, 得益于神经科学 (尤其是计算神经科学)近年来的飞速进展, 研究者可以从部分生物的神经机制中汲取灵感来解决原有方法难以建模的问题, 从而在机器人感知外界环境的过程中具有独特的优势, 对应神经形态硬件的支持也使得机器人能够更长时间的工作.
表 1 机器人类脑感知方向文献总结Table 1 Summary of the literature of robotic brain-inspired perception模态 研究团队 传感器种类 网络结构 主要功能 视觉 Kreiser 等[41] 相机
(iCub机器人)仿脑内回路 识别物体并将注意力转移到感兴趣的物体上 Zhou等[20] 激光雷达 (LiDAR)
(文中未确切指明)层式 感知物体的三维轮廓 Ambrosano等[46] 相机
(iCub机器人)仿视网膜回路 机器人视线追踪 Li等[21] 相机 层式 手势识别和意图推断 Qiao等[25] 相机 层式 物体 (人脸)识别 Tang等[43] 距离传感器和RGB相机 多网络融合 SLAM Yoon等[42] 相机 仿脑内回路 SLAM Hussaini等[22] 相机
(文中未确切指明)层式 地点识别
(Place recognition)听觉 Deng等[27, 47] 麦克风 层式 语音分类 Zou等[4] 麦克风 层式 语音识别 Gao等[29] 麦克风 阵列式 声源定位 Liu等[28] 麦克风 仿脑内回路 声源定位 触觉 Chou等[30] 触觉传感器
(文中未确切指明)仿脑内回路 感知人类的轻抚动作 Feng等[31] 多传感器融合 仿脑内回路 机器人的“痛觉”感知 张超凡等[32] GelStereo触觉传感器 层式 触觉滑动感知 Liu等[39] 基于晶体管的电子皮肤 仿生物触觉回路 触觉记忆与学习
(以痛觉反射为例)Dabbous等[33] 压阻式传感器阵列 层式 触觉模态分类 Jiang等[34] 压电传感器 层式 表面粗糙度感知 Lee等[38] 基于压阻器和忆阻器的触觉神经元 库网络
(Reservoir)生物组织硬度感知 Han等[37] 基于摩擦纳米发电器的人工触觉感受器 层式 广范围 (3 kPa)触觉感知
(以呼吸状态辨识为例)Kang等[35] 事件驱动触觉传感器
NeuTouch[48]层式 触觉对象识别
触觉滑动检测Wen等[40] 基于压阻器和忆阻器的触觉神经元 层式 触觉感知和识别
(以MNIST分类为例)2. 类脑智能在决策方面的应用
随着任务复杂程度的增加, 机器人在实际工作过程中往往会面临各种形式的决策问题. 例如用于避障和探索的路径规划问题, 面向不同性能指标的控制策略切换问题等. 从定义上, 机器人的决策问题是指机器人如何在既定目标下综合目前环境以及自身的各类信息对未来的行为方案进行规划与选择.
作为目前最高水平的智能体, 人脑在决策和推断方面的能力依然是目前人工智能方法无法比拟的. 从机制上, 人脑的神经元可以通过不断地接受学习外部信息来改变其连接方式, 并且可以利用多脑区协同工作的方式解决复杂的问题, 还可以结合已有经验和先验知识来辅助决策, 一些研究以这些特性为灵感, 提出了相应用于解决决策问题的类脑智能算法. 例如, Zhao等[49]受到人类社会认知中理论心智 (Theory of mind, ToM)机制的启发, 提出了一个多智能体心智理论决策模型 (Multi-agent ToM decision-making, MAToM-DM) (图7), 包括一个多智能体心智理论脉冲神经网络 (Multi-agent ToM-spiking neural network, MAToM-SNN)模块和一个决策模块, 该模型利用自身经验和对其他个体的观察来推断其他个体的行为并根据预测来优化自身策略, 其实验结果表明, 引入MAToM-SNN可以提高多智能体强化学习算法在合作和竞争任务上的表现, 使得个体和集体获得更高的奖励. 左国玉等[50]模仿人类大脑中的视觉腹部通路、海马体信息通路和皮质柱信息通路构建了三个网络 (卷积感知网络、记忆图网络和贝叶斯决策网络), 提出一种将物品感知、先验知识和抓取任务融合的认知决策模型, 使机器人能够实现合理、灵活的抓取动作, 实验结果表明该模型能够根据不同的任务选择合适的物品和抓取位置. Sun等[51]受基底神经节中多巴胺反馈调节机制的启发, 从人类大脑的认知决策角度建立了一种强化学习Q-learning模型用于解决与环境交互过程中的仿生智能决策问题, 将提出的方法应用于车辆纵向自动驾驶中, 代替传统的基于规则的决策算法, 实现了类人的独立决策能力, 实验结果表明, 提出的方法能够消除加速度曲线的突变, 提高了车辆纵向自动驾驶的舒适性和适应性. Robertazzi等[52]利用神经调节理论的原理, 考虑了多巴胺、5-羟色胺和去甲肾上腺素等神经递质对元参数 (如探索/利用率、时间折扣因子等)的调节作用, 实现了面向认知控制中“动作抑制”的元学习框架 (动作抑制是认知控制的一个重要组成部分, 指在不同的认知需求之间切换时, 抑制或取消已经计划或正在进行的运动). 实验表明该模型在两种需要抑制运动反应的任务中都表现出了良好的性能, 并且随着参数的动态调整, 全局准确率和正确抑制率有所上升, 反应时间有所降低. Zhao等[53−54]受人类大脑中前额叶皮层和基底神经节的相互作用的启发, 提出一种改进的Actor-Critic算法, 引入了连续的多巴胺奖赏信号和上下文相关的奖赏信息, 以提高强化学习的速度和准确性. 该模型在多无人机的决策任务中进行了验证, 包括避开障碍物、飞越窗户和门等. 实验结果表明, 该模型比传统的Q-learning和Actor-Critic算法更快更准确地学习到正确的策略. Daglarli[55]利用脉冲神经网络、动态神经场、吸引子网络等生物启发式的神经结构, 构建了包括背外侧、腹外侧、前部和内侧四个区域的前额叶皮层计算模型, 采用深度强化学习和隐马尔科夫模型等机器学习方法, 实现了类人机器人 (Humanoid robot)的工作记忆、决策、元认知规划、空间−时间推理和推断等认知功能.
此外, 也有部分研究表明, 即使不考虑复杂的网络拓扑结构, 使用一般结构的SNN也可以解决部分规划和决策问题, 例如Rueckert等[56]提出了一种基于概率推理的循环脉冲神经网络模型用于解决有限和无限时间范围的规划问题, 其神经网络由两层神经元组成, 一层是状态神经元 (State neuron), 用于控制Agent的状态; 另一层是上下文神经元 (Context neuron), 负责提供目标和约束信息, 用该网络控制机械臂实验表明神经网络可以同时表征多个任务解决方案, 并通过激活不同的上下文神经元来适应不同的任务. Skorheim等[57]构建了一个包含三层兴奋性和抑制性神经元的网络模型, 并使用奖励调制的STDP (Reward-modulated spike-timing-dependent plasticity, R-STDP)机制模拟生物的觅食行为, 其实验表明该模型能够通过学习在环境中实现高效的决策制定. Liu等[58]设计了一种受生物侧抑制连接启发的任务切换机制 (图8), 使SNN能够根据传感器感知到的信息在不同的突触上增加输入电流, 从而激活不同的突触, 使SNN在不同的任务之间进行切换. 实验结果表明, 引入该机制后可以实现基于环境信息的避障任务和追踪任务的自动切换, 即移动机器人可以在追踪目标过程中灵活地避障, 在完成避障之后继续追踪目标.
从上述研究可以看出, 对于一个既定的复杂任务, 机器人的决策问题和控制问题常常是共同出现的, 而类脑智能模型在接收外部环境信息后, 通过与控制器串联 (先选择下一步的“行为”, 再让控制器实现所选择的“行为”[53−54])、影响控制器参数[58−59]或者向控制器传递除命令之外的某些信息[49]的方式实现类脑智能决策, 这与人脑执行任务过程中的神经学过程也是类似的. 表2总结了使用类脑模型实现机器人决策的部分文献.
表 2 机器人类脑决策方向文献总结Table 2 Summary of the literature of robotic brain-inspired decision模型结构 研究团队 模型输入 模型输出 主要功能 循环结构 Rueckert等[56] 某一时刻Agent的状态
(例如空间位置)未来一段时间内的位置规划
(位置序列)有限和无限时间范围的任务规划问题 仿脑内回路 Zhao等[53] 视觉图像信息
(State)动作决策
(无人机的前进、后退、向左、向右)无人机飞行 (穿过窗户)过程决策任务 Daglarli等[55] 视觉信息
听觉信息机器人行为序列
决策奖励信号机器人的类人决策
(情感、注意力、意图等推理)左国玉等[50] 关于任务、记忆、 观测Affordance
和标签的词语该物品的Affordance (可以抓取)
或者该物品所需Affordance
的建议 (不可抓取)根据不同的任务选择合适的
物品和抓取位置Robertazzi等[52] 模拟视觉刺激
(方波信号)机器人动作
(向左、向右、保持不动)实现机器人在指定任务需求下的“动作抑制” Huang等[59] 状态预测误差
奖励预测误差预测时域调整量 在MB和MF控制之间
切换 (图9)层式 Liu等[58] 传感器感知到的数据 (转换为脉冲序列) 机器人控制量 (左右轮速度) 针对不同的任务需求激活不同的突触
实现控制策略的切换Skorheim等[57] 视觉感知信息
(7×7矩阵)运动决策信息
(3×3矩阵)实现虚拟环境中Agent的觅食决策 Zhao等[49] 环境上下文信息与对其他
个体状态和行为的观测对其他个体未来行为的预测 多智能体协同决策 3. 类脑智能在控制方面的应用
机器人的控制问题是指控制器如何控制或驱动机器人的硬件设备完成既定决策, 相比于决策问题, 控制问题更关心每一时刻系统的控制量的计算. 本节根据这一定义, 对已有的相关研究进行总结归纳.
3.1 类脑控制在各类机器人上的应用
近年来随着各类仿生机器人的出现和发展, 基于类脑智能的机器人运动控制算法已经逐渐成为了研究人员关注的热点问题. 从控制对象上, 应用类脑方法的机器人主要是仿生移动机器人 (机器鱼、多足爬行机器人等仿生机器人)以及机械臂. 例如, Naya等[5]提出了将SNN应用于六足机器人的移动控制, 降低了运动过程的能量成本. Jiang等[60]提出了一种利用神经形态视觉传感器(Neuromorphic vision sensor, NVS)和脉冲神经网络 (SNN)实现蛇形机器人对管状物体的检测和跟踪的方法, 根据目标物体在传感器图像上的宽度变化和蛇形机器人的运动速度来估计目标物体与机器人之间的距离; 根据目标物体在水平方向上的偏移量和机器人头部的摆动幅度来估计目标物体与机器人中心线之间的偏移量; 最终根据偏移量生成控制信号, 使机器人在保持滑行姿态的同时调整运动方向. Wang等[61]模拟了高等生物运动时神经信号的传递过程, 提出了一种基于脉冲神经元模型和CPG (Central pattern generator)的仿生机器鱼运动控制方法, 完成了机器鱼的直线游动、转弯游动、8字绕环和逆流游动控制, 并指出这种方法在稳定性上具有优势. 也有部分研究利用SNN控制多足机器人在移动过程中的步态, 例如Lele等[62]利用监督学习的方式, 根据期望和实际的神经元放电模式之间的差异来调整突触权重, 实现了六足机器人的前进和左右转向三种步态 (图10). 部分研究指出这种方法具有良好的鲁棒性和适应性.
部分文献表明SNN也可以用于解决传统移动机器人的智能控制问题, 例如, Cao等[63]提出了一种基于脉冲神经网络 (SNN)的移动机器人目标跟踪控制器, 该控制器可以同时处理来自CCD摄像头、编码器和超声波传感器的环境信息和目标信息, 实现了机器人对移动目标的自主追踪. Lu等[64]提出了一种基于R-STDP的简单脉冲神经网络, 用于控制小车通过超声传感器感知障碍物并避障. Bing等[65]针对移动机器人的车道保持任务, 对比了两种基于SNN的控制器 (R-STDP和DQN-SNN)及基于深度强化学习的控制器的控制性能, 结果显示基于R-STDP控制器具有最低的车道中心距离误差和最快的训练时间. 还有诸多研究针对普通移动机器人提出了基于SNN的导航、跟踪与避障控制器[66−75], Lobov等[75]在论文中指出, 基于STDP学习机制的SNN控制器具有环境适应和重新学习的能力, 这种能力是通过突触连接强度的动态变化实现的.
除了上述两种机器人外, 类脑控制方法在机械臂控制问题中也有诸多应用案例, 其中一些文献借鉴了小脑及其相关脑区在人体运动规划与控制中起到的重要作用[76], 开发了基于小脑功能机制的类脑智能控制方法. 例如Xing等[7]利用脉冲卷积神经网络 (用于实现小脑网络的控制功能)、液体状态机 (用于实现类似大脑前额叶皮层的预测功能)等方法模拟了大脑的多个区域 (图11), 将其应用于小空间操作机器人上, 实验表明该控制方法可以实现高精度、动态和无碰撞的运动控制. Abadia 等[6]提出的机械臂控制器利用SNN建模了小脑的颗粒细胞、平行纤维、浦金野细胞等部分 (图12), 模仿了小脑的神经回路和突触可塑性机制, 使得SNN控制器能够通过多种历史信息 (传感器数据、机械臂运动信号)来预测运动指令, 使得整个系统对终端控制指令的未知随机延迟 (文章认为终端解算过程和命令传输过程会造成时间上的延迟)具有鲁棒性, 并通过实际的远程控制实验证明了这一结论. 还有部分文献[73, 77]采用类似结构的小脑模型实现了机械臂末端的轨迹跟踪, 并分别通过实机实验[77]和仿真平台[73]验证了其方法的有效性.
3.2 类脑控制与常见智能控制方法的结合
部分类脑方法与常见的智能控制方法 (强化学习[5, 78−79]、预测控制理论[6−7, 80−83]等)相结合, 取得了性能上的拓展与提升.
强化学习方面, Naya等[5]在论文中分别对比了三种不同的深度强化学习算法 (SAC, TD3和DDPG)与SNN结合前后的性能差异, 其SNN的输入特征为对环境的观察信息, 经过编码模块后转换为脉冲序列, 经SNN处理后再用解码模块转换为动作指令 (Action). 文献[5]定义运输成本为驱动机器人关节所需能量与移动距离和自身重力乘积的比值, 在仿真环境中团队发现原算法与SNN结合后, 机器人的运输成本平均降低了15.8%, 而且在更大的能耗惩罚下也不容易陷入局部最小. Oikonomou等[78]采用了类似的思路, 设计了一种基于Actor-Critic强化学习的双指操作机器人, 将其中的Actor网络类型替换为脉冲神经网络, 用于控制仿真环境中的机械臂执行一个两阶段任务, 在第1阶段 (控制机械臂到达指定位置)中, 仿真实验结果表明与SNN的结合使得整个模型的成功率提升了接近40%, 但执行时间平均延长了1 s; 在第2阶段 (抓取物体并且搬运)中, 仿真实验结果表明SNN使模型成功率提升了30%, 并且具有相当的平均执行时间. SNN在强化学习方向的典型应用如图13[79]所示.
预测控制方面, Cao等[81]提出了一种基于回声状态高斯过程 (Echo state Gaussian process, ESGP)和SNN的非线性预测控制策略, ESGP是将回声状态网络 (Echo state network, ESN)与高斯过程 (Gaussian processes, GP)相结合的用于建模非线性系统的方法. 文章使用ESGP预测气动肌肉执行器 (Pneumatic muscle actuator, PMA)的状态, 再使用另一个单层神经网络(文中将此网络作为Controller)生成控制命令. 文章针对状态跟踪这一控制任务设计了代价函数, 基于这一代价函数的梯度信息调整Controller内部的各项参数, 并在一个PMA实验平台上验证了所提出的控制策略的有效性. 实验结果表明, 该控制策略能够实现PMA的高精度轨迹跟踪, 且具有较好的鲁棒性和适应性. Zhang等[80]在机械臂的控制中提出了类似的思路, 同样使用ESN模拟了小脑中颗粒层的特征 (图14), 通过增量学习与岭回归结合的方式使得该网络能够准确地预测机器人在不同分布的时序控制信号下的运动结果, 随后的纠正网络会根据预测误差生成控制量的矫正量, 进而实现跟踪控制.
表 3 机器人类脑控制方向文献总结Table 3 Summary of the literature of robotic brain-inspired control机器人类型 研究团队 网络输入 网络输出 主要功能 移动机器人 Lobov等[75]
(2-DoF)超声传感器信息
触觉传感器信息控制信号
(电机驱动量)移动控制
(避障)Wang等[68]
(2-DoF)声纳传感器信息 控制信号
(电机驱动量)移动控制
(避障、追踪、沿墙行走)Bing等[65]
(2-DoF)DVS图像信息 控制信号 (电机转速) 移动控制
(车道保持)Liu等[70]
(4-DoF)超声传感器 控制指令
(左、右、前进)移动控制
(避障)Lu等[64]
(4-DoF)超声传感器感知到的距离信息 控制信号 (转换为
左右轮转速)移动控制
(避障)机械臂 Xing等[7]
(未说明)其他网络输出的运动参数 关节驱动力矩 对小空间操作机器人
实现精确控制Chen等[86]
(1-DoF)期望角度与传感器信息 控制信号补偿量 提高机械臂的适应性和鲁棒性 IAbadia等[6]
(6-DoF)轨迹规划器生成的轨迹参数 机械臂控制力矩 通过预测运动指令实现对
指令延迟的鲁棒性Zahra等[87]
(6-DoF)目标运动状态与机械臂内部传感器信息 机械臂驱动量 提高机械臂在操作任务中的
精度和运动协调性Carrillo等[77]
(2-DoF)期望机械臂状态与目标信息 肩部与肘部驱动转动的调整量 机械臂运动控制 Zahra等[82]
(6-DoF)关节角速度变化量 机器人状态预测 降低机械臂的运动误差和执行时间 Zhang等[80]
(2-DoF)原始控制信号 控制信号纠正量 提升机械臂的运动精度 仿生机器人 Naveros等[88]
(3-DoF)感知运动信号
(Sensory-motor signal)眼球转动量 根据机器人头部转动控制机器人
眼球转动 (前庭眼反射)Naya等[5]
(24-DoF)状态观测量
(State)行动
(Action)考虑能耗成本的步态学习 Lele等[62]
(6-DoF)DVS图像信息 步态选择 移动控制 (捕猎) Espinal等[89]
(12-DoF)− 步态生成 作为CPG生成指定的步态 Jiang等[60]
(未说明)NVS图像信息 (神经形态视觉传感器) 控制信号
(传递给CPG生成对应的步态)移动控制
(目标追踪)− Wilson等[83] 控制信号 控制对象的状态预测 实现一般线性系统的自适应控制 注: DoF (Degree of freedom)为对应文献中机器人的自由度. 3.3 基于计算神经建模的类脑控制方法
除了以上关于SNN的机器人控制方法研究外, 近年来还有一些基于神经计算建模的动力学系统控制方法, 其中具有代表性的就是Lechner等[84]提出的基于液态时间常数网络 (Liquid time constant network, LTC)的神经回路策略 (Neural circuit policies, NCP) (图15). 该方法没有采用SNN原有的脉冲交互的机制, 神经元与神经元之间信息交互的方式完全依靠膜外电位的变化, 这一方法从根本上避免了脉冲信号产生的不可导的问题, 从而可以实现基于梯度的模型训练. 该控制器使用车辆上搭载的摄像头拍摄到的道路图片作为输入特征, 经过卷积层提取特征后作为“感知神经元”膜电位动力学模型的外部电流输入量, 经过中间两层神经元的传递后最终到达运动神经元, 将运动神经元的膜外电位转换为车辆方向盘的转角, 从而完成对车辆前进方向的控制. 但由于Lechner 等[84]采用了精确神经元模型, 在求解过程中需要使用数值方法求解复杂的神经元动力学微分方程, 因此对计算资源的要求较高, 也限制了大规模神经网络的开发与训练. 因此其团队的Hasani等[85]在2022年提出了针对LTC的近似解析解(Closed-form continuous-depth network, CfC), 该网络在具有充足表达能力的同时大幅提升了计算效率, 未来有望在机器人系统上取得更广泛的应用. 表3总结了使用类脑模型作为控制器的部分文献.
从上述研究可以看出, 针对于简单的控制任务, 类脑智能方法可以直接构建从外部环境信息到控制量之间的控制器[65], 这一点相对于传统控制方法而言具有端到端的设计简便优势, 对于相对复杂的任务, 类脑控制器往往与第2节中所介绍的类脑决策模型相结合[7]. 除此之外, 类脑智能方法在动力学系统状态预测问题上具有良好的表现, 一方面可以用于在不易建模的系统中实现预测控制[81], 另一方面可以提升整个系统对外部各种干扰的鲁棒性[6].
4. 类脑智能目前的主要问题和未来的发展方向
类脑智能方法作为近年来新兴的研究方向, 已经取得了一定的研究进展和成果, 但还有诸多问题和限制亟待解决, 本节将从软件和硬件两个角度来分别对其进行论述, 并提出目前部分亟需解决的研究问题.
4.1 软件层面
软件层面的主要问题可以进一步划分为类脑智能的模型设计问题和模型训练问题. 其中, 模型设计需要从大量的神经学现象以及多种神经元行为模式中提取出核心机制, 这对神经科学和信息科学等领域的跨学科合作提出了更高的要求. 以类脑智能在机器人感知上的应用为例, 目前的类脑模型往往只局限于大脑机制某一个很小的部分, 这会导致模型无法像真实的大脑皮层一样获取到外部输入的语义、概念、记忆等高级信息[8]. 除了对某一特定模态的建模之外, 目前的类脑模型很少考虑到人脑中多种认知功能的相互作用与调节, 这使得模型的生物合理性受到制约, 也影响了最终模型的智能化水平[10]. 未来需要开展更多脑启发的智能机器人的感知、决策、认知和运动控制方面的研究, 通过系统且全面地模仿生物行为和神经机制, 提高机器人的性能.
与模型设计相对应的是类脑智能模型的训练问题, 有研究指出, 目前的脉冲神经网络(类脑智能的代表性方法)需要有效的学习算法来训练和调整内部的参数, 这些算法目前还不够高效和稳定[90]. 这些限制主要来源于SNN的脉冲活动会导致梯度在时空维度上的传播受到阻碍, 从而造成梯度爆炸或消失的问题, 同时随着SNN模拟步长的增加, 梯度误差会变得愈发严重从而使网络无法收敛[91]. 针对这一问题, 亟需探索更鲁棒、更高效、适用于复杂任务场景和小样本学习的类脑模型训练方法, 也可以利用传统机器学习方法(如CNN)在图像、声音等领域的性能优势[90], 提出类脑智能与传统机器学习相结合的新方案, 实现优势互补.
此外, 生物的神经系统具有随着环境变化和任务进行调整和进化的能力, 这种能力有助于实现机器人的自适应控制和决策. 为了让类脑智能模型具备进化的功能, Shen等[92]提出了基于全局误差和STDP的神经回路进化策略“NeuEvo”, 该模型可以根据当前时刻的表现动态地调整内部神经回路的突触权重, 在多个任务的表现中都取得了具有竞争力的性能. 因此, 类脑模型未来可以在这一方向上开展更多的研究, 从而加强模型在控制和决策任务上的适应性和鲁棒性.
4.2 硬件层面
类脑智能的硬件实现目前主要指SNN在各类硬件平台上的部署, 常见的计算平台有CPU和GPU、基于数字电路或者模拟电路的神经形态芯片 (Neuromorphic chips)以及FPGA[93]. 其中CPU和GPU作为通用的计算平台, 虽然可以用于模拟SNN的时钟驱动模式, 但所需的时间较多且能耗较高(尤其在模拟大规模的SNN时). 神经形态芯片从电路上模拟了神经元的放电模式, 可以在较低的功耗下模拟大规模的SNN, 但同样存在诸多限制. 在性能上, 目前的神经形态芯片大多只是将其他平台训练好的SNN (参数已经固定)以芯片的形式实现出来, 缺乏在线的芯片内学习(On-chip learning)能力[93], 这导致芯片本身不能模拟人脑适应外部变化的特性; 此外, 监督学习中所需要的复杂的突触可塑性模型在目前的芯片上依然难以实现. 在应用上, 除去芯片本身的制造难度之外, 基于模拟电路的神经形态芯片容易受到外部温度、供电电压等外部干扰因素的影响, 鲁棒性较弱[93]; 且神经形态芯片缺乏统一的编程接口和标准[12, 93], 这同样也限制了多个平台之间的互操作性.
上述问题的解决除了依靠芯片制造水平的提升之外, 还可以从其他方面入手. 从机器人开发的角度, 部分任务并不需要大规模的SNN, 因此使用FPGA的SNN部署方案具有相比于神经形态芯片更高的灵活性、更短的设计与实现周期及更好的稳定性. 目前已经有部分基于FPGA实现SNN的研究案例[94−95], 未来可以在此基础上建立FPGA实现类脑智能的研究范式及通用软硬件平台[14], 便于相关研究的共享与验证.
4.3 未来亟需研究的热点问题
1)兼具高表达能力和低计算成本的神经计算模型
神经计算模型是类脑智能机器人用于模拟生物神经系统感知、决策和控制的基础. 目前在类脑智能领域广泛使用的神经元模型和突触模型仍然存在诸多限制, 例如表达能力较弱(目前广泛使用的LIF神经元模型只能模拟非常有限的生物神经元发放行为)、生物可解释性和可信度较低 (常用的神经元模型往往没有建模离子通道的动态特性)等. 而采用具备优秀生物合理性的模型在实际应用中往往需要庞大的计算资源. 这些限制导致部署在机器人上的类脑智能模型很难具备真实生物神经系统丰富的动力学性质和动态可塑的适应能力. 因此, 需要借鉴计算神经科学的先进案例, 开展更多关于神经元和突触计算建模的相关研究.
2)多模态类脑感知融合与注意力机制
在感知外部世界的过程中, 大脑各个模态的信息 (视、听、触、味觉)并不是孤立的, 大脑会整合各个模态中不同尺度的信息, 即使在数据量较少且包含噪声的前提下, 也可以完成对外部环境的感知和学习, 这种特性是目前任何一种智能都难以企及的. 因此, 在机器人感知领域, 需要研究多模态融合的感知方法和模型, 将不同通道的信息进行对齐和融合, 使机器人能够从多个感知模态中学习, 并从中提取更高层次、更丰富的知识和表征. 另一方面, 应该借鉴Transformer在计算机视觉领域取得的巨大成功, 提出低计算成本的基于类脑智能的注意力机制, 使机器人能够动态地调整对不同感知模态的关注程度, 以应对不同任务和环境条件.
3)脑启发的自我学习算法
人脑的另一个主要优势是其具备的持续学习能力, 具体而言, 无论是感知、决策还是控制任务, 人脑都可以根据自身实践获得的经验或者其他资料的帮助对已经学习到的各项技能进行调整和提升. 这种出色的无监督或自监督学习能力可以在很大程度上提升机器人系统面对未知场景的泛化能力, 从而大幅拓展应用场景. 但目前, 类脑智能模型在无监督条件下的总体性能还有较大的提升空间. 因此, 未来可以借鉴目前已有的迁移学习、增量学习以及元学习的相关案例, 对类脑智能模型已有的学习算法进行突破和创新.
5. 结束语
本文针对近年来广受关注的类脑智能方法在机器人领域的理论与应用, 从感知、决策和控制三个机器人的基本功能入手进行了总结与归纳, 同时针对其目前存在的问题和未来发展方向, 从软件和硬件两个方面进行了分析. 概括而言, 随着计算神经科学和类脑深度学习等领域取得的各项进展, 类脑智能方法在机器人领域已经展现出了其独特的优势, 但还有诸多问题需要进一步思考与讨论. 一方面, 针对类脑智能机器人的研究依然需要从实际问题和需求出发, 充分挖掘生物神经系统的行为特性与机制, 未来的研究可能会从计算神经科学汲取灵感, 引入更详细的生物学细节, 平衡计算效率与生物合理性, 提升类脑智能模型的学习能力. 另一方面, 未来相关研究也可以充分挖掘已有类脑智能模型的优势和长处, 并与已经被广泛使用的传统机器学习、智能控制等理论进行更深度的融合, 各取所需、取长补短, 最终提升整个系统的智能化水平.
-
表 1 各种生物特征模板保护算法的比较结果
Table 1 Comparative result of various biometric template protection methods
可撤销方案 转换方式 相似性 缺点 Biohashing[5] 随机投影+二值化处理 汉明距离 原始模板可由折衷密钥推算出来 Wang等[12] 离散傅里叶变换+随机投影 汉明距离 性能下降 Bloom filter[13] Bloom filter (十进制到二进制映射) 汉明距离 易受暴力攻击 P-MCC[17] KL投影+二值化 汉明距离 可撤销性弱 2P-MCC[18] 完全/部分置换 汉明距离 用户需要管理密钥 GRP-based IoM Hashing[10] 多重随机投影+记录最大值索引 欧氏距离 性能下降 URP-based IoM Hashing[10] 置换+记录最大值索引 欧氏距离 性能下降 BioEncoding[19] 布尔函数 汉明距离 易受ARM攻击 表 2 WSE哈希处理效率(s) (${m}=1 000$, ${k}=3$)
Table 2 Processing efficiency of WSE Hashing (s) (${m}=1 000$, $k=3$)
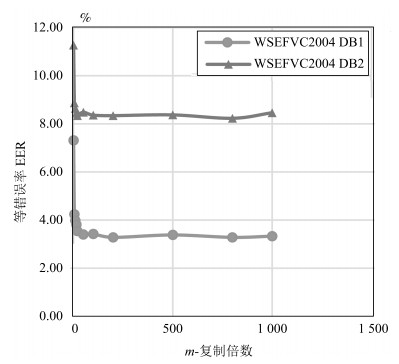
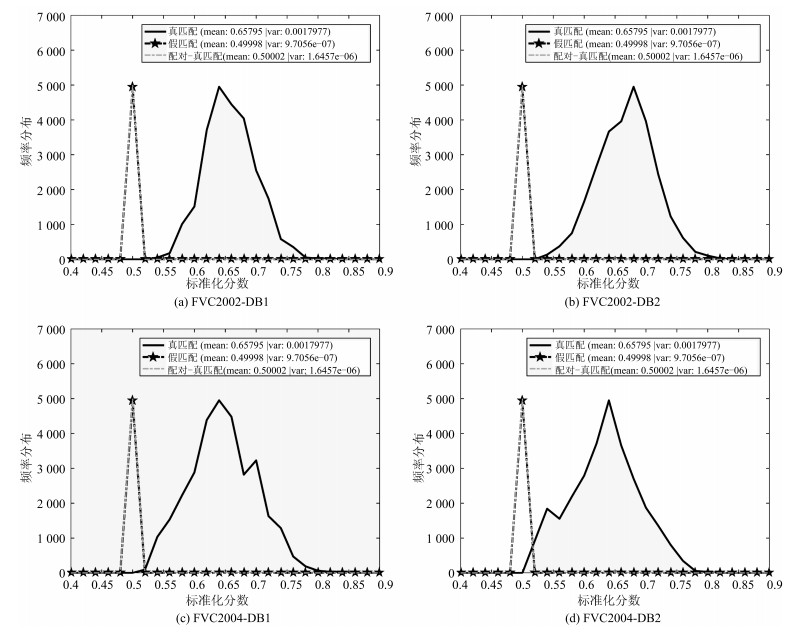
平均时间 FVC2002-DB1 FVC2002-DB2 FVC2004-DB1 FVC2004-DB2 注册阶段 0.035031 0.034884 0.034481 0.033151 验证阶段 0.034896 0.034874 0.034621 0.034647 表 3 不同方法的性能精度对比(EER) (%)
Table 3 EER comparison between proposed method and other methods (%)
方法 FVC2002-DB1 FVC2002-DB2 FVC2004-DB1 FVC2004-DB2 WSE Hashing 0.2 0.62 2.6 7.13 Binary fingerprint vector (Baseline)[11] 0.26 0.12 1.58 4.39 URP-based IoM Hashing[10] 0.46 2.1 4.51 8.02 GRP-based IoM Hashing[10] 0.22 0.47 4.74 4.1 Bloom filter[24] 2.3 1.8 13.4 8.1 2P-MCC$_{64, 64}$[18] 3.3 1.8 6.3 _ EFV Hashing[7] 0.32 0.63 2.62 7.14 表 4 不可链接性的全局度量($D_{\underset\longleftrightarrow{{\rm sys}}}$) (${m}=1 000$, ${k}=3$)
Table 4 Global measure ($D_{\underset\longleftrightarrow{{\rm sys}}}$) of unlinkability (${m}=1 000$, ${k}=3$)
方法 FVC2002-DB1 FVC2002-DB2 FVC2004-DB1 FVC2004-DB2 WSE Hashing 0.0257 0.0235 0.0271 0.0250 EFV Hashing[7] 0.0404 0.0473 0.0465 0.0459 -
[1] 张宁, 臧亚丽, 田捷. 生物特征与密码技术的融合-一种新的安全身份认证方案. 密码学报, 2015, 2(2): 159-176 https://www.cnki.com.cn/Article/CJFDTOTAL-MMXB201502006.htmZhang Ning, Zang Ya-Li, Tian Jie. The integration of biometrics and cryptography-a new solution for secure identity authentication. Journal of Cryptologic Research, 2015, 2(2): 159-176 https://www.cnki.com.cn/Article/CJFDTOTAL-MMXB201502006.htm [2] 许秋旺, 张雪锋. 基于细节点邻域信息的可撤销指纹模板生成算法. 自动化学报, 2017, 43(4): 645-652 doi: 10.16383/j.aas.2017.c160069Xu Qiu-Wang, Zhang Xue-Feng. Generating cancelable flngerprint templates using minutiae local information. Acta Automatica Sinica, 2017, 43(4): 645-652 doi: 10.16383/j.aas.2017.c160069 [3] 王慧珊, 张雪锋. 基于Biohashing的指纹模板保护算法. 自动化学报, 2018, 44(4): 760-768 doi: 10.16383/j.aas.2017.c170056Wang Hui-Shan, Zhang Xue-Feng. Improved biohashing fingerprint template protection algorithms. Acta Automatica Sinica, 2018, 44(4): 760-768 doi: 10.16383/j.aas.2017.c170056 [4] 梁耀, 冯冬芹, 徐珊珊, 陈思媛, 高梦州. 加密传输在工控系统安全中的可行性研究. 自动化学报, 2018, 44(3): 434-442 doi: 10.16383/j.aas.2018.c160399Liang Yao, Feng Dong-Qin, Xu Shan-Shan, Chen Si-Yuan, Gao Meng-Zhou. Feasibility analysis of encrypted transmission on security of industrial control systems. Acta Automatica Sinica, 2018, 44(3): 434-442 doi: 10.16383/j.aas.2018.c160399 [5] Jin A T B, Ling D N C, Goh A. Biohashing: two factor authentication featuring fingerprint data and tokenised random number. Pattern Recognition, 2004, 37(11): 2245-2255 doi: 10.1016/j.patcog.2004.04.011 [6] Patel V M, Ratha N K, Chellappa R. Cancelable biometrics: A review. IEEE Signal Processing Magazine, 2015, 32(5): 54-65 doi: 10.1109/MSP.2015.2434151 [7] Lee M J, Jin Z, Teoh A B J. One-factor cancellable scheme for fingerprint template protection: extended feature vector (EFV) Hashing. In: Proceedings of the 2018 IEEE International Workshop on Information Forensics and Security. New York, USA: IEEE, 2018. 1-7 [8] Jin Z, Lim M H, Teoh A B J, Goi B M, Tay Y H. Generating fixed-length representation from minutiae using kernel methods for fingerprint authentication. IEEE Transactions on Systems, Man, and Cybernetics: Systems, 2016, 46(10): 1415-1428 doi: 10.1109/TSMC.2015.2499725 [9] Wang S, Deng G, Hu J K. A partial Hadamard transform approach to the design of cancelable fingerprint templates containing binary biometric representations. Pattern Recognition, 2017, 61: 447-458 doi: 10.1016/j.patcog.2016.08.017 [10] Jin Z, Hwang J Y, Lai Y L, Kim S, Teoh A B J. Ranking-based locality sensitive hashing-enabled cancelable biometrics: Index-of-max hashing. IEEE Transactions on Information Forensics and Security, 2018, 13(2): 393-407 doi: 10.1109/TIFS.2017.2753172 [11] Cheung K H, Kong A W K, You J, Zhang D. An analysis on accuracy of cancelable biometrics based on biohashing. In: Proceedings of the 2005 International Conference on Imaging Science, Systems, and Technology. Berlin, Germany: Springer-Verlag, 2005. 40-45 [12] Wang S, Hu J K. Alignment-free cancelable fingerprint template design: A densely infinite-to-one mapping (DITOM) approach. Pattern Recognition, 2012, 45(12): 4129-4137 doi: 10.1016/j.patcog.2012.05.004 [13] Rathgeb C, Breitinger F, Busch C, Baier H. On application of bloom filters to iris biometrics. IET Biometrics, 2014, 3(4): 207-218 doi: 10.1049/iet-bmt.2013.0049 [14] Hermans J, Mennink B, You J, Peeters R. When a bloom filter is a doom filter: Security assessment of a novel iris biometric te mplate protection system. In: Proceedings of the 2014 Biometrics Special Interest Group. New York, USA: IEEE, 2014. 1-6 [15] Bringer J, Morel C, Rathgeb C. Security analysis of bloom filter-based iris biometric template protection. In: Proceedings of the 2015 International Conference on Biometrics. New York, USA: IEEE, 2015. 527-534 [16] Cappelli R, Ferrara M, Maltoni D. Minutia cylinder-code: A new representation and matching technique for fingerprint recognition. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2010, 32(12): 21-28 http://ieeexplore.ieee.org/document/5432197 [17] Ferrara M, Maltoni D, Busch C, Cappelli R. Noninvertible minutia cylinder-code representation. IEEE Transactions on Information Forensics and Security, 2012, 7(6): 1727-1737 doi: 10.1109/TIFS.2012.2215326 [18] Ferrara M, Maltoni D, Cappelli R. A two-factor protection scheme for MCC fingerprint templates. In: Proceedings of the 2014 Biometrics Special Interest Group. New York, USA: IEEE, 2014. 1-8 [19] Ouda O, Tsumura N, Nakaguchi T. Tokenless cancelable biometrics scheme for protecting iriscodes. In: Proceedings of the 2010 International Conference on Pattern Recognition. New York, USA: IEEE, 2010. 882-885 [20] Kang J, Nyang D H, Lee K H. Two-factor face authentication using matrix permutation transformation and a user password. Information Sciences, 2014, 269(8): 1-20 [21] Maio D, Maltoni D, Cappelli R, Wayman J, Jain A K. FVC2002: Second fingerprint verification competition. In: Proceedings of the 16th International Conference on Pattern Recognition. New York, USA: IEEE, 2002. 811-814 [22] Maio D, Maltoni D, Cappelli R, Wayman J, Jain A K. FVC2004: Third fingerprint verification competition. Biometric Authentication. Berlin: Springer-Verlag, 2004. 1-7 [23] Cappelli R, Maio D, Maltoni D, Wayman J L, Jain A K. Performance evaluation of fingerprint verification systems. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2006, 28(1): 3-18 doi: 10.1109/TPAMI.2006.20 [24] Li G Q, Yang B, Rathgeb C, Busch C. Towards generating protected fingerprint templates based on bloom filters. In: Proceedings of the 2015 International Workshop on Biometrics and Forensics. New York, USA: IEEE, 2015. 1-6 [25] Gomez-Barrero M, Galbally J, Rathgeb C. General framework to evaluate unlinkability in biometric template protection systems. IEEE Transactions on Information Forensics and Security, 2018, 13(6): 1406-1420 doi: 10.1109/TIFS.2017.2788000 [26] Tams B, Mihailescu P, Munk A. Security considerations in minutiae-based fuzzy vaults. IEEE Transactions on Information Forensics and Security, 2017, 10(5): 985-998 http://smartsearch.nstl.gov.cn/paper_detail.html?id=5786ed8a1699649291bcd06d0610383d 期刊类型引用(0)
其他类型引用(7)
-





 下载:
下载:















 下载:
下载: