Survey on Causality Analysis of Multivariate Time Series
-
摘要:
多元时间序列的因果关系分析是数据挖掘领域的研究热点. 时间序列数据包含着与时间动态有关的、未知的、有价值的信息, 因此若能挖掘出这些知识进而对时间序列未来趋势进行预测或干预, 具有重要的现实意义. 为此, 本文综述了多元时间序列因果关系分析的研究进展、应用与展望. 首先, 本文归纳了主要的因果分析方法, 包括Granger因果关系分析、基于信息理论的因果分析和基于状态空间的因果分析; 然后, 总结了不同方法的优缺点、适用范围和发展方向, 并概述了其在不同领域的典型应用; 最后, 讨论了多元时间序列因果分析方法待解决的问题和未来研究趋势.
-
关键词:
- 多元时间序列 /
- Granger因果分析 /
- 转移熵 /
- 状态空间
Abstract:The causality analysis of multivariate time series is a research hotspot in data mining. Time series data contains unknown, valuable information related to temporal dynamics. Therefore, it is of great practical significance to be able to mine these knowledge and then predict or intervene the future trend of time series. For this reason, this paper reviews the research progress, application and prospects of causality analysis of multivariate time series. Firstly, this paper summarizes the main causality analysis methods, including Granger causality analysis, causality analysis based on information theory and causality analysis based on state space. Then, we summarize the advantages and disadvantages, scope of application and development directions of different methods, and outline their typical applications in different fields. Finally, the problems to be solved and future research trends of the causality analysis methods of multivariate time series are discussed.
-
Key words:
- Multivariate time series /
- Granger causality analysis /
- transfer entropy /
- state space
-
表 1 Granger因果关系分析及其改进方法
Table 1 Granger causality analysis and its improvement methods
类别 研究者 发表年份 方法名称 文献 Granger因果模型 Granger 1969 Granger 因果指数 (GCI) [15] 条件Granger因果模型 Geweke 1982 条件 Granger 因果指数 (CGCI) [23] Chen 等 2004 条件扩展 Granger 因果指数 (CEGCI) [24] Siggiridou 等 2016 限制条件 Granger 因果指数 (RCGCI) [25] Lasso-Granger因果模型 Arnold 等 2007 Lasso-Granger 因果模型 [26] Shojaie 等 2010 截断 Lasso-Granger 因果模型 [27] Bolstad 等 2011 Grouped-Lasso-Granger 因果模型 [28] Yang 等 2017 Grouped-Lasso 非线性条件 Granger 因果模型 [29] 非线性Granger因果模型 Ancona 等 2004 RBF-Granger 因果模型 [30] Marinazzo 等 2008 Kernel-Granger 因果模型 [31-32] Wu 等 2011 KCCA-Granger 因果模型 [33] Hu 等 2014 Copula-Granger 因果模型 [34] Montalto 等 2015 NN-Granger 因果模型 [35] 频域Granger因果模型 Geweke 1982 Spectral-Granger 因果模型 [23] Baccalá 等 2001 偏定向相干性 (PDC) [36] Kamiński 等 2001 直接传递函数 (DTF) [37] 表 2 基于信息理论的因果关系分析方法
Table 2 Causality analysis methods based on information theory
表 3 因果分析方法应用范围比较
Table 3 Comparison of application range of causality analysis methods
研究者 方法名称 非线性 多变量 非平稳 文献 Granger Granger 因果指数 [15] Geweke 条件 Granger 因果指数 √ [23] Chen 等 条件扩展 Granger 因果指数 √ √ [24] Siggiridou 等 限制条件 Granger 因果指数 √ √ [25] Arnold 等 Lasso-Granger 因果模型 √ [26] Shojaie 等 截断 Lasso-Granger 因果模型 √ [27] Bolstad 等 Grouped-Lasso-Granger 因果模型 √ [28] Yang 等 Grouped-Lasso 非线性条件 Granger 因果模型 √ √ [29] Ancona 等 RBF-Granger 因果模型 √ [30] Marinazzo 等 Kernel-Granger 因果模型 √ √ [31-32] Wu 等 KCCA-Granger 因果模型 √ √ [33] Hu 等 Copula-Granger 因果模型 √ √ [34] Montalto 等 NN-Granger 因果模型 √ √ [35] Geweke Spectral-Granger 因果模型 √ [23] Baccalá 等 偏定向相干性 √ [36] Kamiński 等 直接传递函数 √ [37] Schreiber 转移熵 √ [40] Staniek 等 符号转移熵 √ √ [42] Kugiumtzis 偏符号转移熵 √ √ √ [43] Faes 等 条件熵 √ √ [44] Frenzel 等 偏互信息 √ √ [45] Kugiumtzis 基于混合嵌入的偏互信息 √ √ [46] Arnhold 等 非线性相互依赖指标 S 和 H √ [61] Quiroga 等 非线性相互依赖指标 N √ [62] Andrzejak 等 非线性相互依赖指标 M √ [63] Chicharro 等 非线性相互依赖指标 L √ √ [64] Sugihara 等 收敛交叉映射 √ [65] -
[1] 高月, 宿翀, 李宏光. 一类基于非线性PCA和深度置信网络的混合分类器及其在PM2.5浓度预测和影响因素诊断中的应用. 自动化学报, 2018, 44(2): 318−329Gao Yue, Su Chong, Li Hong-Guang. A kind of deep belief networks based on nonlinear features extraction with application to PM2.5 concentration prediction and diagnosis. Acta Automatica Sinica, 2018, 44(2): 318−329 [2] Han M, Liu X X. Feature selection techniques with class separability for multivariate time series. Neurocomputing, 2013, 110: 29−34 doi: 10.1016/j.neucom.2012.12.006 [3] He J Y, Shang P J. Comparison of transfer entropy methods for financial time series. Physica A: Statistical Mechanics and Its Applications, 2017, 482: 772−785 doi: 10.1016/j.physa.2017.04.089 [4] Baek S, Kim D Y. Empirical sensitivity analysis of discretization parameters for fault pattern extraction from multivariate time series data. IEEE Transactions on Cybernetics, 2017, 47(5): 1198−1209 doi: 10.1109/TCYB.2016.2540657 [5] 周平, 刘记平. 基于数据驱动多输出ARMAX建模的高炉十字测温中心温度在线估计. 自动化学报, 2018, 44(3): 552−561Zhou Ping, Liu Ji-Ping. Data-driven multi-output ARMAX modeling for online estimation of central temperatures for cross temperature measuring in blast furnace ironmaking. Acta Automatica Sinica, 2018, 44(3): 552−561 [6] Fu T C. A review on time series data mining. Engineering Applications of Artificial Intelligence, 2011, 24(1): 164−181 doi: 10.1016/j.engappai.2010.09.007 [7] Esling P, Agon C. Time-series data mining. ACM Computing Surveys, 2012, 45(1): 12 [8] 刘强, 秦泗钊. 过程工业大数据建模研究展望. 自动化学报, 2016, 42(2): 161−171Liu Qiang, Qin S Joe. Perspectives on big data modeling of process industries. Acta Automatica Sinica, 2016, 42(2): 161−171 [9] Hardoon D R, Szedmak S, Shawe-Taylor J. Canonical correlation analysis: An overview with application to learning methods. Neural Computation, 2004, 16(12): 2639−2664 doi: 10.1162/0899766042321814 [10] Han M, Ren W J. Global mutual information-based feature selection approach using single-objective and multi-objective optimization. Neurocomputing, 2015, 168: 47−54 doi: 10.1016/j.neucom.2015.06.016 [11] Reshef D N, Reshef Y A, Finucane H K, Grossman S R, McVean G, Turnbaugh P J, et al. Detecting novel associations in large data sets. Science, 2011, 334(6062): 1518−1524 doi: 10.1126/science.1205438 [12] Shi J, Ding Z H, Lee W J, Yang Y P, Liu Y Q, Zhang M M. Hybrid forecasting model for very-short term wind power forecasting based on grey relational analysis and wind speed distribution features. IEEE Transactions on Smart Grid, 2014, 5(1): 521−526 doi: 10.1109/TSG.2013.2283269 [13] Liebscher E. Copula-based dependence measures. Dependence Modeling, 2014, 2(1): 49−64 [14] Sun Y Q, Li J Y, Liu J X, Chow C W, Sun B Y, Wang R J. Using causal discovery for feature selection in multivariate numerical time series. Machine Learning, 2015, 101(1-3): 377−395 doi: 10.1007/s10994-014-5460-1 [15] Granger C W J. Investigating causal relations by econometric models and cross-spectral methods. Econometrica, 1969, 37(3): 424−438 doi: 10.2307/1912791 [16] Barnett L, Seth A K. The MVGC multivariate Granger causality toolbox: A new approach to Granger-causal inference. Journal of Neuroscience Methods, 2014, 223: 50−68 doi: 10.1016/j.jneumeth.2013.10.018 [17] Hlaváčková-Schindler K, Paluš M, Vejmelka M, Bhattacharya J. Causality detection based on information-theoretic approaches in time series analysis. Physics Reports, 2007, 441(1): 1−46 doi: 10.1016/j.physrep.2006.12.004 [18] Cummins B, Gedeon T, Spendlove K. On the efficacy of state space reconstruction methods in determining causality. SIAM Journal on Applied Dynamical Systems, 2015, 14(1): 335−381 doi: 10.1137/130946344 [19] Zou C L, Feng J F. Granger causality vs. dynamic Bayesian network inference: A comparative study. BMC Bioinformatics, 2009, 10(1): 122−122 doi: 10.1186/1471-2105-10-122 [20] Kleinberg S, Hripcsak G. A review of causal inference for biomedical informatics. Journal of Biomedical Informatics, 2011, 44(6): 1102−1112 doi: 10.1016/j.jbi.2011.07.001 [21] Porta A, Faes L. Wiener-Granger causality in network physiology with applications to cardiovascular control and neuroscience. Proceedings of the IEEE, 2016, 104(2): 282−309 doi: 10.1109/JPROC.2015.2476824 [22] Seth A K, Barrett A B, Barnett L. Granger causality analysis in neuroscience and neuroimaging. The Journal of Neuroscience, 2015, 35(8): 3293−3297 doi: 10.1523/JNEUROSCI.4399-14.2015 [23] Geweke J. Measurement of linear dependence and feedback between multiple time series. Journal of the American Statistical Association, 1982, 77(378): 304−313 doi: 10.1080/01621459.1982.10477803 [24] Chen Y H, Rangarajan G, Feng J F, Ding M Z. Analyzing multiple nonlinear time series with extended Granger causality. Physics Letters A, 2004, 324(1): 26−35 doi: 10.1016/j.physleta.2004.02.032 [25] Siggiridou E, Kugiumtzis D. Granger causality in multivariate time series using a time-ordered restricted vector autoregressive model. IEEE Transactions on Signal Processing, 2016, 64(7): 1759−1773 doi: 10.1109/TSP.2015.2500893 [26] Arnold A, Liu Y, Abe N. Temporal causal modeling with graphical granger methods. In: Proceedings of the 13th ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. San Jose, California, USA: ACM, 2007. 66−75 [27] Shojaie A, Michailidis G. Discovering graphical Granger causality using the truncating lasso penalty. Bioinformatics, 2010, 26(18): i517−i523 doi: 10.1093/bioinformatics/btq377 [28] Bolstad A, Van Veen B D, Nowak R. Causal network inference via group sparse regularization. IEEE Transactions on Signal Processing, 2011, 59(6): 2628−2641 doi: 10.1109/TSP.2011.2129515 [29] Yang G X, Wang L, Wang X F. Reconstruction of complex directional networks with group lasso nonlinear conditional Granger causality. Scientific Reports, 2017, 7(1): 2991 doi: 10.1038/s41598-017-02762-5 [30] Ancona N, Marinazzo D, Stramaglia S. Radial basis function approach to nonlinear Granger causality of time series. Physical Review E, 2004, 70(5): 056221 doi: 10.1103/PhysRevE.70.056221 [31] Marinazzo D, Pellicoro M, Stramaglia S. Kernel method for nonlinear Granger causality. Physical Review Letters, 2008, 100(14): 144103 doi: 10.1103/PhysRevLett.100.144103 [32] Marinazzo D, Pellicoro M, Stramaglia S. Kernel-Granger causality and the analysis of dynamical networks. Physical Review E, 2008, 77(5): 056215 doi: 10.1103/PhysRevE.77.056215 [33] Wu G R, Duan X J, Liao W, Gao Q, Chen H F. Kernel canonical-correlation Granger causality for multiple time series. Physical Review E, 2011, 83(4): 041921 doi: 10.1103/PhysRevE.83.041921 [34] Hu M, Liang H L. A copula approach to assessing Granger causality. NeuroImage, 2014, 100: 125−134 doi: 10.1016/j.neuroimage.2014.06.013 [35] Montalto A, Stramaglia S, Faes L, Tessitore G, Prevete R, Marinazzo D. Neural networks with non-uniform embedding and explicit validation phase to assess Granger causality. Neural Networks, 2015, 71: 159−171 doi: 10.1016/j.neunet.2015.08.003 [36] Baccalá L A, Sameshima K. Partial directed coherence: A new concept in neural structure determination. Biological Cybernetics, 2001, 84(6): 463−474 doi: 10.1007/PL00007990 [37] Kamiński M, Ding M Z, Truccolo W A, Bressler S L. Evaluating causal relations in neural systems: Granger causality, directed transfer function and statistical assessment of significance. Biological Cybernetics, 2001, 85(2): 145−157 doi: 10.1007/s004220000235 [38] Stokes P A, Purdon P L. A study of problems encountered in Granger causality analysis from a neuroscience perspective. Proceedings of the National Academy of Sciences, 2017, 114(34): E7063−E7072 doi: 10.1073/pnas.1704663114 [39] Barrett A B, Barnett L, Seth A K. Multivariate Granger causality and generalized variance. Physical Review E, 2010, 81(4): 041907 doi: 10.1103/PhysRevE.81.041907 [40] Schreiber T. Measuring information transfer. Physical Review Letters, 2000, 85(2): 461−464 doi: 10.1103/PhysRevLett.85.461 [41] Barnett L, Barrett A B, Seth A K. Granger causality and transfer entropy are equivalent for Gaussian variables. Physical Review Letters, 2009, 103(23): 238701 doi: 10.1103/PhysRevLett.103.238701 [42] Staniek M, Lehnertz K. Symbolic transfer entropy. Physical Review Letters, 2008, 100(15): 158101 doi: 10.1103/PhysRevLett.100.158101 [43] Kugiumtzis D. Partial transfer entropy on rank vectors. The European Physical Journal Special Topics, 2013, 222(2): 401−420 doi: 10.1140/epjst/e2013-01849-4 [44] Faes L, Nollo G, Porta A. Information-based detection of nonlinear Granger causality in multivariate processes via a nonuniform embedding technique. Physical Review E, 2011, 83(5): 051112 doi: 10.1103/PhysRevE.83.051112 [45] Frenzel S, Pompe B. Partial mutual information for coupling analysis of multivariate time series. Physical Review Letters, 2007, 99(20): 204101 doi: 10.1103/PhysRevLett.99.204101 [46] Kugiumtzis D. Direct-coupling information measure from nonuniform embedding. Physical Review E, 2013, 87(6): 062918 doi: 10.1103/PhysRevE.87.062918 [47] Vlachos I, Kugiumtzis D. Nonuniform state-space reconstruction and coupling detection. Physical Review E, 2010, 82(1): 016207 doi: 10.1103/PhysRevE.82.016207 [48] Runge J, Heitzig J, Petoukhov V, Kurths J. Escaping the curse of dimensionality in estimating multivariate transfer entropy. Physical Review Letters, 2012, 108(25): 258701 doi: 10.1103/PhysRevLett.108.258701 [49] Takens F. Detecting strange attractors in turbulence. Dynamical Systems and Turbulence. Heidelberg, Germany: Springer-Verlag, 1981. 366−381 [50] Kalman R E. A new approach to linear filtering and prediction problems. Journal of Basic Engineering, 1960, 82(1): 35−45 doi: 10.1115/1.3662552 [51] Solo V. State-space analysis of Granger-Geweke causality measures with application to fMRI. Neural Computation, 2016, 28(5): 914−949 doi: 10.1162/NECO_a_00828 [52] Jinno K, Xu S G, Berndtsson R, Kawamura A, Matsumoto M. Prediction of unspots using reconstructed chaotic system equations. Journal of Geophysical Research: Space Physics, 1995, 100(A8): 14773−14781 doi: 10.1029/95JA01167 [53] Hong M, Wang D, Wang Y K, Zeng X K, Ge S S, Yan H Q, Singh V P. Mid-and long-term runoff predictions by an improved phase-space reconstruction model. Environmental Research, 2016, 148: 560−573 doi: 10.1016/j.envres.2015.11.024 [54] 殷礼胜, 何怡刚, 董学平, 鲁照权. 交通流量VNNTF神经网络模型多步预测研究. 自动化学报, 2014, 40(9): 2066−2072Yin Li-Sheng, He Yi-Gang, Dong Xue-Ping, Lu Zhao-Quan. Research on the multi-step prediction of Volterra neural network for traffic flow. Acta Automatica Sinica, 2014, 40(9): 2066−2072 [55] Luo S H, Gao C H, Zeng J S, Huang J. Blast furnace system modeling by multivariate phase space reconstruction and neural networks. Asian Journal of Control, 2013, 15(2): 553−561 doi: 10.1002/asjc.574 [56] Cao L. Practical method for determining the minimum embedding dimension of a scalar time series. Physica D: Nonlinear Phenomena, 1997, 110(1): 43−50 [57] Molkov Y I, Mukhin D N, Loskutov E M, Feigin A M, Fidelin G A. Using the minimum description length principle for global reconstruction of dynamic systems from noisy time series. Physical Review E, 2009, 80(4): 046207 doi: 10.1103/PhysRevE.80.046207 [58] Kugiumtzis D. State space reconstruction parameters in the analysis of chaotic time series-the role of the time window length. Physica D: Nonlinear Phenomena, 1996, 95(1): 13−28 doi: 10.1016/0167-2789(96)00054-1 [59] Kim H, Eykholt R, Salas J D. Nonlinear dynamics, delay times, and embedding windows. Physica D: Nonlinear Phenomena, 1999, 127(1−2): 48−60 doi: 10.1016/S0167-2789(98)00240-1 [60] Shen M, Chen W N, Zhang J, Chung H S H, Kaynak O. Optimal selection of parameters for nonuniform embedding of chaotic time series using ant colony optimization. IEEE Transactions on Cybernetics, 2013, 43(2): 790−802 doi: 10.1109/TSMCB.2012.2219859 [61] Arnhold J, Grassberger P, Lehnertz K, Elger C E. A robust method for detecting interdependences: application to intracranially recorded EEG. Physica D: Nonlinear Phenomena, 1999, 134(4): 419−430 doi: 10.1016/S0167-2789(99)00140-2 [62] Quiroga R Q, Arnhold J, Grassberger P. Learning driver-response relationships from synchronization patterns. Physical Review E, 2000, 61(5): 5142 doi: 10.1103/PhysRevE.61.5142 [63] Andrzejak R G, Kraskov A, Stögbauer H, Mormann F, Kreuz T. Bivariate surrogate techniques: Necessity, strengths, and caveats. Physical Review E, 2003, 68(6): 066202 doi: 10.1103/PhysRevE.68.066202 [64] Chicharro D, Andrzejak R G. Reliable detection of directional couplings using rank statistics. Physical Review E, 2009, 80(2): 026217 doi: 10.1103/PhysRevE.80.026217 [65] Sugihara G, May R, Ye H, Hsieh C H, Deyle E, Fogarty M, Munch S. Detecting causality in complex ecosystems. Science, 2012, 338(6106): 496−500 doi: 10.1126/science.1227079 [66] Schäck T, Muma M, Feng M L, Guan C T, Zoubir A M. Robust nonlinear causality analysis of nonstationary multivariate physiological time series. IEEE Transactions on Biomedical Engineering, 2017, 65(6): 1213−1225 [67] Montalto A, Faes L, Marinazzo D. MuTE: A MATLAB toolbox to compare established and novel estimators of the multivariate transfer entropy. PloS One, 2014, 9(10): e109462 doi: 10.1371/journal.pone.0109462 [68] Ma H F, Aihara K, Chen L N. Detecting causality from nonlinear dynamics with short-term time series. Scientific Reports, 2014, 4: 7464 [69] Clark A T, Ye H, Isbell F, Deyle E R, Cowles J, Tilman G D, Sugihara G. Spatial convergent cross mapping to detect causal relationships from short time. Ecology, 2015, 96(5): 1174−1181 doi: 10.1890/14-1479.1 [70] Mønster D, Fusaroli R, Tylén K, Roepstorff A, Sherson J F. Causal inference from noisy time-series data—testing the convergent cross-mapping algorithm in the presence of noise and external influence. Future Generation Computer Systems, 2017, 73: 52−62 doi: 10.1016/j.future.2016.12.009 [71] Zhu J Y, Zhang C, Zhang H C, Zhi S, Li V O K, Han J W, Zheng Y. pg-Causality: Identifying spatiotemporal causal pathways for air pollutants with urban big data. IEEE Transactions on Big Data, 2018, 4(4): 571−585 doi: 10.1109/TBDATA.2017.2723899 [72] Liang X S. Unraveling the cause-effect relation between time series. Physical Review E, 2014, 90(5): 052150 doi: 10.1103/PhysRevE.90.052150 [73] Faybishenko B. Detecting dynamic causal inference in nonlinear two-phase fracture flow. Advances in Water Resources, 2017, 106: 111−120 doi: 10.1016/j.advwatres.2017.02.011 [74] Zhu J Y, Sun C, Li V O K. An extended spatio-temporal Granger causality model for air quality estimation with heterogeneous urban big data. IEEE Transactions on Big Data, 2017, 3(3): 307−319 doi: 10.1109/TBDATA.2017.2651898 [75] Chen Z Y, Cai J, Gao B B, Xu B, Dai S, He B, Xie X M. Detecting the causality influence of individual meteorological factors on local PM 2.5 concentration in the Jing-Jin-Ji region. Scientific Reports, 2017, 7: 40735 doi: 10.1038/srep40735 [76] Hu S Q, Dai G J, Worrell G A, Dai Q H, Liang H L. Causality analysis of neural connectivity: Critical examination of existing methods and advances of new methods. IEEE Transactions on Neural Networks, 2011, 22(6): 829−844 doi: 10.1109/TNN.2011.2123917 [77] Dhamala M, Rangarajan G, Ding M Z. Analyzing information flow in brain networks with nonparametric Granger causality. NeuroImage, 2008, 41(2): 354−362 doi: 10.1016/j.neuroimage.2008.02.020 [78] Wu G R, Chen F Y, Kang D Z, Zhang X Y, Marinazzo D, Chen H F. Multiscale causal connectivity analysis by canonical correlation: Theory and application to epileptic brain. IEEE Transactions on Biomedical Engineering, 2011, 58(11): 3088−3096 doi: 10.1109/TBME.2011.2162669 [79] Li P Y, Huang X Y, Li F L, Wang X R, Zhou W W, Liu H, et al. Robust Granger analysis in Lp norm space for directed EEG network analysis. IEEE Transactions on Neural Systems and Rehabilitation Engineering, 2017, 25(11): 1959−1969 doi: 10.1109/TNSRE.2017.2711264 [80] Hu M, Li W, Liang H L. A copula-based Granger causality measure for the analysis of neural spike train data. IEEE/ACM Transactions on Computational Biology and Bioinformatics, 2018, 15(2): 562−569 doi: 10.1109/TCBB.2014.2388311 [81] Faes L, Marinazzo D, Montalto A, Nollo G. Lag-specific transfer entropy as a tool to assess cardiovascular and cardiorespiratory information transfer. IEEE Transactions on Biomedical Engineering, 2014, 61(10): 2556−2568 doi: 10.1109/TBME.2014.2323131 [82] Wang Z, Alahmadi A, Zhu D C, Li T T. Causality analysis of fMRI data based on the directed information theory framework. IEEE Transactions on Biomedical Engineering, 2016, 63(5): 1002−1015 doi: 10.1109/TBME.2015.2481723 [83] Heskamp L, Meel-van den Abeelen A S, Lagro J, Claassen J A. Convergent cross mapping: A promising technique for cerebral autoregulation estimation. International Journal of Clinical Neurosciences and Mental Health, 2014, 1(1): S20 [84] Wang S, Li Q, Fang C, Zhou C. The relationship between economic growth, energy consumption, and CO2 emissions: Empirical evidence from China. Science of the Total Environment, 2016, 542: 360−371 doi: 10.1016/j.scitotenv.2015.10.027 [85] Zhou C S, Wang S J, Feng K S. Examining the socioeconomic determinants of CO2 emissions in China: A historical and prospective analysis. Resources, Conservation and Recycling, 2018, 130: 1−11 doi: 10.1016/j.resconrec.2017.11.007 [86] Rafindadi A A, Ozturk I. Impacts of renewable energy consumption on the German economic growth: Evidence from combined cointegration test. Renewable and Sustainable Energy Reviews, 2017, 75: 1130−1141 doi: 10.1016/j.rser.2016.11.093 [87] Tiwari A K. Causality between wholesale price and consumer price indices in India: An empirical investigation in the frequency domain. Indian Growth and Development Review, 2012, 5(2): 151−172 doi: 10.1108/17538251211268071 [88] Bekiros S, Nguyen D K, Junior L S, Uddin G S. Information diffusion, cluster formation and entropy-based network dynamics in equity and commodity markets. European Journal of Operational Research, 2017, 256(3): 945−961 doi: 10.1016/j.ejor.2016.06.052 [89] Papana A, Kyrtsou C, Kugiumtzis D, Diks C. Detecting causality in non-stationary time series using partial symbolic transfer entropy: evidence in financial data. Computational Economics, 2016, 47(3): 341−365 doi: 10.1007/s10614-015-9491-x -




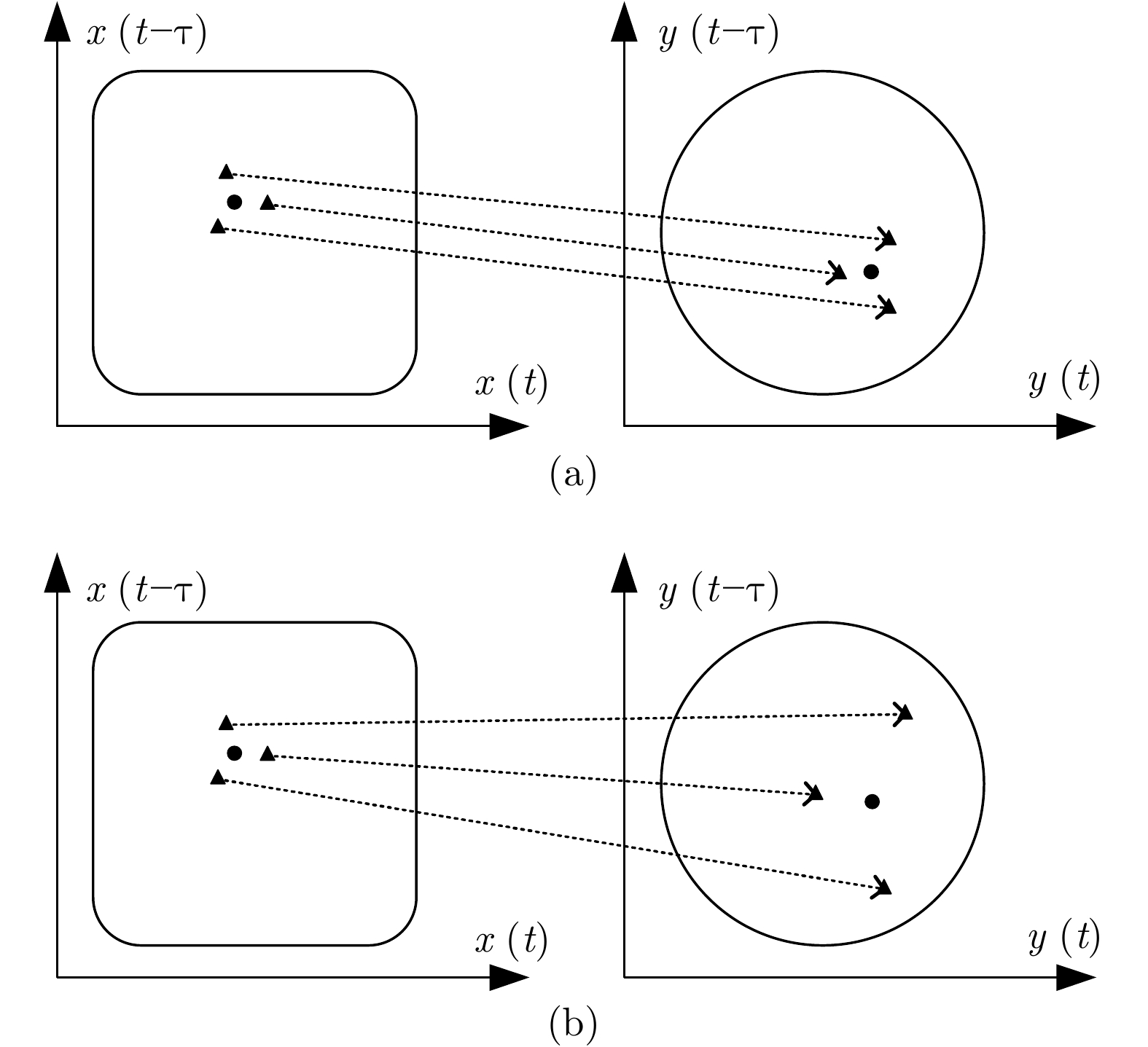
 下载:
下载:

