Combining Concatenated Random Forests and Active Contour for the 3D MR Images Segmentation
-
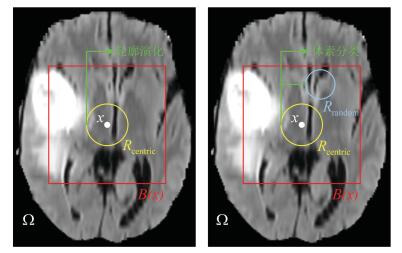
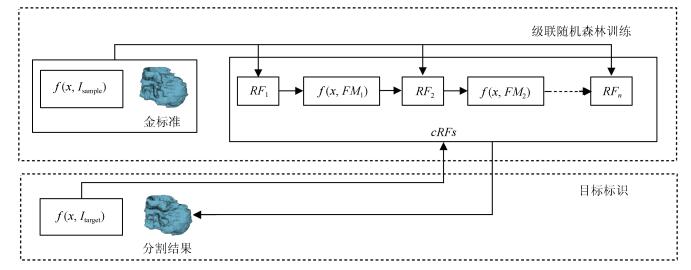
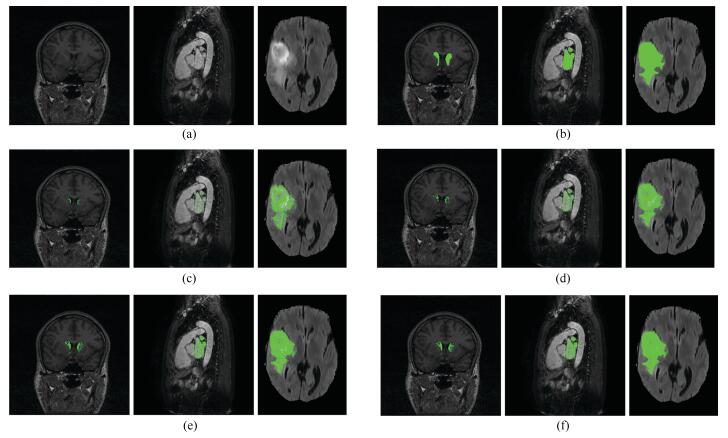
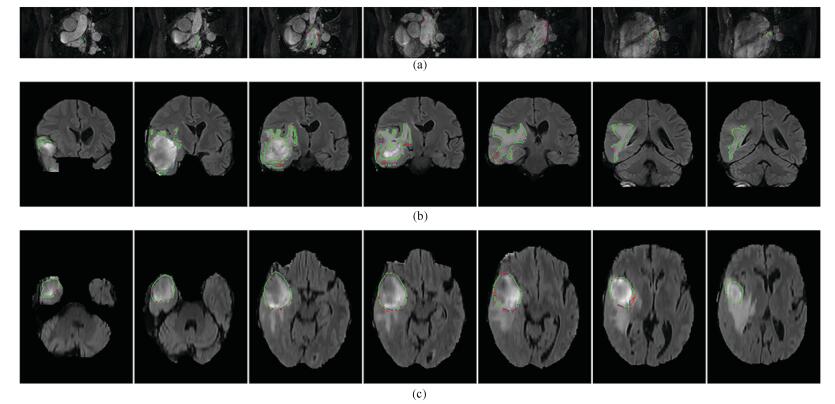
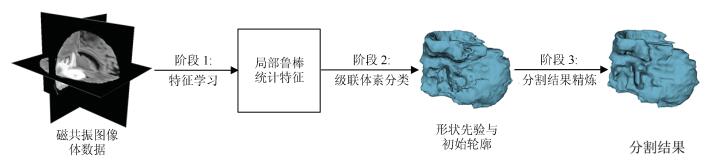
摘要: 针对医学磁共振(Magnetic resonance,MR)图像三维分割中随机森林(Random forest,RF)方法难以获得具有几何约束的结果以及活动轮廓模型(Active contour model,ACM)不能自动分割发生信号混叠的组织结构的问题,提出了一种整合了级联随机森林与活动轮廓模型的磁共振图像三维分割方法.该方法首先从多模态磁共振体数据中提取图像多尺度局部鲁棒统计信息,以此驱动级联随机森林对磁共振图像进行迭代的体素分类,从而获得对组织结构的初步分割结果,进一步将此结果作为初始轮廓与形状先验,整合进一个尺度可调的活动轮廓模型中,将独立的体素分类转化为轮廓曲线演化,最终得到具有几何约束的精确分割结果.在公开数据集上的实验结果表明,本文的自动化分割方法在分割精度和鲁棒性等方面,相比其他同类方法具有较大的性能提升.Abstract: Since it is difficult for the random forest (RF) method to achieve geometrically constrained result and the active contour model (ACM) can not segment tissue structures with overlapped signals automatically, when segmenting medical magnetic resonance (MR) images in three dimensions, a combined concatenated random forests and active contour model approach is proposed in this work for the 3D segmentation of medical magnetic resonance images. The multiscale local robust statistics image information is extracted from the multimodal magnetic resonance volumetric data, and then is used to drive the random forest to perform voxel classification iteratively. As a consequence, the initial segmentation result for the tissue structure is achieved. Furthermore, the initial result is integrated into a scale scalable active contour model as the initial contour and shape prior. In this way, the independent voxel classification is reformulated as contour evolution, and the final accurate and geometrically constrained segmentation result is achieved. Experimental results on publicly available datasets demonstrate that, compared to several related methods, the proposed automated segmentation method has considerable improvement in terms of segmentation accuracy and robustness.
-
Key words:
- Medical image segmentation /
- random forest (RF) /
- active contour model /
- 3D segmentation /
- shape prior
1) 本文责任编委 张道强 -
表 1 不同分割方法在左心房数据集上的DC系数对比
Table 1 Dice coefficients (DC) of different methods on the left atrial dataset
数据集 Proposed1 Proposed2 ACM RF Competitive contours ${\rm{M^3AS}}$ CRF CRF-AC 训练集 0.78$\, \pm\, $0.006 0.94$\, \pm\, $0.04 0.72$\, \pm\, $0.20 0.69$\, \pm\, $0.12 --- --- --- --- 测试集 0.74$\, \pm\, $0.07 0.90$\, \pm\, $0.03 0.71$\, \pm\, $0.20 0.63$\, \pm\, $0.14 0.92 0.88$\, \pm\, $0.06 0.74$\, \pm\, $0.07 0.93$\, \pm\, $0.05 表 2 不同方法在BRATS15测试集上的对比
Table 2 Comparison of different methods on the BRATS15 test set
Methods DC PPV Sensitivity Complete Core Enh Complete Core Enh Complete Core Enh Proposed 2 0.87 0.74 0.64 0.85 0.81 0.61 0.91 0.74 0.73 ${\rm{I_{NPUT}C_{ASCADE}CNN}}$ 0.88 0.79 0.73 --- --- --- 0.87 0.79 0.80 CNNs 0.78 0.65 0.75 --- --- --- --- --- FCNNs-CRFs 0.84 0.73 0.62 0.89 0.76 0.63 0.82 0.76 0.67 DeepMedic 0.85 0.67 0.63 0.85 0.85 0.63 0.88 0.61 0.66 -
[1] Peng P, Lekadir K, Gooya A, Shao L, Petersen S E, Frangi A F. A review of heart chamber segmentation for structural and functional analysis using cardiac magnetic resonance imaging. Magnetic Resonance Materials in Physics, Biology and Medicine, 2016, 29(2):155-195 doi: 10.1007/s10334-015-0521-4 [2] García-Lorenzo D, Francis S, Narayanan S, Arnold D L, Collins D L. Review of automatic segmentation methods of multiple sclerosis white matter lesions on conventional magnetic resonance imaging. Medical Image Analysis, 2013, 17(1):1-18 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=1737353876083cb89dc7f525b401d114 [3] Kamnitsas K, Ledig C, Newcombe V F J, Simpson J P, Kane A D, Menon D K, et al. Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation. Medical Image Analysis, 2016, 36:61-78 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=3433800aff8722882a11ce5ba7c75de7 [4] Menze B H, Jakab A, Bauer S, Kalpathy-Cramer J, Farahani K, Kirby J, et al. The multimodal brain tumor image segmentation benchmark (BRATS). IEEE Transactions on Medical Imaging, 2015, 34(10):1993-2024 doi: 10.1109/TMI.2014.2377694 [5] Wang K Q, Ma C. A robust statistics driven volume-scalable active contour for segmenting anatomical structures in volumetric medical images with complex conditions. Biomedical Engineering Online, 2016, 15: Article No.39 [6] Ma C, Luo G N, Wang K Q. A combined random forests and active contour model approach for fully automatic segmentation of the left atrium in volumetric MRI. BioMed Research International, 2017, 2017: Article No.8381094 [7] 郑强, 董恩清.窄带主动轮廓模型及在医学和纹理图像局部分割中的应用.自动化学报, 2014, 39(1):21-30 http://www.aas.net.cn/CN/abstract/abstract17809.shtmlZheng Qiang, Dong En-Qing. Narrow band active contour model for local segmentation of medical and texture images. Acta Automatica Sinica, 2014, 39(1):21-30 http://www.aas.net.cn/CN/abstract/abstract17809.shtml [8] Breiman L. Random forests. Machine Learning, 2001, 45(1):5-32 http://d.old.wanfangdata.com.cn/Periodical/gtzyyg201404010 [9] Criminisi A, Criminisi J, Criminisi E. Decision forests:a unified framework for classification, regression, density estimation, manifold learning and semi-supervised learning. Foundations and Trend in Computer Graphics and Vision, 2012, 7(2-3):81-227 https://dl.acm.org/citation.cfm?id=2185838 [10] Tustison N J, Shrinidhi K L, Wintermark M, Durst C R, Kandel B M, Gee J C, et al. Optimal symmetric multimodal templates and concatenated random forests for supervised brain tumor segmentation (Simplified) with ANTsR. Neuroinformatics, 2015, 13(2):209-225 doi: 10.1007/s12021-014-9245-2 [11] Petitjean C, Zuluaga M A, Bai W J, Dacher J N, Grosgeorge D, Caudron J, et al. Right ventricle segmentation from cardiac MRI:a collation study. Medical Image Analysis, 2015, 19(1):187-202 doi: 10.1016/j.media.2014.10.004 [12] Schneider M, Hirsch S, Weber B, Székely G, Menze B H. Joint 3-D vessel segmentation and centerline extraction using oblique Hough forests with steerable filters. Medical Image Analysis, 2015, 19(1):220-249 doi: 10.1016/j.media.2014.09.007 [13] Chao J, Shi F, Xiang D H, Jiang X Q, Zhang B, Wang X M, et al. 3D fast automatic segmentation of kidney based on modified AAM and random forest. IEEE Transactions on Medical Imaging, 2016, 35(6):1395-1407 doi: 10.1109/TMI.2015.2512606 [14] Krizhevsky A, Sutskever I, Hinton G E. ImageNet classification with deep convolutional neural networks. In: Proceedings of the 25th International Conference on Neural Information Processing Systems. Lake Tahoe, Nevada: Curran Associates Inc., 2012. 1097-1105 [15] Wang L, Gao Y Z, Shi F, Li G, Gilmore J H, Lin W L, et al. LINKS:learning-based multi-source IntegratioN frameworK for Segmentation of infant brain images. NeuroImage, 2015, 108:160-172 doi: 10.1016/j.neuroimage.2014.12.042 [16] Wang L, He L, Mishra A, Li C M. Active contours driven by local Gaussian distribution fitting energy. Signal Processing, 2009, 89(12):2435-2447 doi: 10.1016/j.sigpro.2009.03.014 [17] Avendi M R, Kheradvar A, Jafarkhani H. A combined deep-learning and deformable-model approach to fully automatic segmentation of the left ventricle in cardiac MRI. Medical Image Analysis, 2016, 30:108-119 doi: 10.1016/j.media.2016.01.005 [18] Ngo T A, Lu Z, Carneiro G. Combining deep learning and level set for the automated segmentation of the left ventricle of the heart from cardiac cine magnetic resonance. Medical Image Analysis, 2017, 35:159-171 doi: 10.1016/j.media.2016.05.009 [19] Hoogi A, Beaulieu C F, Cunha G M, Heba E, Sirlin C B, Napel S, et al. Adaptive local window for level set segmentation of CT and MRI liver lesions. Medical Image Analysis, 2017, 37:46-55 doi: 10.1016/j.media.2017.01.002 [20] Wang Y, Seguro F, Kao E, Zhang Y, Faraji F, Zhu C C, et al. Segmentation of lumen and outer wall of abdominal aortic aneurysms from 3D black-blood MRI with a registration based geodesic active contour model. Medical Image Analysis, 2017, 40:1-10 doi: 10.1016/j.media.2017.05.005 [21] 孙文燕, 董恩清, 曹祝楼, 郑强.一种基于模糊主动轮廓的鲁棒局部分割方法.自动化学报, 2017, 43(4):611-621 http://www.aas.net.cn/CN/abstract/abstract19039.shtmlSun Wen-Yan, Dong En-Qing, Cao Zhu-Lou, Zheng Qiang. A robust local segmentation method based on fuzzy-energy based active contour. Acta Automatica Sinica, 2017, 43(4):611-621 http://www.aas.net.cn/CN/abstract/abstract19039.shtml [22] Wang X F, Min H, Zou L, Zhang Y G. A novel level set method for image segmentation by incorporating local statistical analysis and global similarity measurement. Pattern Recognition, 2015, 48(1):189-204 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=bbc49485755fad49fe894495ee66ecd0 [23] Wright J, Ma Y, Mairal J, Sapiro G, Huang T S, Yan S C. Sparse representation for computer vision and pattern recognition. Proceedings of the IEEE, 2010, 98(6):1031-1044 doi: 10.1109/JPROC.2010.2044470 [24] Srivastava N, Hinton G, Krizhevsky A, Sutskever, Salakhutdinov R. Dropout:a simple way to prevent neural networks from overfitting. The Journal of Machine Learning Research, 2014, 15(1):1929-1958 http://d.old.wanfangdata.com.cn/Periodical/kzyjc200606005 [25] Wang L, Shi F, Lin W L, Gilmore J H, Shen D G. Automatic segmentation of neonatal images using convex optimization and coupled level sets. NeuroImage, 2011, 58(3):805-817 doi: 10.1016/j.neuroimage.2011.06.064 [26] Chan T F, Vese L A. Active contours without edges. IEEE Transactions on Image Processing, 2001, 10(2):266-277 doi: 10.1109/83.902291 [27] Van Ginneken B, Heimann T, Styner M. 3D segmentation in the clinic: a grand challenge. In: Proceedings of the 2007 International Conference on Medical Image Computing and Computer Assisted Intervention. The Netherlands: MICCAI, 2007. 7-15 [28] Tobon-Gomez C, Geers A J, Peters J, Weese J, Pinto K, Karim R, et al. Benchmark for algorithms segmenting the left atrium from 3D CT and MRI datasets. IEEE Transactions on Medical Imaging, 2015, 34(7):1460-1473 doi: 10.1109/TMI.2015.2398818 [29] Li C M, Kao C Y, Gore J C, Ding Z H. Minimization of region-scalable fitting energy for image segmentation. IEEE Transactions on Image Processing, 2008, 17(10):1940-1949 doi: 10.1109/TIP.2008.2002304 [30] Institute SCaI. "Seg3D" volumetric image segmentation and visualization. Scientific Computing and Imaging Institute (SCI).[Online], available: http://www.seg3d.org, May 20, 2017 [31] Margeta J, McLeod K, Criminisi A, Ayache N. Decision forests for segmentation of the left atrium from 3D MRI. In: Proceedings of the 4th International Workshop on Statistical Atlases and Computational Models of the Heart. Nagoya, Japan: Springer, 2014. 49-56 [32] Morais P, Vilaça J L, Queirós S, Bourier F, Deisenhofer I, Tavares J M R S, et al. A competitive strategy for atrial and aortic tract segmentation based on deformable models. Medical Image Analysis, 2017, 42:102-116 doi: 10.1016/j.media.2017.07.007 [33] Zhuang X H, Shen J. Multi-scale patch and multi-modality atlases for whole heart segmentation of MRI. Medical Image Analysis, 2016, 31:77-87 doi: 10.1016/j.media.2016.02.006 [34] Zhao X M, Wu Y H, Song G D, Li Z, Zhang Y Y, Fan Y. A deep learning model integrating FCNNs and CRFs for brain tumor segmentation. Medical Image Analysis, 2018, 43:98-111 doi: 10.1016/j.media.2017.10.002 [35] Havaei M, Davy A, Warde-Farley D, Biard A, Courville A, Bengio Y, et al. Brain tumor segmentation with Deep Neural Networks. Medical Image Analysis, 2017, 35:18-31 doi: 10.1016/j.media.2016.05.004 [36] Pereira S, Pinto A, Alves V, Silva C A. Brain tumor segmentation using convolutional neural networks in MRI images. IEEE Transactions on Medical Imaging, 2016, 35(5):1240-1251 doi: 10.1109/TMI.2016.2538465 -




 下载:
下载: