Discriminative Feature-oriented Dictionary Learning Method With Fisher Criterion for Histopathological Image Classification
-
摘要: 针对当前面向组织病理图像特征提取的字典学习方法中存在着学习的无病字典与有病字典相似程度高,判别性弱的问题,本文提出一种新的面向判别性特征字典学习方法(Discriminative feature-oriented dictionary learning based on Fisher criterion,FCDFDL).该方法基于Fisher准则构造目标函数的惩罚项,最小化学习字典的类内距离与最大化学习字典的类间距离,大大降低无病字典与有病字典间的相似性.同时,优化学习字典对同类样本的重构性能,并约束学习字典对非同类样本的重构性能.然后,利用本文学习的无病与有病字典对测试样本进行稀疏表示,采用重构误差向量的统计量构造分类器.最后,分别在ADL数据集与BreaKHis数据集上验证了本文方法的有效性.实验结果表明,本文学习字典的判别性更强,获得了更优的分类性能.Abstract: The problem of high similarity between learned healthy dictionary and diseased dictionary and low discrimination exists in the current dictionary learning methods for histopathological image feature extraction. In this paper, we present a novel discriminative feature-oriented dictionary learning method based on Fisher criterion (FCDFDL). This method constructs a penalty item of the objective function using Fisher criterion to minimize the intra-class distance of learned dictionaries and maximize the inter-class distance of learned dictionaries. Thus, the similarity between healthy and diseased dictionaries is reduced. Furthermore, the reconstruction of the same class samples is improved over the learned dictionaries, while reconstruction of different class samples is suppressed. Then, the sparse representation of test samples is respectively performed on the learned healthy dictionary and the diseased dictionary, and the classifier is constructed by employing the reconstruction error vector of test samples. Finally, the proposed FCDFDL is tested on ADL and BreaKHis datasets, and experimental results show that the learned dictionaries have stronger discrimination and improved classification performance as compared to the other dictionary learning methods, for histopathological image.
-
Key words:
- Histopathological image /
- Fisher criterion /
- dictionary learning /
- discriminative feature
-
组织病理图像包含大量复杂的病理信息, 具有丰富的空间几何结构, 细胞类型多样且形态各异.目前, 组织病理图像的分析, 主要依赖具有临床经验的病理学家寻找图像中的病理特征.随着远程治疗与精准治疗的提出, 组织病理图像数量呈指数级增长, 极大地增加了工作量.近几年来, 机器学习与计算机辅助诊断技术(Computer aided diagnosis, CAD)得到了迅速的发展, 自动提取组织病理图像中的判别性特征, 辅助疾病诊断, 已成为研究热点, 并迅速引起国内外学者的关注[1-3].
病理图像的特征提取与分类是组织病理图像CAD系统的关键环节, 对疾病诊断有着极其重要的作用.为此诸多学者提出了很多解决办法, 主要分为两大类. 1)基于像素级特征的分类.如细胞的大小与形态特征[1, 4]、图像的灰度或彩色信息[5]、纹理特征[5-9]等. Tabesh等[5]提取了前列腺癌病理图像的颜色、纹理和形态学特征, 并基于监督学习框架进行特征组合, 然后对比了K-NN、支持向量机(Support vector machine, SVM)等分类方法的性能. Doyle等[6]尝试采用纹理和细胞核结构特征构造特征集, 并采用SVM实现了乳腺癌患病等级的鉴定. Li等[7]结合随机投影、局部二值模式(Local binary patterns, LBP)与独立子空间分析, 提取了直肠息肉的三维纹理特征来鉴定疾病等级. Linder等[8]提取了肿瘤上皮与基质组织的LBP、LBP/C特征, 并采用SVM进行分类. 2)基于空间结构与多尺度特征的分类.如尺度不变特征(Scale invariant feature, SIFT)[10]、小波特征[11]等; Irshad等[10]比较了纹理特征、SIFT特征、多级最大化模型(Hierarchical MAX, HMAX)特征对组织病理图像分类性能的影响. Ergin等[11]提取了方向梯度直方图(Histogram of oriented gradients, HOG)、稠密尺度不变特征(Dense scale invariant feature, DSIFT)与局部结构特征, 并应用于乳腺癌组织病理图像的分类.上述方法提取的均为手工特征, 特征冗余度高, 且较适合特定图像集的分类问题, 应用范围受到一定的限制.
近年来, Wright等[12]提出了基于稀疏表示的分类方法, 并在组织病理图像[13-22]、语音信号[23]、SAR图像[24]、人脸图像[25-27]和图像超分辨算法[28]等领域得到了广泛应用. Srinivas等[13-14]提出一种同步稀疏模型, 将组织病理图像中训练样本RGB三通道值作为字典, 并利用测试样本的稀疏重构误差进行分类. Nayak等[15]提出了一种带稀疏约束的受限玻尔兹曼机(Restricted Boltzmann machine, RBM)模型, 实现肿瘤组织病理图像的特征提取及分类. Chang等[16-17]提出一种基于堆叠预测稀疏分解的字典学习方法, 利用空间金字塔匹配(Spatial pyramid matching, SPM)方法对稀疏表示系数进行编码, 并采用SVM实现了肿瘤的病理状态分类. Shi等[18]提出了一种基于联合稀疏编码的空间金字塔匹配方法, 该方法利用RGB三个颜色通道信息, 通过联合稀疏编码将灰度描述算子转化为彩色描述算子, 提高了组织病理图像分类性能. Zhou等[19]提出一种面向组织病理图像的多光谱特征学习模型, 该模型基于卷积稀疏编码自动学习一组卷积滤波算子, 利用学习的滤波算子提取多通道的光谱特征, 并采用SVM进行分类. Shi等[20]基于多模式稀疏表示提出了一种肺部组织病理图像的分类方法(Multimodal sparse representation-based classification, mSRC), 该方法利用遗传算法引导了颜色、形状和纹理三个子字典的学习, 然后结合稀疏重构误差和多数投票算法对肺部组织病理图像进行分类. Xu等[21]基于堆栈式稀疏自编码器(Stacked sparse autoencoder, SSAE)进行乳腺癌组织病理图像的特征提取, 并利用Softmax实现了组织病理图像中细胞核的检测. Zhang等[22]基于图方法实现了具有细胞核图像的全局与局部特征的融合, 然后结合排序与多数投票算法对乳腺癌组织病理图像进行分类, 并取得较好的效果.
上述方法引入图像的稀疏性可以有效提取图像特征, 均属于无监督方式, 提取的特征具有较好的重构性, 但并不一定具有较好的判别性. Zhang等[25]利用监督学习思想, 提出了一种判别性K-SVD (Discriminative K-SVD, DK-SVD)字典学习方法, 该方法主要通过优化分类器参数来提升字典的判别性. Jiang等[26]提出了基于类标一致K-SVD (Label consistent K-SVD, LC-KSVD)的字典学习方法, 通过引入样本类标信息, 增加稀疏表示系数的判别性. Yang[27]提出一种Fisher判别字典学习(Fisher discrimination dictionary learning, FDDL)方法, 该方法通过稀疏表示系数的Fisher准则约束来提高分类性能.上述文献主要通过约束分类器参数或者稀疏表示系数来间接提升字典的判别性能.
最近, Vu等[29]提出了一种面向判别性特征的字典学习(Discriminative feature-oriented dictionary learning, DFDL)方法, 并应用于组织病理图像分类. DFDL方法引入了训练样本的类标信息, 直接学习无病字典与有病的字典, 并取得一定的分类性能.但是, 组织病理图像空间几何结构丰富, 细胞类型多样, 同类图像中细胞形态与几何结构变化可能较大, 非同类图像中细胞却存在一定的相似性, 导致类内图像特征间的距离有可能大于类间图像特征间的距离.因此, DFDL方法所学习的有病字典与无病字典相似程度较高, 对无病样本与有病样本的判别性仍然较低, 分类性能依然有待于提高
本文基于Fisher准则, 提出了一种新的面向判别性特征的字典学习方法(Discriminative feature-oriented dictionary learning based on Fisher criterion, FCDFDL), 并应用于组织病理图像分类.
1. DFDL方法
Vu等[29]于2015年提出了一种面向判别性特征的字典学习方法(Discriminative feature-oriented dictionary learning, DFDL), 并应用于医学组织病理图像分类.其目标函数定义如下:
$ \left\langle {D^*, X_D^*, \bar X_D^* } \right\rangle =\notag\\ \qquad \underset{D,{{X}_{D}},{{{\bar{X}}}_{D}}}{\mathop{\arg \min }}\, \bigg( \frac{1}{N}\left\| {Y - DX_D } \right\|_F^2 \, - \notag\\ \qquad \frac{\rho }{{\bar N}}\left\| {\bar Y - D\bar X_D } \right\|_F^2 \bigg)\nonumber \\\, {\rm s.\, t.}~ \left\| {X_D } \right\|_0 < L_1 ~{\rm and}~\left\| {\bar X_D } \right\|_0 < L_1 $
(1) $ \left\langle {\bar D^*, \bar X_{\bar D}^*, X_{\bar D}^* } \right\rangle =\notag\\ \qquad \underset{\bar{D},{{{\bar{X}}}_{{\bar{D}}}},{{X}_{{\bar{D}}}}}{\mathop{\arg \min }}\, \bigg( \frac{1}{{\bar N}}\left\| {\bar Y - \bar D\bar X_{\bar D} } \right\|_F^2\, -\nonumber\\ \qquad \frac{\rho }{N}\left\| {Y - \bar DX_{\bar D} } \right\|_F^2 \bigg)\nonumber\\ \, {\rm s.\, t.}~ \left\| {\bar X_{\bar D} } \right\|_0 < L_2 ~{\rm and}~\left\| {X_{\bar D} } \right\|_0 < L_2 $
(2) 其中, $Y$和$\bar Y$分别代表无病与有病的训练样本, $D$和$\bar D$分别代表无病与有病的字典, 在本文中统称字典. ${{X}_{D}}$和${{\bar{X}}_{D}}$分别代表无病与有病样本在$D$下的稀疏表示系数, ${{X}_{{\bar{D}}}}$和${{\bar{X}}_{{\bar{D}}}}$分别代表无病与有病样本在$\bar D$下的稀疏表示系数. $N$和$\bar N$分别代表$Y$和$\bar Y$的样本个数, ${{L}_{1}}$, ${{L}_{2}}$为稀疏度, $\rho $为正则化参数, 且$\rho >0$.
式(1)和式(2)中, 第1项都表示学习字典对同类样本的稀疏重构误差, 第2项都表示学习字典对非同类样本的稀疏重构误差.通过最小化第1项并最大化第2项, 可以直接学习无病字典与有病字典. DFDL方法在学习过程中没有考虑无病字典$D$与有病字典$\bar D$之间的差异, 导致所学习的$D$与$\bar D$之间相似程度高, 对无病样本与有病样本的稀疏表示系数判别性仍然较低, 影响了组织病理图像的分类与疾病诊断性能.
2. FCDFDL方法
2.1 Fisher准则
假设$D$, $\bar{D}\in {{\bf R}^{n\times k}}$, $D=[{{d}_{1}}, {{d}_{2}}, \cdots, {{d}_{i}}, \cdots$, ${{d}_{k}}]$, $\bar{D}=[{{{\bar{d}}}_{1}}, {{{\bar{d}}}_{2}}, \cdots, {{{\bar{d}}}_{i}}, \cdots, {{{\bar{d}}}_{k}}]$.其中, $k$为字典原子的个数, 本文以无病字典$D$为例, 字典的类内距离${{S}_{W}}(D)$和类间距离${{S}_{B}}(D)$分别定义如下[27]:
$ {{S}_{W}}(D)=\sum\limits_{{{d}_{i}}\in D}{\left( {{d}_{i}}-m \right)}{{\left( {{d}_{i}}-m \right)}^{\rm T}} $
(3) $ {{S}_{B}}(D)=\left( m-\bar{m} \right){{\left( m-\bar{m} \right)}^{\rm T}} $
(4) 其中, ${{d}_{i}}$是无病字典$D$中第$i$个原子, $m$是无病字典$D$中所有原子的均值, $\bar{m}$是有病字典$\bar{D}$中所有原子的均值.在字典学习阶段要保证无病字典$D$的类内距离更小, 同时要保持与有病字典$\bar{D}$之间的距离更大, 结合Fisher准则, 本文构造的无病字典$D$惩罚项定义如下:
$ \begin{align}\label{formual 5} \Psi ( D ) =&\ \dfrac{1}{N}\left( {\alpha \times{\rm tr}\left( {S_W ( D )} \right) - \beta \times{\rm tr}\left( {S_B \left( D \right)} \right)} \right)=\notag \\[2mm] &\ \dfrac{1}{N}\left( {\alpha \times\left\| {D - M} \right\|_F^2 - \beta \times\left\| {m - \bar m} \right\|_F^2 } \right) \end{align} $
(5) 其中, 矩阵$M$中列向量均为$m$, tr表示矩阵的迹.
类似地, 为保证有病字典$\bar{D}$的类内距离更小, 同时保持与无病字典$D$之间的距离更大.因此, 有病字典$\bar{D}$的惩罚项可定义为
$ \begin{align}\label{formual 6} \Psi ( {\bar D}) =&\ \dfrac{1}{{\bar N}}\Big( \alpha \times{\rm tr}\left( {S_W \left( {\bar D} \right)} \right) - \nonumber\\ & \ \beta \times{\rm tr}\left( {S_B ( {\bar D} )} \right) \Big)= \nonumber\\ & \ \dfrac{1}{{\bar N}}\left( {\alpha \times\left\| {\bar D - \bar M} \right\|_F^2 - \beta \times\left\| {\bar m - m} \right\|_F^2 } \right) \end{align} $
(6) 其中, ${{\bar{d}}_{i}}$是有病字典$\bar{D}$中第$i$个原子, 矩阵$\bar{M}$中列向量均为$\bar{m}$. $\alpha $, $\beta >0$, 分别代表字典的类内距离与类间距离的惩罚因子.
2.2 FCDFDL模型构建及其优化求解
针对DFDL方法的不足, 本文结合Fisher准则, 提出一种FCDFDL方法, 该方法最小化学习字典的类内距离的同时最大化学习字典的类间距离, 以提升无病字典与有病字典之间的差异.其模型定义如下:
$ \left\langle {D^*, X_D^*, \bar X_D^* } \right\rangle =\notag\\ \qquad \underset{D,{{X}_{D}},{{{\bar{X}}}_{D}}}{\mathop{\arg \min }}\, \bigg( \frac{1}{N}\left\| {Y - DX_D } \right\|_F^2 \, - \nonumber\\ \qquad\frac{\rho }{{\bar N}}\left\| {\bar Y - D\bar X_D } \right\|_F^2 + \Psi \left( D \right) \Big) \nonumber\\ \, {\rm s.\, t.}~\left\| {X_D } \right\|_0 < L_1 ~{\rm and}~\left\| {\bar X_D } \right\|_0 < L_1 $
(7) $ \begin{align} &\left\langle {\bar D^*, \bar X_{\bar D}^*, X_{\bar D}^* } \right\rangle = \notag\\ &\qquad \mathop{\arg\min}\limits_{\bar D, \bar X_{\bar D}, X_{\bar D} } \bigg( \frac{1}{{\bar N}}\left\| {\bar Y - \bar D\bar X_{\bar D} } \right\|_F^2\, - \nonumber\\ &\qquad \frac{\rho }{N}\left\| {Y - \bar DX_{\bar D} } \right\|_F^2 + \Psi \left( {\bar D} \right)\bigg) \nonumber\\ & \, {\rm s.\, t.}~\left\| {\bar X_{\bar D} } \right\|_0 < L_2 ~{\rm and}~\left\| {X_{\bar D} } \right\|_0 < L_2 \end{align} $
(8) 式(7)和式(8)目标函数中的第1项、第2项与DFDL方法保持一致.不同之处在于第3项, 即基于Fisher准则构造了学习字典的惩罚项; 与FDDL方法不同, 本文利用Fisher准则直接约束了学习字典的类内距离与类间距离, 而不是约束稀疏表示系数.通过交替优化式(7)和式(8), 可以获得以下性能:
1) 无病字典中原子分布更加紧凑, 对无病样本具有更好的稀疏表示性能, 同时抑制了对有病样本的稀疏表示性能.
2) 有病字典中原子分布更加紧凑, 对有病样本具有较好的稀疏表示性能, 同时抑制了对无病样本的稀疏表示性能.
3) 最大化无病字典与有病字典之间的距离, 大大降低了无病字典与有病字典间的相似性, 增强了学习字典对同类样本的重构性与对非同类样本的判别性.
式(7)和式(8)都是非凸优化问题, 其求解一般通过反复执行稀疏编码与字典更新两个步骤直至收敛. FCDFDL模型求解步骤如下:
步骤1. 稀疏编码
步骤1.1. 固定无病字典$D$, 计算训练样本在无病字典$D$下的稀疏表示系数, 式(7)可重新定义为
$ \begin{align}\label{formula 9} & \left\langle {X_D, \bar X_D } \right\rangle = \mathop{\arg\min}_{X_D, \bar{X}_D} \bigg\{ {\frac{1}{N}\left\| {Y - DX_D } \right\|_F^2 } \, - \notag \\ &\qquad {\frac{\rho }{{\bar N}}\left\| {\bar Y - D\bar X_D } \right\|_F^2 } \bigg\} \notag\\ &\, {\rm s.\, t.}~\left\| {X_D } \right\|_0 < L_1 ~{\rm and}~\left\| {\bar X_D } \right\|_0 < L_1 \end{align} $
(9) 令训练样本$\overset\frown{Y} = \langle Y, \bar{Y} \rangle $, 稀疏表示系数矩阵${{\overset\frown{X}}_{D}}$ $=$ $ \langle {{X}_{D}}, {{{\bar{X}}}_{D}} \rangle $, ${{L}_{1}}$为稀疏度.则式(9)的优化可以分为两步:无病样本在无病字典$D$下的稀疏表示系数与有病样本在无病字典$D$下的稀疏表示系数的求解, 统一简化如下:
$ \begin{align}\label{formula 10} &\overset\frown{\mathit{X}}_D = \mathop{\arg\min}_{\overset\frown{\mathit{X}}_D}\left\| {\overset\frown{\mathit{Y}} - D\overset\frown{\mathit{X}} _D } \right\|_F^2\notag \\ &\, {\rm s.\, t.}~\left\| {\overset\frown{\mathit{X}} _D } \right\|_0 < L_1 \end{align} $
(10) 稀疏解为$\overset\frown{\mathit{X}}_D= \langle {{X}_{D}}, {{{\bar{X}}}_{D}} \rangle $.
步骤1.2. 固定有病字典$\bar{D}$, 计算训练样本在有病字典$\bar{D}$下的稀疏表示系数, 式(8)可重新定义为
$ \begin{align}\label{formula 11} & \left\langle {\bar X_{\bar D}, X_{\bar D} } \right\rangle = \mathop{\arg\min}_{\bar{X}_D, X_D}\bigg\{ {\frac{1}{{\bar N}}\left\| {\bar Y - \bar D\bar X_{\bar D} } \right\|_F^2 \, - } \notag \\ &\qquad \frac{\rho }{N}\left\| {Y - \bar DX_{\bar D} } \right\|_F^2 \bigg\} \notag \\ &\, {\rm s.\, t.}~\left\| {\bar X_{\bar D} } \right\|_0 < L_2 ~{\rm and}~\left\| {X_{\bar D} } \right\|_0 < L_2 \end{align} $
(11) 类似地, 式(11)的优化也分为两步:有病样本在有病字典$\bar{D}$下的稀疏表示系数与无病样本在有病字典$\bar{D}$下的稀疏表示系数的求解, 式(11)可简化成式(10)进行求解, 稀疏解为$\overset\frown{\mathit{X}}_{\bar{D}}= \langle {{{\bar{X}}}_{{\bar{D}}}}, {{X}_{{\bar{D}}}} \rangle $.
本文利用SPAMS工具箱1中的OMP[30]算法求解式(10).
1http://spams-devel.gforge.inria.fr/
步骤2. 字典更新
步骤2.1. 固定无病字典$D$下的稀疏编码系数, 更新无病字典$D$, 式(7)重新定义为
$ \begin{align}\label{formula 12} D =&\ \mathop{\arg\min}_D \bigg\{ \frac{1}{N}\left\| {Y - DX_D } \right\|_F^2 \, - \nonumber\\&\ \frac{\rho } {{\bar N}}\left\| {\bar Y - D\bar X_D } \right\|_F^2 \, + \nonumber\\ &\ \frac{1}{N}\left( {\alpha \times\left\| {D - M} \right\|_F^2 - \beta \times\left\| {m - \bar m} \right\|_F^2 } \right) \bigg\} \end{align} $
(12) 由矩阵的迹可知, $\left\| W \right\|_F^2 = {\rm tr}({W^{{\rm T}}}W) = {\rm tr}(W{W^{{\rm T}}})$, 则式(12)简化为
$ \begin{align}\label{formula 13} D = &\ \mathop{\arg\min}_D \left\{ {\frac{1}{N}{\rm tr} \left( {\left( {Y - D{X_D}} \right){{\left( {Y - D{X_D}} \right)}^{{\rm T}}}} \right) - } \right. \nonumber\\&\ \frac{\rho }{{\bar N}}{\rm tr}\left( {\left( {\bar Y - D{{\bar X}_D}} \right){{\left( {\bar Y - D{{\bar X}_D}} \right)}^{{\rm T}}}} \right)+ \nonumber\\&\ \frac{\alpha }{N}{\rm tr}\left( {\left( {D - M} \right) {{\left( {D - M} \right)}^{{\rm T}}}} \right) - \nonumber\\&\ \left. {\frac{\beta }{N}{\rm tr}\left( {\left( {m - \bar m} \right) {{\left( {m - \bar m} \right)}^{{\rm T}}}} \right)} \right\} \end{align} $
(13) 同时, 忽略式(13)中的常数项, 式(13)可化简为
$ \begin{align}\label{formula 14} &D = \nonumber\\ &~~~~~\mathop{\arg\min}_D \left\{ {2{\rm tr}\left( {\left( { - \frac{1}{N}YX_D^{{\rm T}} + \frac{\rho }{{\bar N}}\bar Y\bar X_D^{{\rm T}}} \right){D^{{\rm T}}}} \right) - } \right. \nonumber\\ &~~~~~~{\rm tr}\left( {D\left( { - \frac{1}{N}{X_D}X_D^{{\rm T}} + \frac{\rho }{{\bar N}}{{\bar X}_D}\bar X_D^{{\rm T}}} \right){D^{{\rm T}}}} \right)+ \nonumber\\ &~~~~~ \frac{\alpha }{N}{\rm tr}\left( {D{D^{{\rm T}}} - 2D{M^{{\rm T}}}} \right) - \\ & \ \ \ \ \left. \frac{\beta }{N}{\rm tr}\left( {m{m^{{\rm T}}} - 2m{{\bar m}^{{\rm T}}}} \right) \right\}\end{align} $
(14) 令$P = \frac{1}{N}YX_D^{{\rm T}} - \frac{\rho }{{\bar N}}\bar Y\bar X_D^{{\rm T}}$, $Q = \frac{1}{N}{X_D}X_D^{{\rm T}} - \frac{\rho }{{\bar N}}{\bar X_D}\bar X_D^{{\rm T}}$, 则式(14)可简化为
$ \begin{align}\label{15} D =&\ \mathop{\arg\min}_D \bigg\{ - 2{\rm tr}\left( {P{D^{{\rm T}}}} \right) +{\rm tr}\left( {DQ{D^{{\rm T}}}} \right) + \notag \\ &\ \frac{\alpha }{N}{\rm tr}\left( {D{D^{{\rm T}}} - 2D{M^{{\rm T}}}} \right) -\notag\\ &\ \frac{\beta }{N}{\rm tr}\left( {m{m^{{\rm T}}} - 2m{{\bar m}^{{\rm T}}}} \right) \bigg\} \end{align} $
(15) 步骤2.2. 固定有病字典$\bar{D}$下训练样本的稀疏编码$\bar{X}_{{\bar{D}}}=\langle {{{\bar{X}}}_{{\bar{D}}}}, {{X}_{{\bar{D}}}} \rangle $, 更新有病字典$\bar{D}$, 令$I=$ $\frac{1}{{\bar N}}\bar Y\bar X_{\bar D}^{{\rm T}}$ $-$ $\frac{\rho }{N}YX_{\bar D}^{{\rm T}}, $ $J = \frac{1}{{\bar N}}{\bar X_{\bar D}}\bar X_{\bar D}^{{\rm T}} - \frac{\rho }{N}{X_{\bar D}}X_{\bar D}^{{\rm T}}$, 则式(8)可简化为
$ \begin{align}\label{16} \bar D =&\ \mathop{\arg\min}_{\bar D} \bigg\{ - 2{\rm tr}\left( {I{{\bar D}^{{\rm T}}}} \right) + {\rm tr}\left( {\bar DJ{{\bar D}^{{\rm T}}}} \right) + \notag\\ &\ \frac{\alpha }{{\bar N}}{\rm tr}\left( {\bar D{{\bar D}^{{\rm T}}} - 2\bar D{{\bar M}^{{\rm T}}}} \right) -\notag\\ &\ \frac{\beta }{{\bar N}}{\rm tr}\left( {\bar m{{\bar m}^{{\rm T}}} - 2\bar m{m^{{\rm T}}}} \right) \Big\} \end{align} $
(16) 式(15)和式(16)均为凸函数, 本文采用坐标梯度下降法可求出学习字典的最优解$ \overset\frown{\mathit{D}} ^* = \langle {D^*, \bar D^* } \rangle $.
2.3 组织病理图像的分类器构造
基于第2.2节, 利用所学习的字典$\overset\frown{\mathit{D}}$对测试样本进行稀疏表示, 可分别求出测试样本在无病字典${{D}^{*}}$与有病字典${{\bar{D}}^{*}}$下的稀疏重构误差, 构造分类统计量实现组织病理图像的分类, 具体分类步骤如下:
步骤1. 将测试图像分块, 将每个图块展开为一个列向量, 随机选取多个图块组成测试样本$H$, 利用
$ \begin{align*} &{{C}^{*}}=\mathop{\arg\min}_C{{\bigg\{ \big\|H-\overset\frown{\mathit{D}}C \big\|_{F}^{2} \bigg\}}_{{}}}_{{}}\notag\\ &\, {\rm s.\, t.}~~~{{\left\| C\right\|}_{0}}<L\end{align*} $
采用OMP方法求出$H$在$\overset\frown{\mathit{D}}$下的稀疏编码系数${{C}^{*}}$ $=$ $\langle {{X}^{*}}, {{{\bar{X}}}^{*}} \rangle $;
步骤2. 计算测试样本在${{D}^{*}}$与${{\bar{D}}^{*}}$下的重构误差向量, 即
$ \begin{align*} &{\delta _1} = {\rm diag}\left\{ \left( H - D^*X^* \right)\left( H - D^*X^* \right)^{{\rm T}} \right\}\\ &{\delta _2} ={\rm diag}\left\{ \left( H - \bar D^*\bar X^* \right)\left( H - \bar D^*\bar X^* \right)^{\rm T}\right\}\end{align*} $
其中, diag$\{ \cdot \}$表示矩阵主对角线上的元素;
步骤3. 定义分类向量
$ \begin{align*} V\left( i \right)= \begin{cases} 1,&{{\delta }_{1}}\left( i \right)\le {{\delta }_{2}}\left( i \right) \\ 0,&{{\delta }_{1}}\left( i \right)>{{\delta }_{2}}\left( i \right) \end{cases}, \quad i=1, \cdots, {{N}_{ T}}\end{align*} $
其中, ${{N}_{ T}}$为测试样本的个数;
步骤4. 基于分类向量$V$, 计算分类统计量$S$ $=$ ${\sum\nolimits_{i=1}^{{{N}_{\rm T}}}{V\left( i \right)}}/{{{N}_{ T}}}$.
当分类统计量$S$大于阈值$Th$, 测试样本为无病样本; 反之, 测试样本则为有病样本.
2.4 本文方法的具体操作步骤
步骤1. 输入无病训练样本$Y$与有病训练样本的$\bar{Y}$, 并分别从$Y$与$\bar{Y}$中随机提取$K$个列向量初始化$D$与$\bar{D}$, 初始化无病与有病的样本个数$N$与$\bar{N}$, 稀疏度${{L}_{1}}$与${{L}_{2}}$, 迭代次数, 惩罚因子$\rho$, $\alpha$, $\beta$;
步骤2. 固定无病字典$D$, 利用式(10)求$\langle Y, \bar{Y} \rangle $在$D$下的稀疏编码系数$\langle {{X}_{D}}, {{{\bar{X}}}_{D}} \rangle $;
步骤3. 固定有病字典$\bar{D}$, 利用式(10)求$\langle \bar{Y}$, $Y \rangle $在$\bar{D}$的稀疏编码系数$\langle {{{\bar{X}}}_{{\bar{D}}}}, {{X}_{{\bar{D}}}} \rangle $;
步骤4. 固定无病字典$D$的稀疏编码$\langle {{X}_{D}}$, ${{{\bar{X}}}_{D}} \rangle $, 求$P$, $Q$, 优化式(15)更新无病字典$D$;
步骤5. 固定有病字典$\bar{D}$的稀疏编码$\langle {{{\bar{X}}}_{{\bar{D}}}}$, ${{X}_{{\bar{D}}}} \rangle $, 求$I$, $J$, 优化式(16)更新有病字典$\bar{D}$;
步骤6. 判断迭代是否完成, 若没有完成迭代次数, 加1转至步骤2;反之, 迭代完成, 输出学习的字典$\langle {{D}^{*}}$, ${{{\bar{D}}}^{*}} \rangle $;
步骤7. 基于学习字典$\langle {{D}^{*}}, {{{\bar{D}}}^{*}} \rangle $, 计算测试样本在${{D}^{*}}$与${{\bar{D}}^{*}}$下的稀疏重构误差, 结合第2.3节构造分类统计量$S$而进行分类.
3. 实验结果及分析
本文分别在ADL[31]与BreaKHis[32]数据集上验证了FCDFDL方法的有效性, 并与其他方法进行对比分析.
3.1 ADL数据集的实验结果
1) ADL数据集及实验设置
ADL数据集宾夕法尼亚州立大学提供, 包括肺、脾脏、肾脏三类器官, 共计900多张图像.每类器官包括无病和有病两类样本, 各150多张, 尺寸为1 360像素$\times$ 1 024像素.为了提高算法的计算效率, 本文将所有图像归一化为600像素$\times$ 600像素.如图 1所示, 图 1 (a)从左至右依次表示肺、脾脏、肾脏的无病图像, 图 1 (b)从左至右依次表示肺、脾脏、肾脏的有病图像.
针对肺、脾脏、肾脏的彩色图像, 在相应的无病与有病样本中分别随机选取40张图像作为训练集, 剩余的110张图像作为测试集.然后, 从每张训练图像中随机提取250个图块, 则每类器官中无病与有病样本分别有10 000个图块, 并将每个图块的RGB三个通道值串成列向量作为训练样本$Y$, $\bar{Y}$.其中, 肺与脾脏图块尺寸为20像素$\times$ 20像素, 肾脏图块尺寸为30像素$\times$ 30像素.以肺部图像为例, $ Y$, $\bar Y$ $\in$ ${\bf R}^{1\, 200 \times 10\, 000} $, 字典$D$, $\bar{D}\in {{\bf R}^{1\, 200\times 100}}$, 最大迭代次数为50.基于第2.4节的步骤, 分别对肺、脾脏、肾脏图像进行分类.其中, 肺部相关实验参数设置为$\rho$ $=$ $0.001$, $\alpha = 1{\text{E}}$$-$$3$, $\beta = 1{\text{E}}$$-$$3$, 脾脏相关实验参数设置为$\rho = 0.001$, $\alpha = 1{\text{E}}$$-$$2$, $\beta = 0.1$, 肾脏相关实验参数设置为$\rho = 0.001$, $\alpha = 1{\text{E}}$$-$$2$, $\beta= 1{\text{E}}$$-$$4$ (实验参数分析见第3.4节).
2) FCDFDL与其他方法的实验对比
应用本文FCDFDL方法, 在不同组织图像上, 与WND-CHARM[33], SRC[12], SHIRC[13], LC-KSVD[26], FDDL[27]和DFDL[29]进行分类性能对比.其中, WND-CHARM结合了对比度、像素及纹理等特征, 并采用SVM分类; SRC和SHIRC方法中的字典并没有经过学习, 采用稀疏重构误差进行分类.表 1~3分别给出了肺、脾脏、肾脏的分类结果.表中结果是采用不同样本分别进行10次实验所取的平均值.
表 1 不同方法在肺部图像的分类结果对比Table 1 Classification results comparison of different methods on lung imagesThe class of samples Health Diseased Method 0.8875 0.1125 WND-CHARM 0.7250 0.2750 SRC Healthy samples 0.7500 0.2500 SHIRC 0.7703 0.2297 LC-KSVD 0.9325 0.0675 FDDL 0.9234 0.0766 DFDL 0.9509 0.0491 FCDFDL 0.3762 0.6238 WND-CHARM 0.2417 0.7583 SRC Diseased samples 0.15 0.85 SHIRC 0.1607 0.8393 LC-KSVD 0.1000 0.9000 FDDL 0.0576 0.9424 DFDL 0.0375 0.9625 FCDFDL 表 2 不同方法在脾脏图像的分类结果对比The class of samples Health Diseased Method 0.5512 0.4488 WND-CHARM 0.7083 0.2917 SRC 0.6500 0.3500 SHIRC Healthy samples 0.8193 0.1807 LC-KSVD 0.8694 0.1306 FDDL 0.8999 0.1001 DFDL 0.9064 0.0936 FCDFDL 0.1275 0.8725 WND-CHARM 0.2083 0.7917 SRC 0.1167 0.8833 SHIRC Diseased samples 0.1457 0.8543 LC-KSVD 0.0857 0.9143 FDDL 0.0599 0.9401 DFDL 0.0409 0.9591 FCDFDL 表 3 不同方法在肾脏图像的分类结果对比Table 3 Classification results comparison of different methods on kidney imagesThe class of samples Healthy Diseased Method 0.6925 0.3075 WND-CHARM 0.7910 0.2090 SRC 0.811 0.189 SHIRC Healthy samples 0.8215 0.1785 LC-KSVD 0.8409 0.1591 FDDL 0.8723 0.1277 DFDL 0.8809 0.1191 FCDFDL 0.2812 0.7188 WND-CHARM 0.2220 0.7780 SRC 0.1946 0.8054 SHIRC Diseased samples 0.1857 0.8143 LC-KSVD 0.1971 0.8029 FDDL 0.1405 0.8595 DFDL 0.1311 0.8689 FCDFDL 在表 1~3中, 第2行给出了不同方法下无病样本的正分率与错分率, 第3行给出了不同方法下有病样本的错分率与正分率.可以看出, 本文FCDFDL方法在肺、脾脏、肾脏的无病样本与有病样本中正分率都有所提高, 错分率有所下降, 具有更好的疾病诊断性能.特别对肺部图像的分类结果提升尤为明显(表 1), 与DFDL相比, 本文方法的分类精度提升了2 %~3 %.表明本文学习的字典对同类样本具有更好的重构性, 对非同类样本具有更好的判别性.
3.2 BreaKHis数据集的实验结果
1) BreaKHis数据集及相关实验设置
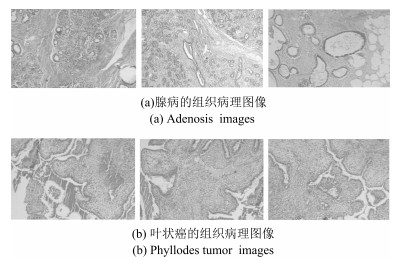
为进一步验证FCDFDL方法的有效性, 本文将其应用于BreaKHis数据集中疾病类型的诊断.该数据集包括不同放大倍数(40$\times$, 100$\times$, 200$\times$, 400$\times$)下82名患者的良性乳腺癌图像, 包括腺病、纤维腺癌、叶状癌和管状腺癌四个类别, 共计2 368张. 40$\times$放大倍数下的腺病与叶状癌两种组织病理图像共计223张, 其中, 腺病图像为114张, 叶状癌图像为109张.图 2 (a)表示腺病的组织病理图像, 图 2 (b)表示叶状癌的组织病理图像.
本文选取40$\times$放大倍数下的腺病与叶状癌两种组织病理图像作为训练样本(这两种病理图像相似度较高), 并将所有图像归一化为600像素$\times$ 600像素.在腺病和叶状癌的彩色图像中各随机选取40张, 每张图像提取250个图块, 块的尺寸为20像素$\times$ 20像素, 则腺病与叶状癌样本分别为10 000个图块.将每个图块的RGB三通道串成列向量作为训练样本, 则$Y$, $\bar{Y}\in {{\bf R}^{1 200\times 10 000}}$, 字典$D$, $\bar{D}$ $\in$ ${{\bf R}^{1 200\times 100}}$, 最大迭代次数为50.采用本文第2.4节的步骤, 分别学习了腺病字典与叶状癌字典, 并利用测试样本在腺病字典与叶状癌字典上的稀疏重构误差进行分类.实验参数设置为: $\rho = 0.001$, $\alpha$ $=$ $1{\text{E}}$$-$$3$, $\beta = 1{\text{E}}$$-$$3$.
2) FCDFDL与其他方法的实验对比
表 4给出了FCDFDL与其他方法在BreaKHis数据集上的分类结果.
表 4 不同方法在BreaKHis数据库上的分类结果对比Table 4 Classification results comparison of different methods on BreaKHis datasetThe class of samples Adenosis Phyllodes tumor Method 0.7225 0.2775 WND-CHARM 0.7875 0.2125 SRC Adenosis samples 0.8775 0.1225 SHIRC 0.8921 0.1079 LC-KSVD 0.8896 0.1104 FDDL 0.9057 0.0943 DFDL 0.9385 0.0615 FCDFDL 0.3192 0.6808 WND-CHARM 0.2925 0.7075 SRC Phylldes tumor samples 0.2875 0.7125 SHIRC 0.1422 0.8578 LC-KSVD 0.1047 0.8953 FDDL 0.0924 0.9076 DFDL 0.0723 0.9277 FCDFDL 可以看出, 与WND-CHARM[33], SRC[12], SHIRC[13], LC-KSVD[26], FDDL[27], DFDL[29]方法相比, 由于本文FCDFDL方法学习了判别性强的腺病字典与叶状癌字典, 更能有效提取图像的分类特征, 取得较好的分类效果.
3.3 学习字典的类间差异
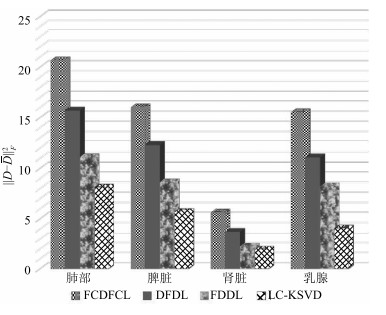
为了进一步探究不同字典学习方法下所获得$D$与$\bar D$的类间差异, 将FCDFDL方法与LC-KSVD, FDDL和DFDL进行主观与客观的比较.图 3为不同方法基于不同组织图像的训练样本所学习的字典示意图.图 3结果显示, 与其他三种方法相比, 本文方法学习的两类字典之间差异明显, 相似程度大大降低.表明结合Fisher准则直接约束学习字典的类内距离与类间距离, 通过优化目标函数式(7)与式(8), 可以最小化学习字典的类内距离与最大化学习字典的类间距离. LC-KSVD, FDDL与DFDL方法学习的$D$与$\bar D$较为相似, 主要原因在于LC-KSVD与FDDL方法仅仅约束稀疏表示系数的判别性, 而DFDL方法在优化过程中并没有考虑学习字典之间的差异.因此, 这三种方法得到的学习字典之间相似度高, 判别性弱.对比分析表明, 本文FCDFDL方法学习的字典包含的细胞结构与纹理更丰富, 颜色信息更全面, 稀疏表示能力更强, 具有更好判别性特征提取能力.
为客观衡量本文方法与LC-KSVD、FDDL和DFDL所学习的字典的类间差异, 采用学习后的字典$D$与$\bar D$的距离作为评价指标(即$\| D-\bar{D} \|_{F}^{2}$), 实验结果如图 4所示.图 4中横坐标表示不同的组织病理图像, 纵坐标表示学习字典的类间距离, 其值越大, 说明两个字典之间的差异越明显.由此可知, 与其他三种方法相比, 本文方法学习字典的类间差异更为明显.因此, 基于Fisher准则构造学习字典的惩罚项, 可以大大降低学习字典之间的相似性, 提高学习字典对非同类样本的判别能力.
3.4 实验参数分析
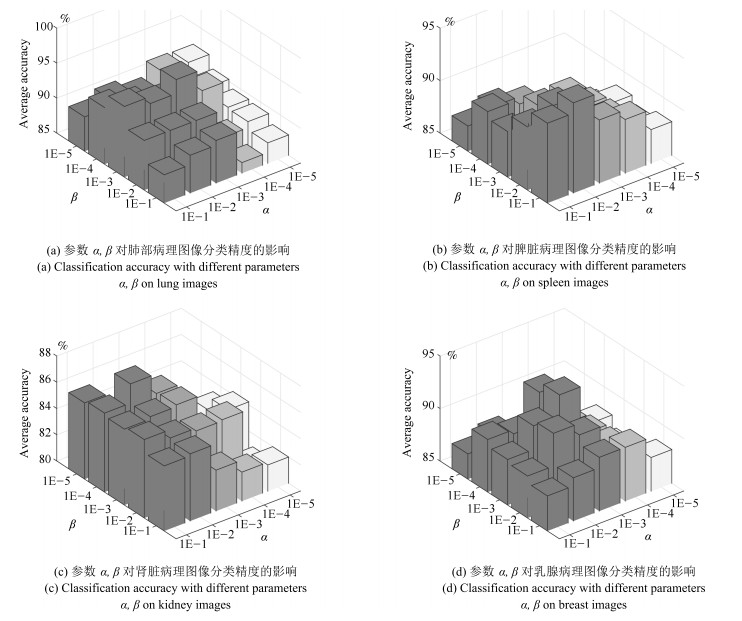
1) 参数$\rho $, $\alpha $, $\beta $的设置
与DFDL方法相同, 本文中参数$\rho$平衡了学习字典对类内样本与类间样本的重构误差, 因此参数$\rho$的设定参考了DFDL方法给出的经验值$\rho$ $=$ $0.001.$不同之处在于FCDFDL方法增加了Fisher准则约束项惩罚因子$\alpha$和$\beta$.图 5给出了随参数$\alpha$, $\beta$变化时, 本文方法在不同组织病理图像的分类精度.从图 5可以看出, 肺部图像在$\alpha$ $=$ $1{\text{E}}$$-$$3$, $\beta = 1{\text{E}}$$-$$3$时分类性能达到最优; 脾脏图像在$\alpha = 1{\text{E}}$$-$$2$, $\beta = 1{\text{E}}$$-$$4$时分类性能达到最优; 肾脏图像在$\alpha = 1{\text{E}}$$-$$3$, $\beta = 1{\text{E}}$$-$$3$时分类性能达到最优; 乳腺图像在$\alpha = 1{\text{E}}$$-$$3$, $\beta = 1{\text{E}}$$-$$3$时分类性能达到最优.
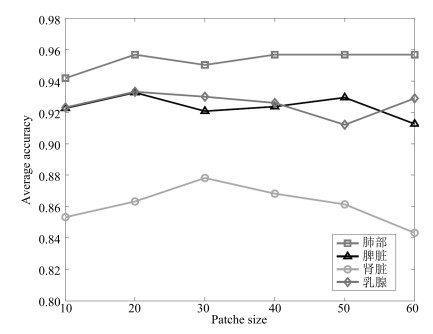
2) 图块尺寸的设置
随着图块尺寸变化, 图 6给出了本文方法在肺、脾脏、肾脏和乳腺的分类精度.由此可知, 肺部、脾脏和乳腺图块尺寸取值为20像素$\times$ 20像素时, 肾脏图块尺寸取值为30像素$\times$ 30像素时, 本文方法的分类性能达到最优.因此, 利用合适尺寸的图块作为训练样本, 能更有效提取图像特征, 取得较佳的分类效果.
4. 总结
针对面向组织病理图像特征提取的字典学习方法存在学习的无病字典与有病字典相似程度高、判别性弱的问题, 本文提出一种新的面向判别性特征的字典学习方法(FCDFDL).利用Fisher准则直接约束无病字典与有病字典的类内距离与类间距离, 构建了字典学习函数的惩罚项, 得到了判别性更强的无病字典与有病字典; 同时, 可最小化学习字典对同类样本的重构误差, 并最大化学习字典对非同类样本的重构误差, 获得了较好稀疏表示性能.最后, 基于学习字典对测试样本的稀疏重构误差构建了分类器, 实现了组织病理图像的二分类.在ADL数据集与BreaKHis数据集上的实验结果表明, 本文方法能有效提取组织病理图像内在的分类特征, 与同类其他算法相比, 具有更好的分类性能.
-
表 1 不同方法在肺部图像的分类结果对比
Table 1 Classification results comparison of different methods on lung images
The class of samples Health Diseased Method 0.8875 0.1125 WND-CHARM 0.7250 0.2750 SRC Healthy samples 0.7500 0.2500 SHIRC 0.7703 0.2297 LC-KSVD 0.9325 0.0675 FDDL 0.9234 0.0766 DFDL 0.9509 0.0491 FCDFDL 0.3762 0.6238 WND-CHARM 0.2417 0.7583 SRC Diseased samples 0.15 0.85 SHIRC 0.1607 0.8393 LC-KSVD 0.1000 0.9000 FDDL 0.0576 0.9424 DFDL 0.0375 0.9625 FCDFDL 表 2 不同方法在脾脏图像的分类结果对比
The class of samples Health Diseased Method 0.5512 0.4488 WND-CHARM 0.7083 0.2917 SRC 0.6500 0.3500 SHIRC Healthy samples 0.8193 0.1807 LC-KSVD 0.8694 0.1306 FDDL 0.8999 0.1001 DFDL 0.9064 0.0936 FCDFDL 0.1275 0.8725 WND-CHARM 0.2083 0.7917 SRC 0.1167 0.8833 SHIRC Diseased samples 0.1457 0.8543 LC-KSVD 0.0857 0.9143 FDDL 0.0599 0.9401 DFDL 0.0409 0.9591 FCDFDL 表 3 不同方法在肾脏图像的分类结果对比
Table 3 Classification results comparison of different methods on kidney images
The class of samples Healthy Diseased Method 0.6925 0.3075 WND-CHARM 0.7910 0.2090 SRC 0.811 0.189 SHIRC Healthy samples 0.8215 0.1785 LC-KSVD 0.8409 0.1591 FDDL 0.8723 0.1277 DFDL 0.8809 0.1191 FCDFDL 0.2812 0.7188 WND-CHARM 0.2220 0.7780 SRC 0.1946 0.8054 SHIRC Diseased samples 0.1857 0.8143 LC-KSVD 0.1971 0.8029 FDDL 0.1405 0.8595 DFDL 0.1311 0.8689 FCDFDL 表 4 不同方法在BreaKHis数据库上的分类结果对比
Table 4 Classification results comparison of different methods on BreaKHis dataset
The class of samples Adenosis Phyllodes tumor Method 0.7225 0.2775 WND-CHARM 0.7875 0.2125 SRC Adenosis samples 0.8775 0.1225 SHIRC 0.8921 0.1079 LC-KSVD 0.8896 0.1104 FDDL 0.9057 0.0943 DFDL 0.9385 0.0615 FCDFDL 0.3192 0.6808 WND-CHARM 0.2925 0.7075 SRC Phylldes tumor samples 0.2875 0.7125 SHIRC 0.1422 0.8578 LC-KSVD 0.1047 0.8953 FDDL 0.0924 0.9076 DFDL 0.0723 0.9277 FCDFDL -
[1] Gurcan M N, Boucheron L E, Can A, Madabhushi A, Rajpoot N M, Yener B. Histopathological image analysis:a review. IEEE Reviews in Biomedical Engineering, 2009, 2:147-171 doi: 10.1109/RBME.2009.2034865 [2] McCann M T, Ozolek J A, Castro C A, Parvin B, Kovacevic J. Automated histology analysis:opportunities for signal processing. IEEE Signal Processing Magazine, 2015, 32(1):78-87 doi: 10.1109/MSP.2014.2346443 [3] Madabhushi A, Lee G. Image analysis and machine learning in digital pathology:challenges and opportunities. Medical Image Analysis, 2016, 33:170-175 doi: 10.1016/j.media.2016.06.037 [4] Dundar M M, Badve S, Bilgin G, Raykar V, Jain R, Sertel O, Gurcan M N. Computerized classification of intraductal breast lesions using histopathological images. IEEE Transactions on Biomedical Engineering, 2011, 58(7):1977-1984 doi: 10.1109/TBME.2011.2110648 [5] Tabesh A, Teverovskiy M, Pang H Y, Kumar V P, Verbel D, Kotsianti A, Saidi O. Multifeature prostate cancer diagnosis and Gleason grading of histological images. IEEE Transactions on Medical Imaging, 2007, 26(10):1366-1378 doi: 10.1109/TMI.2007.898536 [6] Doyle S, Agner S, Madabhushi A, Feldman M, Tomaszewski J. Automated grading of breast cancer histopathology using spectral clustering with textural and architectural image features. In: Proceedings of the 5th IEEE International Symposium on Biomedical Imaging: from Nano to Macro. Paris, France: IEEE, 2008. 496-499 [7] Li W Q, Coats M, Zhang J G, McKenna S J. Discriminating dysplasia:optical tomographic texture analysis of colorectal polyps. Medical Image Analysis, 2015, 26(1):57-69 doi: 10.1016/j.media.2015.08.002 [8] Linder N, Konsti J, Turkki R, Rahtu E, Lundin M, Nordling S, Haglund C, Ahonen T, Pietikäinen M, Lundin J. Identification of tumor epithelium and stroma in tissue microarrays using texture analysis. Diagnostic Pathology, 2012, 7(1):22 doi: 10.1186/1746-1596-7-22 [9] Al-Kadi O S. Texture measures combination for improved meningioma classification of histopathological images. Pattern Recognition, 2015, 43(6):2043-2053 http://www.wanfangdata.com.cn/details/detail.do?_type=perio&id=JJ0215850394 [10] Irshad H, Jalali S, Roux L, Racoceanu D, Hwee L J, Le Naour G, Capron F. Automated mitosis detection using texture, SIFT features and HMAX biologically inspired approach. Journal of Pathology Informatics, 2013, 4(S2):S12 http://d.old.wanfangdata.com.cn/OAPaper/oai_pubmedcentral.nih.gov_3678748 [11] Ergin S, Kilinc O. A new feature extraction framework based on wavelets for breast cancer diagnosis. Computers in Biology and Medicine, 2014, 51:171-182 doi: 10.1016/j.compbiomed.2014.05.008 [12] Wright J, Yang A Y, Ganesh A, Sastry S S, Ma Y. Robust face recognition via sparse representation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2009, 31(2):210-227 doi: 10.1109/TPAMI.2008.79 [13] Srinivas U, Mousavi H, Jeon C, Monga V, Hattel A, Jayarao B. SHIRC: a simultaneous sparsity model for histopathological image representation and classification. In: Proceedings of the 10th IEEE International Symposium on Biomedical Imaging (ISBI). Asilomar, CA, USA: IEEE, 2013. 1118-1121 http://ieeexplore.ieee.org/xpls/abs_all.jsp?arnumber=6556675 [14] Srinivas U, Mousavi H S, Monga V, Hattel A, Jayarao B. Simultaneous sparsity model for histopathological image representation and classification. IEEE Transactions on Medical Imaging, 2014, 33(5):1163-1179 doi: 10.1109/TMI.2014.2306173 [15] Nayak N, Chang H, Borowsky A, Spellman P, Parvin B. Classification of tumor histopathology via sparse feature learning. In: Proceedings of the 10th IEEE International Symposium on Biomedical Imaging (ISBI). Asilomar, CA, USA: IEEE, 2013. 410-413 http://ieeexplore.ieee.org/document/6556499/ [16] Chang H, Zhou Y, Spellman P, Parvin B. Stacked predictive sparse coding for classification of distinct regions in tumor histopathology. In: Proceedings of the 2013 IEEE International Conference on Computer Vision (ICCV). Sydney, NSW, Australia: IEEE, 2013. 169-176 http://ieeexplore.ieee.org/xpl/articleDetails.jsp?arnumber=6751130&filter%3DAND(p_IS_Number%3A6751100) [17] Chang H, Zhou Y, Borowsky A, Barner K, Spellman P, Parvin B. Stacked predictive sparse decomposition for classification of histology sections. International Journal of Computer Vision, 2015, 113(1):3-18 doi: 10.1007/s11263-014-0790-9 [18] Shi J, Li Y, Zhu J, Sun H J, Cai Y. Joint sparse coding based spatial pyramid matching for classification of color medical image. Computerized Medical Imaging and Graphics, 2015, 41:61-66 doi: 10.1016/j.compmedimag.2014.06.002 [19] Zhou Y, Chang H, Barner K, Spellman P, Parvin B. Classification of histology sections via multispectral convolutional sparse coding. In: Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Columbus, OH, USA: IEEE, 2014. 3081-3088 http://dl.acm.org/citation.cfm?id=2679600.2680188 [20] Shi Y H, Gao Y, Yang Y B, Zhang Y, Wang D. Multimodal sparse representation-based classification for lung needle biopsy images. IEEE Transactions on Biomedical Engineering, 2013, 60(10):2675-2685 doi: 10.1109/TBME.2013.2262099 [21] Xu J, Xiang L, Liu Q S, Gilmore H, Wu J Z, Tang J H, Madabhushi A. Stacked sparse autoencoder (SSAE) for nuclei detection on breast cancer histopathology images. IEEE Transactions on Medical Imaging, 2016, 35(1):119-130 doi: 10.1109/TMI.2015.2458702 [22] Zhang X F, Dou H, Ju T, Xu J, Zhang S T. Fusing heterogeneous features from stacked sparse autoencoder for histopathological image analysis. IEEE Journal of Biomedical and Health Informatics, 2016, 20(5):1377-1383 doi: 10.1109/JBHI.2015.2461671 [23] 汤红忠, 张小刚, 陈华, 程炜, 唐美玲.带边界条件约束的非相干字典学习方法及其稀疏表示.自动化学报, 2015, 41(2):312-319 http://www.aas.net.cn/CN/abstract/abstract18610.shtmlTang Hong-Zhong, Zhang Xiao-Gang, Chen Hua, Cheng Wei, Tang Mei-Ling. Incoherent dictionary learning method with border condition constrained for sparse representation. Acta Automatica Sinica, 2015, 41(2):312-319 http://www.aas.net.cn/CN/abstract/abstract18610.shtml [24] 文伟, 王英华, 冯博, 刘宏伟.基于监督非相干字典学习的极化SAR图像舰船目标检测.自动化学报, 2015, 41(11):1926-1940 http://www.aas.net.cn/CN/abstract/abstract18767.shtmlWen Wei, Wang Ying-Hua, Feng Bo, Liu Hong-Wei. Supervised incoherent dictionary learning for ship detection with PolSAR images. Acta Automatica Sinica, 2015, 41(11):1926-1940 http://www.aas.net.cn/CN/abstract/abstract18767.shtml [25] Zhang Q, Li B X. Discriminative K-SVD for dictionary learning in face recognition. In: Proceedings of the 2010 IEEE Computer Vision and Pattern Recognition. San Francisco, CA, USA: IEEE, 2010. 2691-2698 [26] Jiang Z L, Lin Z, Davis L S. Label consistent K-SVD:learning a discriminative dictionary for recognition. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2013, 35(11):2651-2664 doi: 10.1109/TPAMI.2013.88 [27] Yang M, Zhang L, Feng X C, Zhang D. Fisher discrimination dictionary learning for sparse representation. In: Proceedings of the 2011 IEEE International Conference on Computer Vision (ICCV). Barcelona, Spain: IEEE, 2011. 543-550 http://ieeexplore.ieee.org/xpls/abs_all.jsp?arnumber=6126286 [28] 潘宗序, 禹晶, 胡少兴, 孙卫东.基于多尺度结构自相似性的单幅图像超分辨率算法.自动化学报, 2014, 40(4):594-603 http://www.aas.net.cn/CN/abstract/abstract18325.shtmlPan Zong-Xu, Yu Jing, Hu Shao-Xing, Sun Wei-Dong. Single image super resolution based on multi-scale structural self-similarity. Acta Automatica Sinica, 2014, 40(4):594-603 http://www.aas.net.cn/CN/abstract/abstract18325.shtml [29] Vu T H, Mousavi H S, Monga V, Rao U K A, Rao G. DFDL: discriminative feature-oriented dictionary learning for histopathological image classification. In: Proceedings of the 12th International Symposium on Biomedical Imaging (ISBI). New York, NY, USA: IEEE, 2015. 990-994 http://cn.arxiv.org/abs/1502.01032 [30] Tropp J A, Gilbert A C. Signal recovery from random measurements via orthogonal matching pursuit. IEEE Transactions on Information Theory, 2007, 53(12):4655-4666 doi: 10.1109/TIT.2007.909108 [31] Animal Diagnostic Laboratory. Safeguarding animal and human health and facilitating animal agricultureOnline, available: http://vbs.psu.edu/adl, December 12, 2017 [32] Spanhol F A, Oliveira L S, Petitjean C, Heutte L. A dataset for breast cancer histopathological image classification. IEEE Transactions on Biomedical Engineering, 2016, 63(7):1455-1462 doi: 10.1109/TBME.2015.2496264 [33] Orlov N, Shamir L, Macura T, Johnston J, Eckley D M, Goldberg I G. WND-CHARM:multi-purpose image classification using compound image transforms. Pattern Recognition Letters, 2008, 29(11):1684-1693 doi: 10.1016/j.patrec.2008.04.013 期刊类型引用(1)
1. 毛丽珍,汤红忠,范朝冬,曾淑英. 低秩判别性字典学习及组织病理图像分类算法. 小型微型计算机系统. 2019(09): 1881-1885 .  百度学术
百度学术其他类型引用(8)
-





 下载:
下载:

 下载:
下载:
