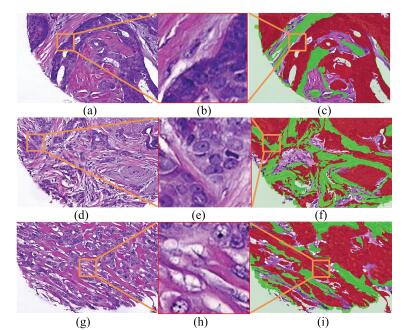
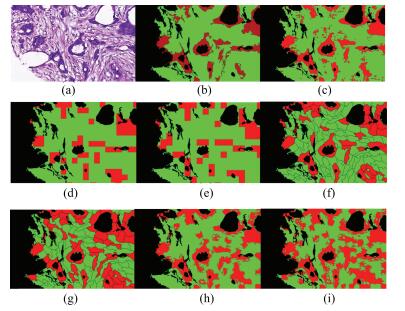
A Deep Convolutional Network for Pixel-wise Segmentation on Epithelial and Stromal Tissues in Histologic Images
-
摘要: 上皮和间质组织是乳腺组织病理图像中最基本的两种组织,约80%的乳腺肿瘤起源于乳腺上皮组织.为了构建基于乳腺组织病理图像分析的计算机辅助诊断系统和分析肿瘤微环境,上皮和间质组织的自动分割是重要的前提条件.本文构建一种基于逐像素点深度卷积网络(CN-PI)模型的上皮和间质组织的自动分割方法.1)以病理医生标注的两类区域边界附近具有类信息为标签的像素点为中心,构建包含该像素点上下文信息的正方形图像块的训练集.2)以每个正方形图像块包含的像素的彩色灰度值作为特征,以这些图像块中心像素类信息为标签训练CN模型.在测试阶段,在待分割的组织病理图像上逐像素点地取包含每个中心像素点上下文信息的正方形图像块,并输入到预先训练好的CN网络模型,以预测该图像块中心像素点的类信息.3)以每个图像块中心像素为基础,逐像素地遍历图像中的每一个像素,将预测结果作为该图像块中心像素点类信息的预测标签,实现对整幅图像的逐像素分割.实验表明,本文提出的CN-PI模型的性能比基于图像块分割的CN网络(CN-PA)模型表现出了更优越的性能.Abstract: Epithelial and stromal tissues are the most common tissue breast cancer pathology images. About 80 percent breast tumors derive from mammary epithelial cells. Therefore, in order to develop computer-aided diagnosis system and analyze the micro-environment of a tumor, it is pre-requisite to segment epithelial and stromal tissues. In this paper, we propose a pixel-wise segmentation based deep convolutional network (CN-PI) model for epithelial and stromal tissues segmentation. The model initially generates two types of training patches whose central pixels are located within annotated epithelial and stromal regions. These context patches accommodate the local spatial dependencies among central pixel and its neighborhoods in the patch. During the testing phase, a square window sliding pixel-by-pixel across the entire image is used to select the context patches. The context patches are then fed to the trained CN-PI model for predicting the class labels of their central pixels. To show the effectiveness of the proposed model, the proposed CN-PI model is compared with 6 patch-wise segmentation based CN models (CN-PA) on two datasets consisting of 106 and 51 hematoxylin and eosin (H&E) stained images of breast cancer, respectively. The proposed model is shown to have F1 classification scores of 90% and 93%; accuracy (ACC) of 90% and 94%, and Matthews correlation coefficients (MCCS) of 80% and 88%, respectively, show improved performances over CN-PA models.1) 本文责任编委 张道强
-
表 1 本文使用的缩写符号及其描述
Table 1 Abbreviated symbols and their meanings in this paper
符号 解释 CN 深度卷积神经网络 LBP 局部区域二值化 SVM 支持向量机 PA 逐图像块 PI 逐像素 $ R^e$ 上皮组织图像块 $ R^s$ 间质组织图像块 SLIC 简单线性迭代聚类算法 Ncut 标准化图割算法 SMC Softmax分类器 EP 上皮组织 ST 间质组织 TMAs 肿瘤组织芯片 IHC 免疫组织化 H & E 苏木精和伊红(染色) DL 深度学习 NKI 荷兰癌症数据所数据集 VGH 温哥华总医院数据集 ReLU 线性纠正函数 LRN 局部响应归一化层 TP 真阳性 FP 假阳性 FN 假阴性 TN 真阴性 TPR 真阳性率 TNR 真阴性率 PPV 阳性预测值 NPV 阴性预测值 FPR 假阳性率 FNR 假阴性率 FDR 伪发现率 ACC 准确率 F1 F1值 MCC 马修斯相关系数 CT 平均每张图像的计算时间 ROC 试者工作特征曲线 AUC ROC曲线下面积 表 2 训练和测试样本的数量
Table 2 The number of training and testing samples
数据集 图像总量 组织 训练图像 测试图像 图像数量 训练集 验证集 图像数量 NKI 106 上皮 85 77 804 41 721 21 间质 70 215 37 625 VGH 51 上皮 41 40 593 16 914 10 间质 36 634 15 264 表 3 本文使用的深度卷积网络结构参数
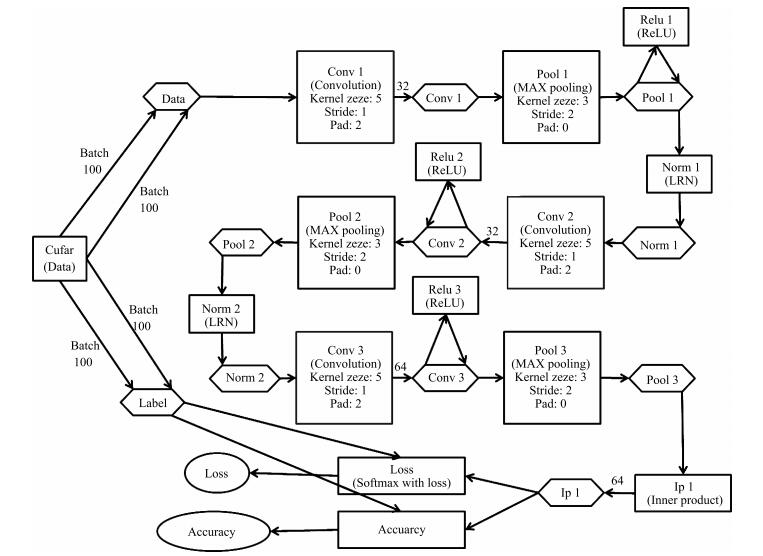
Table 3 The parameters of deep convolution network structure in this paper
层数 操作 通道数 尺寸 步长 边缘填充 激活函数 局部归一化 1 输入 3 - - - - 2 卷积 32 5 1 2 - - 3 池化 32 3 2 0 ReLU LRN 4 卷积 32 5 1 2 ReLU - 5 池化 32 3 2 0 - LRN 6 卷积 64 5 1 2 ReLU - 7 池化 64 3 2 0 - - 8 全连接 64 - - - - - 9 全连接 64 - - - - - 10 输出 2 - - - - - 表 4 本文中不同的对比模型及其描述
Table 4 Different contrast models and their descriptions in this paper
模型 图像块生成方法 图像块尺寸 步长(像素) 网络结构 分类器 预测结果 CN-PA DCNN-SW-SVM[6] 滑动窗+正方形图像块 50×50 25 AlexNet SVM DCNN-SW-SMC[6] 滑动窗+正方形图像块 50×50 25 AlexNet SMC 整个图像 CN-Ncut-SVM[6] 规范化图割+正方形图像块 50×50 AlexNet SVM 区域内像 CN-Ncut-SMC[6] 规范化图割+正方形图像块 50×50 AlexNet SMC 素类信息 CN-SLIC-SVM[6] 简单线性迭代聚类+正方形图像块 50×50 AlexNet SVM 相同 CN-SLIC-SMC[6] 简单线性迭代聚类+正方形图像块 50×50 AlexNet SMC CN-PI 滑动窗+正方形图像块 32×32 1 AlexNet SMC 中心像素类信息 表 5 不同模型分割结果的定量评估(%)
Table 5 Quantitative evaluation of segmentation results for different models (%)
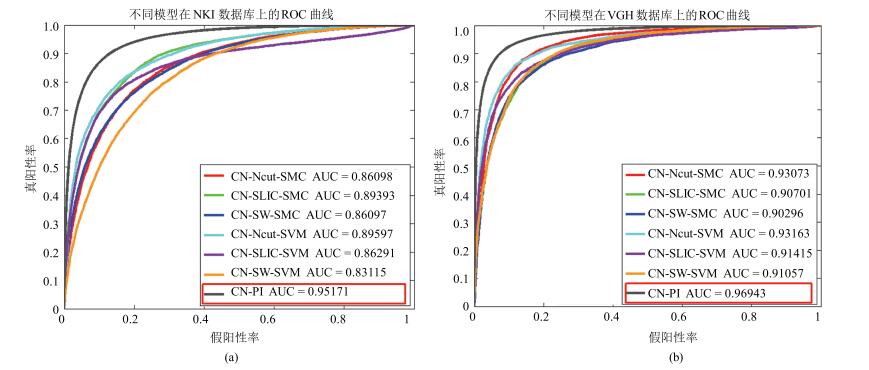
评估指标 模型 数据集 TPR TNR PPV NPV FPR FDR FNR ACC F1 MCC CT(s) CN-PA CN-SW-SVM NKI 70.40 93.87 92.63 74.33 6.13 7.37 29.60 81.60 80.00 65.60 6 VGH 87.01 82.20 85.15 84.36 17.80 14.85 12.99 84.79 86.07 69.36 6 CN-SW-SMC NKI 77.95 80.68 81.63 76.86 19.32 18.37 22.05 79.25 79.25 58.56 3 VGH 82.18 86.12 87.46 80.40 13.88 12.54 17.82 83.99 84.74 68.08 3 CN-Ncut-SVM NKI 81.09 86.39 86.72 80.67 13.61 13.27 18.91 83.62 83.81 67.43 1 265 VGH 88.29 88.40 89.93 86.55 11.60 10.07 11.71 88.34 89.10 76.59 1 265 CN-Ncut-SMC NKI 88.92 67.94 75.23 84.85 32.06 24.77 11.08 78.91 81.05 58.45 1 262 VGH 89.37 86.63 88.67 87.42 13.37 11.31 10.63 88.11 89.03 76.05 1 262 CN-SLIC-SVM NKI 80.63 85.79 86.13 80.18 14.21 13.87 19.37 83.09 83.29 66.37 30 VGH 88.51 83.68 86.41 86.13 16.32 13.59 11.49 86.28 87.45 72.36 30 CN-SLIC-SMC NKI 86.31 82.15 84.11 84.60 17.85 15.89 13.66 84.34 85.21 68.60 26 VGH 87.88 82.13 85.22 85.25 17.87 14.78 12.12 85.23 86.53 70.24 26 CN-PI NKI 91.05 89.54 90.90 89.71 10.46 9.10 8.95 90.34 90.97 80.59 1 742 VGH 95.44 93.41 91.95 96.29 6.59 8.06 4.56 94.30 93.66 88.54 1 742 -
[1] Chen W Q, Zheng R S, Baade P D, Zhang S W, Zeng H M, Bray F, Jemal A, Yu X Q, He J. Cancer statistics in China, 2015. CA:A Cancer Journal for Clinicians, 2016, 66(2):115-132 doi: 10.3322/caac.21338 [2] Siegel R L, Miller K D, Jemal A. Cancer statistics, 2015. CA:A Cancer Journal for Clinicians, 2015, 65(1):5-29 doi: 10.3322/caac.21254 [3] Rubin R, Strayer D S. Rubin's Pathology:Clinicopathologic Foundations of Medicine (5th Edition). Baltimore, MD:Lippincott Williams and Wilkins, 2008. [4] Downey C L, Simpkins S A, White J, Holliday D L, Jones J L, Jordan L B, Kulka J, Pollock S, Rajan S S, Thygesen H H, Hanby A M, Speirs V. The prognostic significance of tumour-stroma ratio in oestrogen receptor-positive breast cancer. British Journal of Cancer, 2014, 110(7):1744-1747 doi: 10.1038/bjc.2014.69 [5] Yuan Y Y, Failmezger H, Rueda O M, Ali H R, Gräf S, Chin S F, Schwarz R F, Curtis C, Dunning M J, Bardwell H, Johnson N, Doyle S, Turashvili G, Provenzano E, Aparicio S, Caldas C, Markowetz F. Quantitative image analysis of cellular heterogeneity in breast tumors complements genomic profiling. Science Translational Medicine, 2012, 4(157):157ra143 http://test.europepmc.org/abstract/MED/23100629 [6] Xu J, Luo X F, Wang G H, Gilmore H, Madabhushi A. A deep convolutional neural network for segmenting and classifying epithelial and stromal regions in histopathological images. Neurocomputing, 2016, 191:214-223 doi: 10.1016/j.neucom.2016.01.034 [7] Linder N, Konsti J, Turkki R, Rahtu E, Lundin M, Nordling S, Haglund C, Ahonen T, Pietikäinen M, Lundin J. Identification of tumor epithelium and stroma in tissue microarrays using texture analysis. Diagnostic Pathology, 2012, 7:22 doi: 10.1186/1746-1596-7-22 [8] Ojala T, Pietikainen M, Maenpaa T. Multiresolution gray-scale and rotation invariant texture classification with local binary patterns. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2002, 24(7):971-987 doi: 10.1109/TPAMI.2002.1017623 [9] Vapnik V N. Statistical Learning Theory. New York:Wiley, 1998. [10] Dalal N, Triggs B. Histograms of oriented gradients for human detection. In:Proceedings of the 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition. San Diego, CA, USA:IEEE, 2005. 886-893 http://dl.acm.org/citation.cfm?id=1068507.1069007 [11] Beck A H, Sangoi A R, Leung S, Marinelli R J, Nielsen T O, van de Vijver M J, West R B, van de Rijn M, Koller D. Systematic analysis of breast cancer morphology uncovers stromal features associated with survival. Science Translational Medicine, 2011, 3(108):108ra113 http://europepmc.org/abstract/MED/22072638 [12] Bourzac K. Software:the computer will see you now. Nature, 2013, 502(7473):S92-S94 doi: 10.1038/502S92a [13] Ren X, Malik J. Learning a classification model for segmentation. In:Proceedings of the 9th IEEE International Conference on Computer Vision. Nice, France:IEEE, 2003. 10-17 http://dl.acm.org/citation.cfm?id=946677 [14] Ali S, Lewis J, Madabhushi A. Spatially aware cell cluster (SpACCl) graphs:predicting outcome in oropharyngeal pl6+ tumors. In:Medical Image Computing and Computer-assisted Intervention:MICCAI International Conference on Medical Image Computing and Computer-assisted Intervention. Berlin Heidelberg:Springer, 2013. 412-419 http://europepmc.org/abstract/MED/24505693 [15] Hiary H, Alomari R S, Saadah M, Chaudhary V. Automated segmentation of stromal tissue in histology images using a voting Bayesian model. Signal, Image and Video Processing, 2012, 7(6):1229-1237 doi: 10.1007/s11760-012-0393-2 [16] Eramian M, Daley M, Neilson D, Daley T. Segmentation of epithelium in H&E stained odontogenic cysts. Journal of Microscopy, 2011, 244(3):273-292 doi: 10.1111/jmi.2011.244.issue-3 [17] Amaral T, McKenna S, Robertson K, Thompson A. Classification and immunohistochemical scoring of breast tissue microarray spots. IEEE Transactions on Biomedical Engineering, 2013, 60(10):2806-2814 doi: 10.1109/TBME.2013.2264871 [18] Bejnordi B E, Balkenhol M, Litjens G, Holland R, Bult P, Karssemeijer N, van der Laak J A W M. Automated detection of DCIS in whole-slide H&E stained breast histopathology images. IEEE Transactions on Medical Imaging, 2016, 35(9):2141-2150 doi: 10.1109/TMI.2016.2550620 [19] Cirećsan D C, Giusti A, Gambardella L M, Schmidhuber J. Mitosis detection in breast cancer histology images with deep neural networks. In:Medical Image Computing and Computer-assisted Intervention:MICCAI International Conference on Medical Image Computing and Computer-assisted Intervention. Berlin Heidelberg:Springer, 2013. 411-418 doi: 10.1007/978-3-642-40763-5_51 [20] Lécun Y, Bottou L, Bengio Y, Haffner P. Gradient-based learning applied to document recognition. Proceedings of the IEEE, 1998, 86(11):2278-2324 doi: 10.1109/5.726791 [21] Serre T, Wolf L, Poggio T. Object recognition with features inspired by visual cortex. In:Proceedings of the 2005 IEEE Computer Society Conference on Computer Vision and Pattern Recognition. San Diego, CA, USA:IEEE, 2005. 994-1000 http://ieeexplore.ieee.org/xpls/icp.jsp?arnumber=1467551 [22] Cruz-Roa A A, Ovalle J E A, Madabhushi A, Osorio F A G. A deep learning architecture for image representation, visual interpretability and automated basal-cell carcinoma cancer detection. In:Medical Image Computing and Computer-assisted Intervention:MICCAI International Conference on Medical Image Computing and Computer-assisted Intervention. Berlin Heidelberg:Springer, 2013. 403-410 doi: 10.1007/978-3-642-40763-5_50 [23] Su H, Xing F, Kong X F, Xie Y P, Zhang S T, Yang L. Robust cell detection and segmentation in histopathological images using sparse reconstruction and stacked denoising autoencoders. Medical Image Computing and Computer-Assisted Intervention——MICCAI 2015. Switzerland:Springer International Publishing, 2015. 383-390 doi: 10.1007/978-3-319-24574-4_46 [24] Xu J, Xiang L, Liu Q S, Gilmore H, Wu J Z, Tang J H, Madabhushi A. Stacked sparse autoencoder (SSAE) for nuclei detection on breast cancer histopathology images. IEEE Transactions on Medical Imaging, 2016, 35(1):119-130 doi: 10.1109/TMI.2015.2458702 [25] Wang H B, Cruz-Roa A, Basavanhally A, Gilmore H, Shih N, Feldman M, Tomaszewski J, Gonzalez F, Madabhushi A. Mitosis detection in breast cancer pathology images by combining handcrafted and convolutional neural network features. Journal of Medical Imaging, 2014, 1(3):034003 doi: 10.1117/1.JMI.1.3.034003 [26] Krizhevsky A, Sutskever I, Hinton G E. Imagenet classification with deep convolutional neural networks. In:Advances in Neural Information Processing Systems 25:Proceedings of the 2012 Conference. Cambridge:MIT Press, 2012. http://dl.acm.org/citation.cfm?id=3065386 [27] Glorot X, Bordes A, Bengio Y. Deep sparse rectifier neural networks. Journal of Machine Learning Research, 2010, 15:315-323 https://hal.archives-ouvertes.fr/hal-00752497 [28] Reinhard E, Adhikhmin M, Gooch B, Shirley P. Color transfer between images. IEEE Computer Graphics and Applications, 2001, 21(5):34-41 http://dl.acm.org/citation.cfm?id=618848 [29] Jia Y Q, Shelhamer E, Donahue J, Karayev S, Long J, Girshick R, Guadarrama S, Darrell T. Caffe:convolutional architecture for fast feature embedding. Eprint Arxiv, 2014. arXiv:1408.5093v1 http://dl.acm.org/citation.cfm?id=2654889&preflayout=flat [30] Schölkopf B, Platt J, Hofmann T. Greedy layerwise training of deep networks. In:Advances in Neural Information Processing Systems 19:Proceedings of the 2006 Conference. Cambridge:MIT Press, 2007. 153-160 -





 下载:
下载: