-
摘要: 现有子空间聚类方法通常以数据全局线性为前提,将每个样本点表示为其他样本点的线性组合,因而导致常见子空间聚类方法不能很好地应用于非线性数据.为克服全局线性表示的局限,借鉴流形学习思想,用k近邻局部线性表示代替全局线性表示,与稀疏子空间聚类和最小二乘子空间聚类方法相结合,提出局部稀疏子空间聚类和局部最小二乘子空间聚类方法,统称局部子空间聚类方法.在双月形数据、6个图像数据集和4个基因表达数据集上进行实验,实验结果表明该方法是有效的.Abstract: Existing subspace clustering methods usually rest on a global linear data set, which expresses each data point as a linear combination of all other data points, and thus common methods are not well suited for the nonlinear data. To overcome this limitation, the local sparse subspace clustering and local least squares regression subspace clustering are proposed. The idea of the two new methods comes from manifold learning which expresses each data point as a linear combination of its k nearest neighbors, and is combined with sparse subspace clustering and least squares subspace clustering respectively. Experimental results show that our method is effective on two-moon synthetic data, six image data sets and four gene expression data sets.
-
Key words:
- Local linear /
- k nearest neighbors /
- subspace clustering /
- image data /
- gene expression data
-
近年来, 人工智能领域成为研究热点, 机器学习、深度学习方面的研究取得了长足的进步. 深度学习在搜索技术、数据挖掘、机器翻译、多媒体学习、语音、计算机视觉以及其他相关领域都取得了很多成果, 广泛应用于医疗、生物、金融、自动驾驶等各个领域[1]. 2006年, Hinton等人[2-3]探讨大脑中的图模型, 提出自编码器来降低数据的维度, 引发了深度学习在研究领域和应用领域的发展热潮. 2011年, Glorot等[4]提出ReLU激活函数, 该激活函数能够有效的抑制梯度消失问题. 2012年, Krizhevsky等人提出[5]提出AlexNet网络模型, 将ImageNet图片分类问题的错误率由26%降低至 15%, 并藉此取得了当年ILSVRC比赛分类项目的冠军, 此后, 卷积神经网络 (Convolutional neural networks, CNN)吸引到了众多研究者的注意, 深度学习进入爆发期. AlexNet 网络模型取得成功后, 随着研究工作的进行, 研究者们又提出了其他的完善方法, 包括ZFNet[6]、VGGNet[7]、GoogleNet[8]和 ResNet[9]等.
深度神经网络(Deep Neural Networks, DNN)在解决复杂问题方面取得了惊人的成功, 广泛应用于生活中各个领域[1], 但是最近的研究表明, 深度神经网络容易受到精心设计的对抗样本的攻击, 导致网络模型输出错误的预测结果[10]. 2014年, Szegedy等人[11]首先在图像分类的背景下发现了深度神经网络的一个弱点, 他们发现深度神经网络虽然预测准确率很高, 但是却极易受到对抗攻击的影响, 并介绍了L-BFGS方法. 对抗攻击通过对模型的输入样本图像加以精心设计的轻微扰动, 诱导模型得出完全偏离真实值的结果. 这种扰动通常非常微小, 无法被人类视觉系统察觉, 但是可以使得神经网络分类器完全改变其关于图像的预测. 更加糟糕的是, 受到攻击的模型对错误预测表示出很高的置信度, 而且相同的图像扰动可以欺骗多个网络模型分类器诱导其产生错误结果[12]. 2015 年, Goodfellow 等人[13]提出一种基于梯度的对抗样本生成算法——快速梯度攻击 (Fast Gradient Sign Method, FGSM), 算法通过寻找深度学习模型梯度变化最大的方向, 根据此方向生成扰动, 藉此增加图像分类器的损失. FGSM 提出的梯度攻击方式操作简单、效果良好, 继而衍生出了许多如PGD等基于梯度的攻击算法[14-16].
对抗样本的攻击能够诱导模型得出完全偏离真实值的结果, 原因在于, 在图片识别任务中, DNN只提取了其中很少的特征, 导致其无法识别甚至误分类具有部分差异的样本, 表现为对现实世界事物理解的局限性, 即在整个模式识别的任务中DNN具有反直觉(counterintuitive)的特性[11]. 对抗攻击产生的结果所导致的深远影响, 引起了研究人员们对对抗攻击以及深度学习的防御攻击的广泛兴趣和研究. 当前深度神经网络容易受到对抗攻击影响的问题, 为当前快速发展的深度学习发展进程敲响警钟, 也引发了许多相关领域人员的研究. 探究能否利用对抗性攻击来训练网络分类器从而提高其鲁棒性是目前深度学习研究的重要方向之一[17]. 防御对抗攻击的方式主要有基于统计检验的检测[15]、修改训练过程或输入样本[18-24]、修改神经网络模型[25-28]、使用附加网络[29-32]等几类方法. 总的来说, 针对不同类型的对抗样本, 都需要一些额外的工作来确保分类器对于新增攻击手段的鲁棒性. 对抗攻击是深度神经网络发展过程中必须克服的一大障碍, 设计一种无需直接修改目标网络模型, 且能够防御多种对抗攻击的算法, 具有强鲁棒性的防御模型是有效推动对抗攻击防御的方向之一. 目前已有的防御方式需要对大量对抗样本挖掘其统计规律, 修改目标网络模型耗费时间、资源, 修改输入样本需要耗费大量人力物力对抗样本及原始样本进行附加预处理步骤, 且鲁棒性较差, 无法同时防御多种攻击算法, 从效果和成本方面考虑, 在工程上耗费了大量时间和资源但成效不佳. 本文将介绍一种更为高效、强鲁棒性的对抗攻击防御模型, 该算法在工程上有效地节约了资源, 在训练结束后, 具有强鲁棒性, 可以防御多种对抗攻击.
2014年, Goodfellow等人[35]提出生成式对抗网络(Generative adversarial networks, GAN). 此后, Radford等[34]人将卷积网络引入GAN的结构, 在CNN的基础上设计了DCGAN. Mirza等人[35]提出着重关注那些阐述样本相关的统计特征并忽略不太相关的局部特征的CGAN. 2017年, Odena 等人[36]提出ACGAN(Auxiliary Classifier GAN), 它将目标函数设置为真实数据样本似然和正确分类标签似然的和, 从而细分调节损失函数使得分类正确率更高, 进一步地提高了网络的生成和判别能力. 生成对抗网络理论提出至今, 其具有的广泛的应用场景使得该理论支持下的衍生模型层出不穷, 在图像生成、超分辨率、风格转换、图像修复等应用中取得显著的进展[37].
鉴于生成式对抗网络在数据表达和分布学习上的优势, 本文将生成对抗网络和现有的攻击算法结合, 提出一种具有强鲁棒性的基于生成对抗网络的对抗攻击防御模型, AC-DefGAN. 本文针对对抗攻击的防御方法进行研究, 利用对抗攻击模型生成攻击样本作为GAN的训练样本, 同时在网络中加入条件约束来指导GAN的训练过程, 最终建立基于生成式对抗网络的对抗攻击防御模型AC-DefGAN, 该模型能够自定义神经网络分类器要防御的攻击算法来完成分类器的防御训练, 使得分类器能够抵挡来自各类攻击算法产生的对抗样本的攻击, 且其训练过程不需要知道目标模型的结构和参数. 在实验部分, 通过MNIST、CIFAR-10和ImageNet数据集验证本文所提出模型的有效性, 对AC-DefGAN对于各攻击算法的防御效果进行验证, 并与其他已有防御方法的防御效果进行比较分析. 实验证明, 本文提出的防御模型能有效防御各类攻击算法产生的对抗样本的攻击, 本模型中的分类器对对抗样本具有较高的分类准确率, 较之其他防御算法, 在保证防御效果的基础上, 具有更强的鲁棒性.
1. 相关工作
自从Szegedy等人发现深度神经网络极易受到攻击以来, 有关的对抗性攻击模型和相应的攻击防御模型在不断的研究中发展, 研究者们提出了各种攻击方法和防御手段. 本节简单介绍一下对抗性攻击方式、对抗性攻击的防御模型和GAN.
在本节中, 我们将回顾近年来关于对抗攻击样本生成、对抗攻击防御和生成式对抗网络的研究工作.
1.1 对抗攻击的方法
对抗样本是对原始样本添加了特定扰动后的样本, 对抗样本能欺骗机器学习技术, 如深度神经网络, 使得分类模型对新构造的样本产生误判. 训练模型的样本集不可能覆盖到所有的可能性, 且很有可能只覆盖到小部分, 所以不可能训练出一个覆盖所有样本的模型, 这就导致了训练的模型边界和真实决策的模型边界有差异, 因此深度神经网络容易受到对抗样本的攻击. 对抗攻击这个领域在深度学习领域中十分活跃, 研究者们提出了各种攻击方法. 下面介绍其中的几种攻击算法.
1、L-BFGS方法
Szegedy在提出对抗样本概念时, 介绍了L-BFGS方法, 即通过寻找最小扰动项
$r$ 来生成对抗样本的方法, 其对应公式如下[11]:$$\mathop {\arg \min }\limits_r f\left( {x + r} \right) = l {\rm{s}}.{\rm{t}}. \left( {x + r} \right) \in D$$ (1) 其中,
$x$ 是能被$f$ 正确分类的原始样本,$r$ 是扰动项, 对抗样本$x' = x + r$ ,$l$ 对抗样本对应的输出标签. 用L-BFGS优化算法求解上述方程, 最终解出最小的扰动添加项$r$ , 根据$x' = x + r$ 得到对抗样本. 该方法在生成对抗性样本时计算量很大.2、FGSM
Goodfellow等[13]在高维度线性假设的基础上提出FGSM(Fast Gradient Sign Method)方法, 是一种基于梯度来生成对抗样本的算法, 算法利用了损失函数的一阶近似来构建对抗样本. 该方法认为如果变化量与梯度的变化方向一致, 就能对分类结果产生最大化的影响. 在整个优化过程中, 需满足
${L_\infty }$ 约束${\left\| {{x^*} - x} \right\|_\infty } \leqslant \varepsilon $ , 即原始样本与对抗样本的误差要在一定范围之内.$${x^*} = x + \varepsilon \cdot {\rm{sign}}\left( {\nabla xJ\left( {x,y} \right)} \right)$$ (2) 其中,
${x^ * }$ 表示对抗样本,$x$ 表示原始样本,$J$ 是训练过程中的损失函数,$\nabla x$ 表示损失函数在$x$ 处的梯度,$\varepsilon $ 表示控制扰动幅度的偏移量级. 相对于其他攻击方式, FGSM只需要进行一次梯度计算就能生成对抗样本, 具有计算量更小、更快的特点.3、BIM
Kurakin等人介绍了BIM(Basic Iterative Method)方法[14], 通过迭代的方式, 沿着梯度增加的方向进行多步小扰动, 并且在每一小步后, 重新计算梯度方向, 其迭代过程表达式如下:
$$I_\rho ^{i + 1} = Cli{p_\varepsilon }\left\{ {I_\rho ^i + \alpha {\rm{sign}}\left( {\nabla J\left( {\theta ,I_\rho ^i,l} \right)} \right)} \right\}$$ (3) 其中,
$I_\rho ^i$ 表示第$i$ 次迭代时的扰动图像,$Cli{p_\varepsilon }\left\{ \cdot \right\}$ 表示当参数为$\varepsilon $ 时图像的像素值,$\alpha $ 表示步长(通常$\alpha {\rm{ = }}1$ ). 对于一个训练好的分类器, BIM方法产生的对抗样本通常与真实类完全不同, 所以这种攻击方法会导致有趣的错误分类.1.2 对抗攻击防御机制
1、APE-GAN
Shen等人[38]提出了一个基于GAN, 利用对抗样本来生成与原始样本相似的重构图像的框架APE-GAN. APE-GAN的最终目标是训练一个生成模型
$G$ , 能够为输入的对抗样本生成对应的与原始样本分布一致的重构样本, 将扰动从对抗样本中完全脱离出来. 训练的优化目标是:$${\mathop \theta \limits^ \wedge _G} = \arg \mathop {\min }\limits_{{\theta _G}} \frac{1}{N}\sum\limits_{k = 1}^N {{l_{ape}}\left( {{G_{{\theta _G}}}\left( {X_k^{adv}} \right),{X_k}} \right)} $$ (4) 其中,
${X_k}$ 表示原始图像,$X_k^{adv}$ 表示对抗样本,${\theta _G}$ 表示生成器网络参数. APE-GAN使用经过训练的网络来消除对抗扰动, 然后将预处理后的样本提供给分类网络, 要实现更好的分类结果需与其他防御网络相结合. 该框架先将样本进行对抗扰动的消除, 需要再让分类网络进行分类, 可以被视为分类之前的附加或预处理步骤, 无法对样本直接进行分类, 样本都需经过该框架的预处理再输入到分类网络中, 消耗较多时间与资源.2、Defense-GAN
Samangouei等[30]提出Defense-GAN, 这是一种利用生成对抗网络来降低对抗性扰动效率的机制, 其核心思想是在将真实图像输入分类器之前, 通过最小化重构误差, 将输入图像“投影”到生成器的范围内. 生成器采用随机噪声输入进行训练, 通过训练后可以模拟未受干扰图像的分布, 从而大大减少了潜在的对抗扰动. 在输入对抗样本时, 会生成一个满足干净样本分布的该对抗样本的近似样本, 然后再将该重构的近似样本
$G\left( z \right)$ 输入到分类器进行分类. 训练目标的数学描述如下式:$${E_{x \sim {p_{data}}}}\left[ {\mathop {\min }\limits_z {{\left\| {{G_t}\left( z \right) - x} \right\|}_2}} \right] \to 0$$ (5) 其中
${G_t}$ 是训练算法训练$t$ 步之后的生成器. Defense-GAN经过训练后, 模拟未受干扰图像的分布, 在输入对抗样本时, 会生成一个该对抗样本的满足干净样本分布的近似样本, 然后再将该样本输入到分类器进行分类. 该防御机制可以被视为分类之前的附加或预处理步骤, 无法对样本直接进行分类.1.3 生成对抗网络
GAN是Goodfellow等在2014年提出的一种生成模型[33]. 该理论基于博弈论场景, 生成器和判别器为博弈双方, 生成器拟合数据的产生过程生成模型样本, 即生成器网络通过与对手竞争来学习变换由某些简单的输入分布到图像空间的分布[39].
GAN由两个模型构成, 生成模型
$G$ 和判别模型$D$ , 其流程图如图1所示. 生成模型$G$ 捕捉真实数据样本的潜在分布, 并生成新的数据样本; 判别模型$D$ 是一个二分类器, 判别输入是从训练数据抽取的样本还是生成器生成的样本. 在学习过程中, 生成器和判别器这两位玩家都试图去最大化自己的收益, 因此GAN 的训练可以看作一个极小极大化优化过程:$$\begin{split} &\mathop {\min }\limits_G \mathop {\max }\limits_D V\left( {D,G} \right) = {E_{x \sim {P_{data}}\left( x \right)}}\left[ {\log D\left( x \right)} \right] + \\ &\quad\quad\quad\quad {E_{z \sim {P_z}\left( z \right)}}\left[ {\log \left( {1 - D\left( {G\left( z \right)} \right)} \right)} \right] \end{split}$$ (6) 其中,
${P_{data}}\left( x \right)$ 和${P_z}\left( z \right)$ 分别表示真实样本概率分布和先验分布.$x$ 表示真实样本,$D\left( x \right)$ 表示$x$ 通过判别网络判断其为真实样本的概率.$z$ 表示输入生成样本的噪声,$G\left( z \right)$ 表示生成网络由噪声$z$ 生成的样本,$D\left( {G\left( z \right)} \right)$ 表示生成样本通过判别网络后, 判断其为真实样本的概率. 生成网络的目的是让生成样本被判别网络判别为真实样本, 即$D\left( {G\left( z \right)} \right)$ 越接近1越好, 这时$V\left( {D,G} \right)$ 会变小; 而判别网络的目的是将$x$ 判别为真实样本, 将生成样本判别为非真实样本, 即让$D\left( x \right)$ 接近1, 而$D\left( {G\left( z \right)} \right)$ 接近 0, 此时$V\left( {D,G} \right)$ 会增大.DCGAN将卷积网络引入GAN的结构, 并通过对网络拓扑结构和超参数的精心设计, 使得 DCGAN 在图像合成任务上表现非常好[34], DCGAN相比于原始GAN有以下特点[40]:
1. 在判别器网络中使用带步幅的卷积层(Strided Convolutions)替换传统卷积神经网路中的池化层(Pooling), 并在生成器网络中使用微步幅卷积层(Fractionally-strided Convolution)完成从随机噪声到图片的生成过程;
2. 在判别器网络和生成器网络中均使用批量归一化(Batch Normalization), 此举通过对隐藏层各神经元的输入作标准化处理, 能够提高神经网络训练速度. 同时可以使前面层的权重变化对后面层造成的影响减小, 整体网络更加健壮;
3. 移除全连接层, 此举以牺牲网络收敛性来增加模型稳定性;
4. 判别器网络中的所有层使用 Leaky ReLU激活函数. 生成器网络中除了输出层以外都使用 ReLU 激活函数, 而输出层则使用 Tanh 激活函数.
ACGAN 针对 GAN 本身不可控的缺点, 在生成模型
$G$ 的建模中加入条件$c$ 监督信息以指导GAN网络生成. 条件$c$ 可以是任意信息, 比如类别信息或者其他模态信息. ACGAN 将目标函数设置为真实数据样本似然和正确分类标签似然的和, 利用输入生成器的标注信息来生成对应的图像标签, 同时还可以在判别器扩展调节损失函数, 从而进一步提高对抗网络的生成和判别能力. 相应的 ACGAN 的目标函数修改为${V_{tf}}\left( {{\theta ^{\left( G \right)}},{\theta ^{\left( D \right)}}} \right)$ (其中,${P_r}$ 和${P_z}$ 分别表示真实样本概率分布和噪声分布,${P_c}$ 表示类别分布):$$\begin{split} &{V_{tf}}\left( {{\theta ^{\left( G \right)}},{\theta ^{\left( D \right)}}} \right) = {E_{x \sim {P_r}}}\left[ {\log D\left( x \right)} \right] + \\ &\quad\quad\quad\quad {E_{z \sim {P_z},c \sim {P_c}}}\left[ {\log \left( {1 - D\left( {G\left( {z,c} \right)} \right)} \right)} \right] \end{split}$$ (7) 同时ACGAN还加入一个分类目标函数
${V_{cls}}\left( {{\theta ^{\left( G \right)}},{\theta ^{\left( D \right)}}} \right)$ :$$\begin{split} & {V_{cls}}\left( {{\theta ^{\left( G \right)}},{\theta ^{\left( D \right)}}} \right) = {E_{x \sim {P_r}}}\left[ {{L_D}\left( {{c_x}|x} \right)} \right] + \\ &\quad\quad\quad\quad {E_{z \sim {P_z},c \sim {P_c}}}\left[ {{L_D}\left( {c|G\left( {z,c} \right)} \right)} \right] \end{split}$$ (8) 最后, ACGAN由上述两个目标函数共同训练. 当条件
$c$ 被定义为标签$y$ 的时候, 则可以认为无监督的GAN模型改进为了有监督模型.2. 基于生成对抗网络的对抗攻击防御模型
通过对生成对抗网络和对抗攻击发展情况的研究, 本文将生成对抗网络和现有的攻击算法结合, 提出一种基于生成对抗网络的对抗攻击防御模型, AC-DefGAN. 该模型提供一种具有强鲁棒性的神经网络分类器的训练方法, 该训练方法可以自定义分类器需要防御的攻击算法, 根据攻击算法来完成分类器的防御训练, 使得分类器能够防御来自对抗样本的攻击. 本文提出的方法与已有算法相比, 可以防御多种攻击算法的攻击, 训练后的分类器可以直接对对抗样本进行正确判别与分类, 鲁棒性强. 对抗训练是防御对抗样本攻击的有效方法, 但是它也存在着局限性. 对抗训练是通过不断输入新类型的对抗样本进行训练, 从而不断提升模型的鲁棒性. 为了保证有效性, 该方法需要使用高强度的对抗样本, 并且网络架构要有充足的表达能力. 对抗训练的过程是在训练集上进行的, 由于训练数据集的经验分布与真实数据分布并非完全相同, 那么从真实数据分布中提取的测试点可能位于训练数据集经验分布的“盲点”中, 不被对抗训练过程“覆盖”, 存在泛化差距, 影响对抗训练的有效性[41]. 一方面, 如果网络对图像分布有更深入的了解, 则可以提高对抗训练分类器的鲁棒性. 在生成对抗网络中, 生成器提供了关于数据分布的更多信息, 利用GAN在数据学习上的优势, 可以进行有效的数据扩充, 改善对抗训练过程, 提高训练后分类器的鲁棒性. 另一方面, 对抗样本能够很容易“欺骗”分类器, 在GAN的训练过程中, 生成器寻找能够“欺骗”判别器的样本, 当判别器能识别出一种模式的对抗样本时, 生成器很容易就能够找到另一种“欺骗”判别器的样本, 在这种情况下训练难以达到纳什平衡. 将GAN和对抗训练相结合, 在GAN训练的过程中将对抗样本作为训练样本加入到训练中, 可以促使GAN的训练更加稳定, 提高收敛速度, 最终训练得到鲁棒性更强、防御效果更好的分类器. 相较于其他以GAN为基础的防御方法, 本文所提出模型更为高效, 更加节省资源. Defense-GAN的主要思想是利用生成器重构对抗样本来实现防御, 通过训练, 可以模拟未受干扰图像的分布, 在输入对抗样本时, 生成器生成一个该对抗样本的满足干净样本分布的近似样本, 然后再将该近似样本输入到分类器进行分类. APE-GAN防御方法与Defense-GAN相似, 也是以利用生成器重构对抗样本为目的, 来实现防御. 而本文方法的主要思想是利用判别器作为分类器对对抗样本进行正确分类, 通过生成器和判别器的博弈, 训练判别器能不被扰动所干扰, 能对对抗样本进行正确分类, 以达到防御目的. Defense-GAN防御方法在对样本进行分类时, 需要先对对抗样本进行图像重构, 再将重构图像输入到分类器中进行图像分类. 本文方法在对样本进行分类时, 直接利用判别器进行图像分类. 另外, 当未知样本为对抗样本或是原始样本时, AC-DefGAN可以对样本进行直接分类; 而对于Defense-GAN方法, 需要确定样本是对抗样本再进行使用, 否则对于原始样本进行重构再分类耗费资源且无法保证分类正确性.
在生成对抗模型中, 生成器学习将白噪声转换为在判别器看来可信的图像, 生成器和判别器互相博弈, 加强彼此. 生成式对抗网络中的判别器在通过训练后, 能提高对样本的判别能力, 最小化自身损失[42]. 对抗攻击算法对原始样本添加扰动, 使得对抗样本在能被人眼识别的同时使得已训练好的分类器对其产生误分类, 且对抗样本具有迁移性, 利用攻击算法攻击目标网络可以产生对抗样本. 通过将对抗样本加入到生成对抗网络的训练中, 可以利用GAN在数据学习上的优势提高分类器在对抗训练中的鲁棒性, 同时对抗攻击也能提高GAN的优化效率从而更好地训练生成器判别器, 提高训练后分类器的鲁棒性, 使得该模型具有更强的泛化能力. 因此, 方法的目标如下: 给定噪声信号
$z$ 、类别信号${c_{fake}}$ 、真实样本${x_{real}}$ 、${x_{real}}$ 对应类别${c_{real}}$ , 在判别损失和分类损失等损失函数的指导下交替训练生成器和判别器, 从而引导判别器逐渐提高对样本的判别能力和分类能力.本文提出的AC-DefGAN的整体框架如图2所示. 框架由四个部分组成: 攻击算法集合
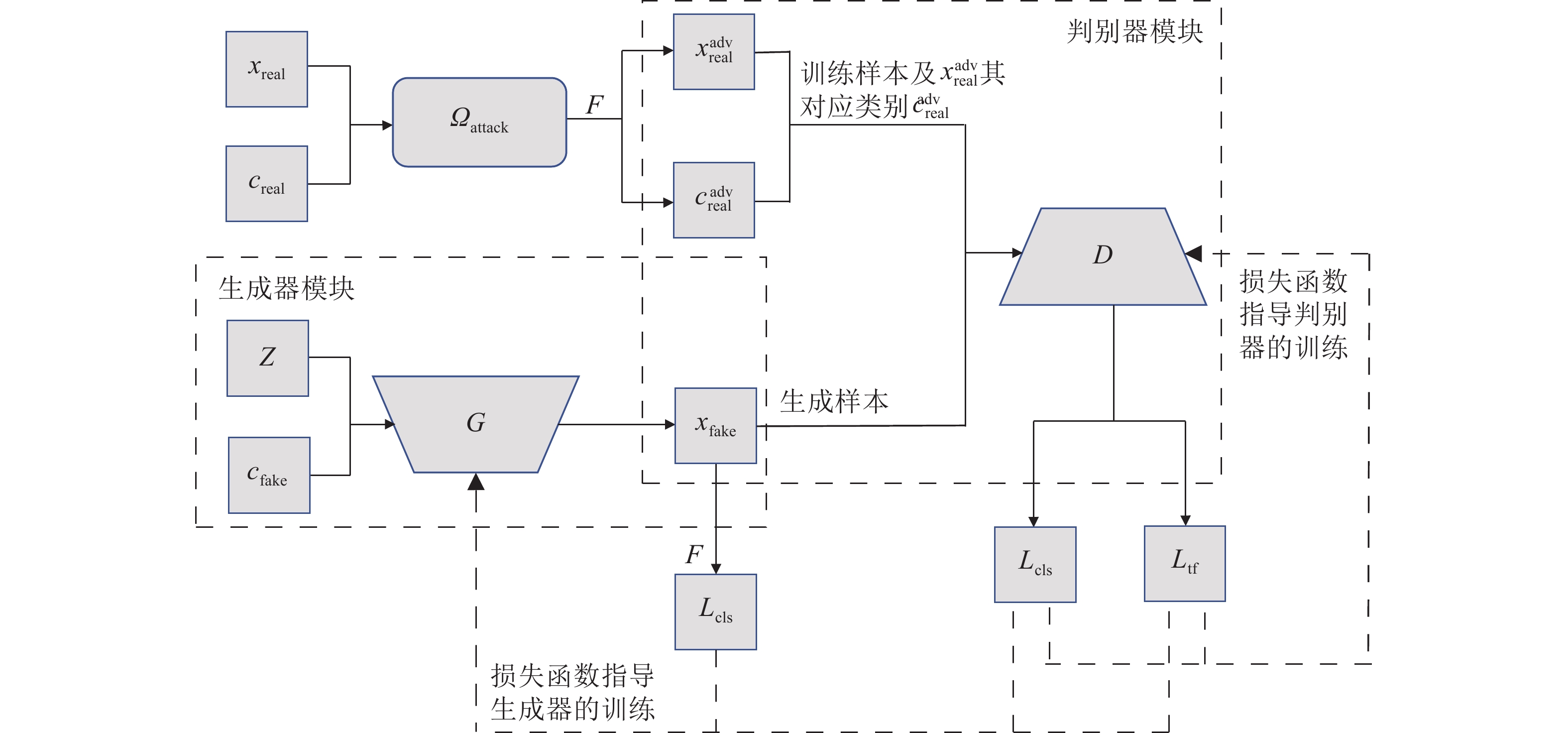
${\Omega _{attack}}$ 、被攻击的目标网络$F$ 、生成器$G$ 、判别器$D$ . 其中,${\Omega _{attack}}$ 由对神经网络分类器造成威胁的攻击算法组成, 例如Fast Gradient Sign Method (FGSM)、iterative least-likely class method (iter_FGSM)、Basic Iterative Methods(BIM)、Carlini and Wagner Attacks (C&W)等各类对抗攻击算法, 该部分负责完成对抗样本的生成, 作为模型的训练集; 被攻击的目标网络$F$ 负责指导训练生成器$G$ 和对抗样本; 生成器$G$ 负责生成带扰动的逼真图像; 判别器$D$ 判别输入是真实数据还是生成的样本, 并将样本进行正确分类. 为了生成具有指定语义的样本, 需要控制所生成样本的标签, 因此本文借鉴了ACGAN中的方法将类别信号引入生成器, 从而引导模型训练; 最后为了让生成样本具有攻击性, 修改 C&W 攻击的优化函数作为对抗损失函数.${\Omega _{attack}}$ 接收真实样本${x_{real}}$ 及其对应类别${c_{real}}$ 作为输入, 以分类器$F$ 作为攻击目标, 输出攻击样本$x_{real}^{adv}$ 和攻击样本对应的正确类别$c_{real}^{adv}$ . 生成器$G$ 接收随机噪声$z$ 和随机条件向量${c_{fake}}$ 作为输入数据, 生成虚假图像${x_{fake}}$ .$D$ 对${x_{fake}}$ 的真假判别损失${L_{tf}}(G)$ 和分类损失${L_{cls}}(G)$ . 分类器$F$ 对${x_{fake}}$ 进行分类, 得到对抗损失${L_{adv}}(G)$ . 损失函数${L_{tf}}(G)$ 、${L_{cls}}(G)$ 和${L_{adv}}(G)$ 一同指导训练生成器$G$ .$D$ 接收$x_{real}^{adv}$ , 输出对$x_{real}^{adv}$ 真假样本判定损失$L_{tf}^{adv}(D)$ 和分类损失$L_{cls}^{adv}(D)$ .$D$ 接收${x_{fake}}$ , 输出对${x_{fake}}$ 真假样本判定损失$L_{tf}^{fake}(D)$ 和分类损失$L_{cls}^{fake}(D)$ . 损失函数$L_{tf}^{adv}(D)$ 、$L_{cls}^{adv}(D)$ 、$L_{tf}^{fake}(D)$ 和$L_{cls}^{fake}(D)$ 一同指导判别器$D$ 的训练.生成式对抗网络理论基于博弈论场景, 其中生成器网络通过与对手竞争来学习变换由某些简单的输入分布到图像空间的分布; 作为对手, 判别器则试图区分从训练数据抽取的样本和从生成器中生成的样本. 这个过程中, 判别器的分类能力和鲁棒性都得到提升. 生成器
$G$ 的目标是生成具有攻击性的逼真样本, 判别器$D$ 的目标是区分生成的假样本和真实数据, 并对样本进行正确分类. 在此模型中, 判别器接收经过攻击的真实数据及其对应类别信息, 同时生成器捕捉真实数据样本的潜在分布, 并生成新的数据样本输入到判别器中; 作为对手, 判别器试图区分是生成的假样本还是真实数据并试图对其进行正确分类. 在博弈过程中, 生成器的目标是“欺骗”判别器使其对于生成样本做出“误判”, 判别器试图区分真假样本并对其进行正确分类, 生成器和判别器互相竞争, 共同进步. 通过这种方式, 两者试图最大化各自收益, 最小化各自损失, 最终使得生成器生成的样本越来越逼真越来越具有攻击性, 判别器对样本的真假判定及分类能力越来越高. 判别损失${L_{tf}}$ 引导${x_{fake}}$ 越来越接近真实样本, 并且引导$D$ 不断提高对生成样本和真实样本的判别能力; 对抗损失${L_{adv}}$ 引导${x_{fake}}$ 越来越具有攻击性, 从而引导$D$ 提高对样本分类能力; 分类损失${L_{cls}}$ 使得$D$ 能够有效对样本进行分类, 促使$G$ 能够根据类别信号${c_{fake}}$ 生成指定样本.为训练生成对抗网络, 使得生成器和判别器达到各自目标,
$G$ 和$D$ 的损失函数设计如下:对于生成器
$G$ , 其输入为随机噪声$z$ 和类别信息${c_{fake}}$ , 输出生成样本${x_{fake}}$ .$G$ 的损失函数$L(G)$ 定义为三个损失函数的加权和, 分别是判别器$D$ 对${x_{fake}}$ 进行真实样本或生成样本判定的判别损失${L_{tf}}(G)$ , 判别器$D$ 对${x_{fake}}$ 进行分类预测的分类损失${L_{cls}}(G)$ , 以及将${x_{fake}}$ 作为目标攻击网络$F$ 的输入得到的对抗损失${L_{adv}}(G)$ . 其中,$\alpha $ 、$\beta $ 和$\delta $ 负责控制每个损失函数之间的相对重要程度. 其中${L_{tf}}(G)$ 、${L_{cls}}(G)$ 训练生成器学习样本的原始数据分布, 使得生成器生成的样本越来越逼近于真实样本, 即生成器分布逼近目标分布, 生成能“欺骗”判别器的生成样本;${L_{adv}}(G)$ 训练生成器学习扰动对样本造成的影响, 生成能够欺骗分类器带扰动的样本, 保证生成样本具有攻击性, 该损失表示目标网络预测类别与真实类别的距离的负值. 生成器在损失函数的指导下不断更新, 使得生成的样本越来越逼真越来越具有攻击性, 促使判别器在训练中能不断提升对于样本的真假及类别的判别能力. 训练时${L_{tf}}(G)$ 、${L_{cls}}(G)$ 所占权重过高,${L_{adv}}(G)$ 所占权重过低, 会导致训练得到的生成器生成的样本攻击性弱, 造成判别器对于对抗攻击的防御性能不佳, 鲁棒性差; 训练时${L_{adv}}(G)$ 所占权重过高,${L_{tf}}(G)$ 、${L_{cls}}(G)$ 所占权重过低, 会导致训练生成器无法完美的学习到训练样本的分布, 造成判别器对于原始样本的判别能力不佳, 最终影响判别器的分类性能. 在训练时, 对于各损失函数的权重设置, 应在保证生成器学习到训练样本的数据分布的情况下提高生成样本的攻击性, 以保证最终训练得到的判别器对于样本的判别能力更强.$${L_{tf}}{\rm{(}}G{\rm{) = }}{E_{z \sim {P_z},{c_{fake}} \sim {P_c}}}[\log (1 - D(G(z,{c_{fake}})))]$$ (9) $${L_{cls}}{\rm{(}}G{\rm{)\! = }}{{\rm{E}}_{z \sim {P_z},{c_{fake}} \sim {P_c}}}[{L_D}({c_{fake}}|G(z,\!{c_{fake}}))]$$ (10) $$\begin{split} {L_{adv}}(G) = & - {E_{z \sim {P_z},{c_{fake}} \sim {P_{c,}}{x_{fake}} \sim {P_G}}} \\ & [{L_F}({c_{fake}}|{x_{fake}} + G(z))] \\ \end{split} $$ (11) $$L{\rm{(}}G{\rm{) = }}\alpha {L_{tf}}(G) + \beta {L_{cls}}{\rm{(}}G{\rm{)}} + \delta {L_{adv}}(G)$$ (12) 对于判别器
$D$ , 其输入为生成器输出的生成样本${x_{fake}}$ 、攻击样本$x_{real}^{adv}$ 和攻击样本对应的正确类别$c_{real}^{adv}$ . 其中,$x_{real}^{adv}$ 是${\Omega _{attack}}$ 以${x_{real}}$ 作为输入, 以目标网络$F$ 作为攻击目标输出的攻击样本;$c_{real}^{adv}$ 是攻击样本$x_{real}^{adv}$ 对应的所属正确类别.$D$ 的损失函数$L(D)$ 定义为四个损失函数的加权和, 分别是判别器$D$ 对$x_{real}^{adv}$ 进行真假样本判别的判别损失$L_{tf}^{adv}(D)$ , 判别器$D$ 对$x_{real}^{adv}$ 进行分类预测的分类损失$L_{cls}^{adv}(D)$ , 判别器$D$ 对${x_{fake}}$ 真假样本判别的判定损失$L_{tf}^{fake}(D)$ , 以及判别器$D$ 对${x_{fake}}$ 进行分类预测的分类损失$L_{cls}^{fake}(D)$ . 其中,$L_{tf}^{adv}(D)$ 和$L_{cls}^{adv}(D)$ 训练判别器对训练集中添加了扰动的真实样本的判别能力,$L_{tf}^{fake}(D)$ 和$L_{cls}^{fake}(D)$ 训练判别器对生成器生成的样本的判别能力,$L_{tf}^{adv}(D)$ 和$L_{cls}^{adv}(D)$ 的权重影响判别器对原始数据分布的判别能力, 影响生成器对于训练样本分布的学习;$L_{tf}^{fake}(D)$ 和$L_{cls}^{fake}(D)$ 的权重影响判别器对于对抗样本的判别能力, 影响生成器生成的样本的攻击性.$L_{tf}^{adv}(D)$ 、$L_{cls}^{adv}(D)$ 、$L_{tf}^{fake}(D)$ 和$L_{cls}^{fake}(D)$ 共同指导下, 训练判别器对于各类样本的分类性能.$$\begin{split} L_{tf}^{adv}(D) = & - {E_{{x_{real}} \sim {P_{real}},{f_{attack}} \in {\Omega _{attack}}}}\\ & {[\log D({f_{attack}}({x_{real}}))]} \end{split}$$ (13) $$\begin{split} L_{cls}^{adv}(D) = & {E_{{x_{real}} \sim {P_{real}},{f_{attack}} \in {\Omega _{attack}}}}\\ &{[{L_D}(c_{real}^{adv}|{f_{attack}}({x_{real}}))]} \end{split}$$ (14) $$L_{tf}^{fake}(D) = - {E_{z \sim {P_z},{c_{fake}} \sim {P_c}}}\left[\log (1 - D(G(z,c{}_{fake})))\right]$$ (15) $$L_{cls}^{fake}{\rm{(D) = }} - {{\rm{E}}_{z \sim {P_z},{c_{fake}} \sim {P_c}}}\left[{L_D}({c_{fake}}|G(z,{c_{fake}}))\right]$$ (16) $$L(D) = L_{tf}^{fake}(D) + L_{tf}^{adv}(D) + L_{cls}^{fake}(D) + L_{cls}^{adv}(D)$$ (17) 以上述各损失函数作为指导, 依次固定生成器
$G$ 和判别器$D$ 的参数, 对$G$ 和$D$ 进行交替训练. 在训练过程中, 采用批次训练方案, 采用 Adam优化器优化损失函数, 在$G$ 和$D$ 的训练比例设置上应使得$G$ 和$D$ 保持对抗平衡.3. 实验
为了验证本文所提算法的有效性, 本文在MNIST、CIFAR-10和ImageNet数据集上进行实验分析.
1)MNIST数据集包含0~9的10类手写数字灰度图像, 图像大小为28像素×28像素, 整个数据集有60000个训练样本, 10000个测试样本[43].
2)CIFAR-10数据集包含10类彩色图像, 图像大小为32像素×32像素, 数据集有50000个训练样本, 10000测试样本[44].
3)ImageNet是一个计算机视觉系统识别项目, ImageNet数据集是目前世界上图像识别最大的数据库, 提供超过1000000个样本, 包含1000多个类[45].
实验环境: 计算机处理器为 32 Intel(R) Xeon(R) CPU E5-2620 v4 @ 2.10 GHz, 64 GB 运行内存 (RAM), 两块 NVIDIA Tesla P4 GPU, PyTorch 框架.
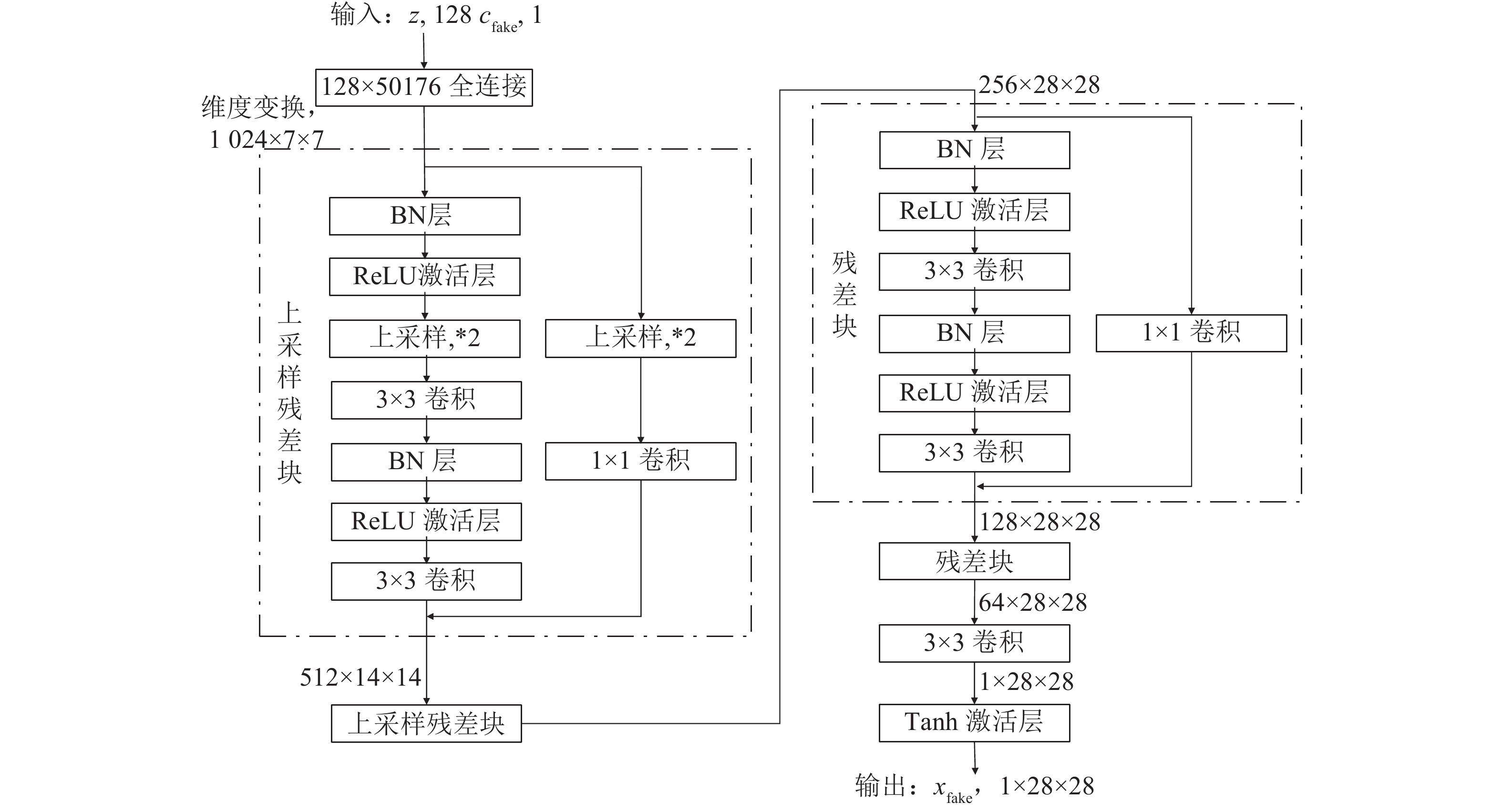
实验中使用的生成器和判别器结构分别如图3和图4所示(以MNIST手写数字数据集为例). 我们采用以残差单元为基础的简单残差网络作为生成器和判别器的架构. 在生成器和判别器中, 均去除反卷积, 只保留普通卷积层; 通过 UpSampling2D 和 AvgPooling2D 实现上采样和下采样; 卷积核的大小统一使用3×3, 步长为1. 生成器除了最后一层使用Tanh激活函数, 其他层使用ReLU激活函数; 判别器使用ReLU作为激活函数. 生成器模型中Batch normalization(BN) 层将对隐藏层的输入进行归一化; 另外, 由于Batch Normalization 会引入同一个batch中不同样本的相互依赖关系, 而本文模型需要对每个样本独立地施加梯度惩罚, 所以判别器的模型架构中不使用BN.
在训练过程中, 采用批训练的方法, 交替训练生成器和判别器. 训练生成器时, 固定判别器参数, 获取batch-size大小的随机噪声
$z$ 和随机条件向量${c_{fake}}$ , 输入生成器中生成batch-size大小的生成样本, 生成样本传入判别器和分类器中, 得到生成器的损失函数, 反向传播并更新生成器的参数. 训练判别器时, 固定生成器参数, 从训练样本中获取batch-size大小的数据输入判别器中以得到对应的损失函数, 获取batch-size大小的噪声$z$ 和随机条件向量${c_{fake}}$ , 输入生成器中生成batch-size大小的生成样本, 生成样本传入判别器中得到对应的损失函数, 反向传播并更新判别器的参数. 重复交替训练生成器和判别器, 直至网络训练完成, 每完整循环一次训练样本即完成一轮训练, 保存生成器和判别器的相关参数.${\Omega _{attack}}$ 由对神经网络分类器造成威胁的攻击算法组成, 生成攻击样本. 攻击算法流程图如图5所示, 从攻击算法集合中随机选取其中一种攻击算法攻击目标网络, 若攻击成功则保留样本, 直至攻击样本数量大小等于batch-size. 在实验中, 采用FGSM、BIM、DeepFool、C&W (Carlini and Wagner Attacks)[46]、PGD[17]等多种不同的攻击方法组成攻击算法集合来产生对抗样本, 用于模型的训练中.3.1 MNIST实验
在训练过程中, 使用图3和图4的结构作为 AC-DefGAN的生成器和判别器对 MNIST 数据集进行训练, 超参数设置如下:
1. Batch设置为64;
2. Epoch 设置为25轮;
3. 为了保持对抗平衡, 设置判别器与生成器的迭代次数为2: 1;
4. Adam 优化器的学习速率设置为0.0002, 一阶矩估计的指数衰减率为0.5, 二阶矩估计的指数衰减率为0.9;
5. 根据各个损失函数对训练结果的影响, 为保证训练生成器完美的学习到训练样本的分布且生成的样本具有攻击性, 以使得训练得到的判别器对于原始样本和对抗样本具有较强的分类, 将损失函数
${L_{tf}}(G)$ 、${L_{cls}}(G)$ 和${L_{adv}}(G)$ 的权重$\alpha $ :$\beta $ :$\delta $ 设置为10:10:1.3.1.1 MNIST实验结果分析
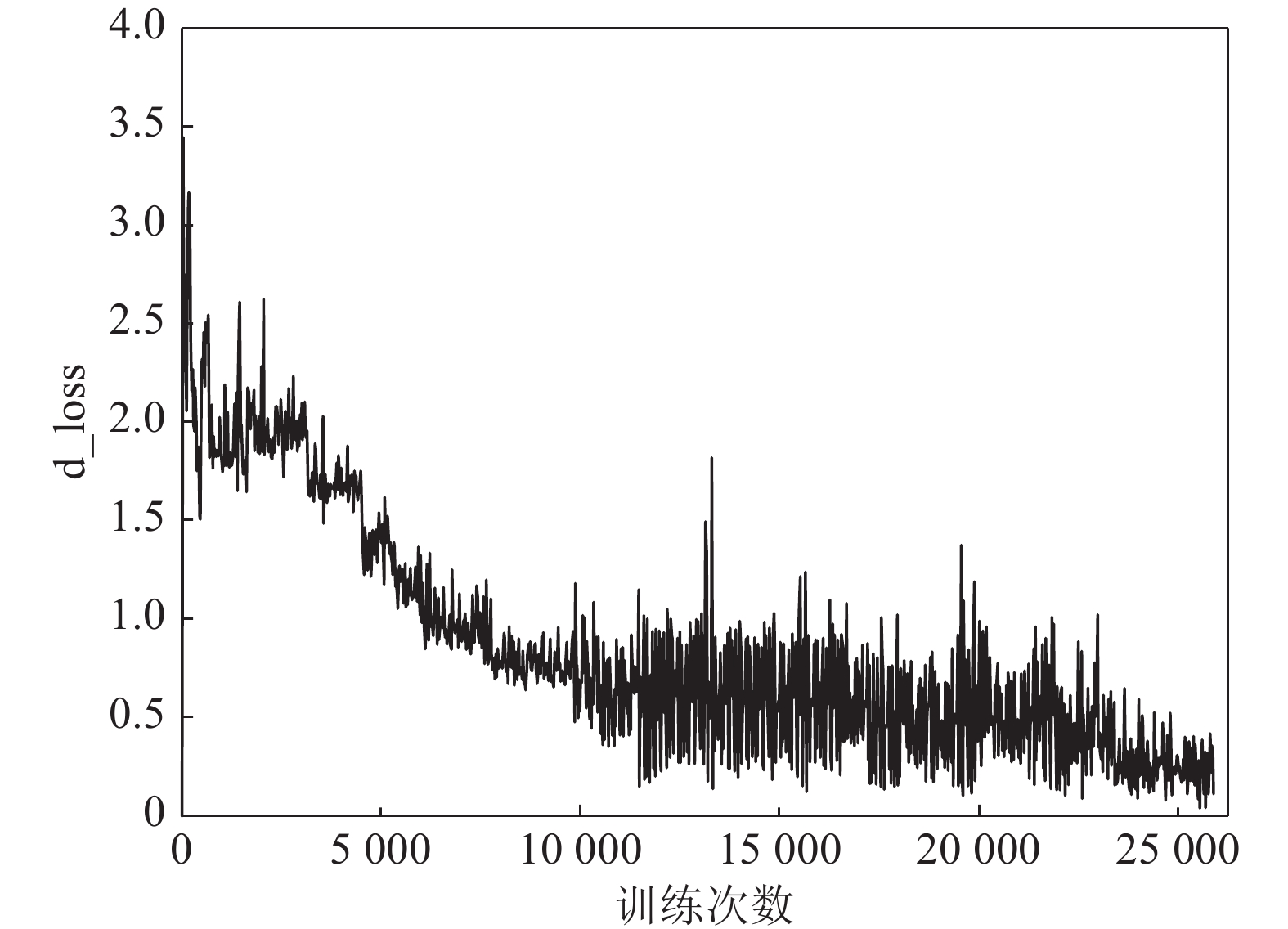
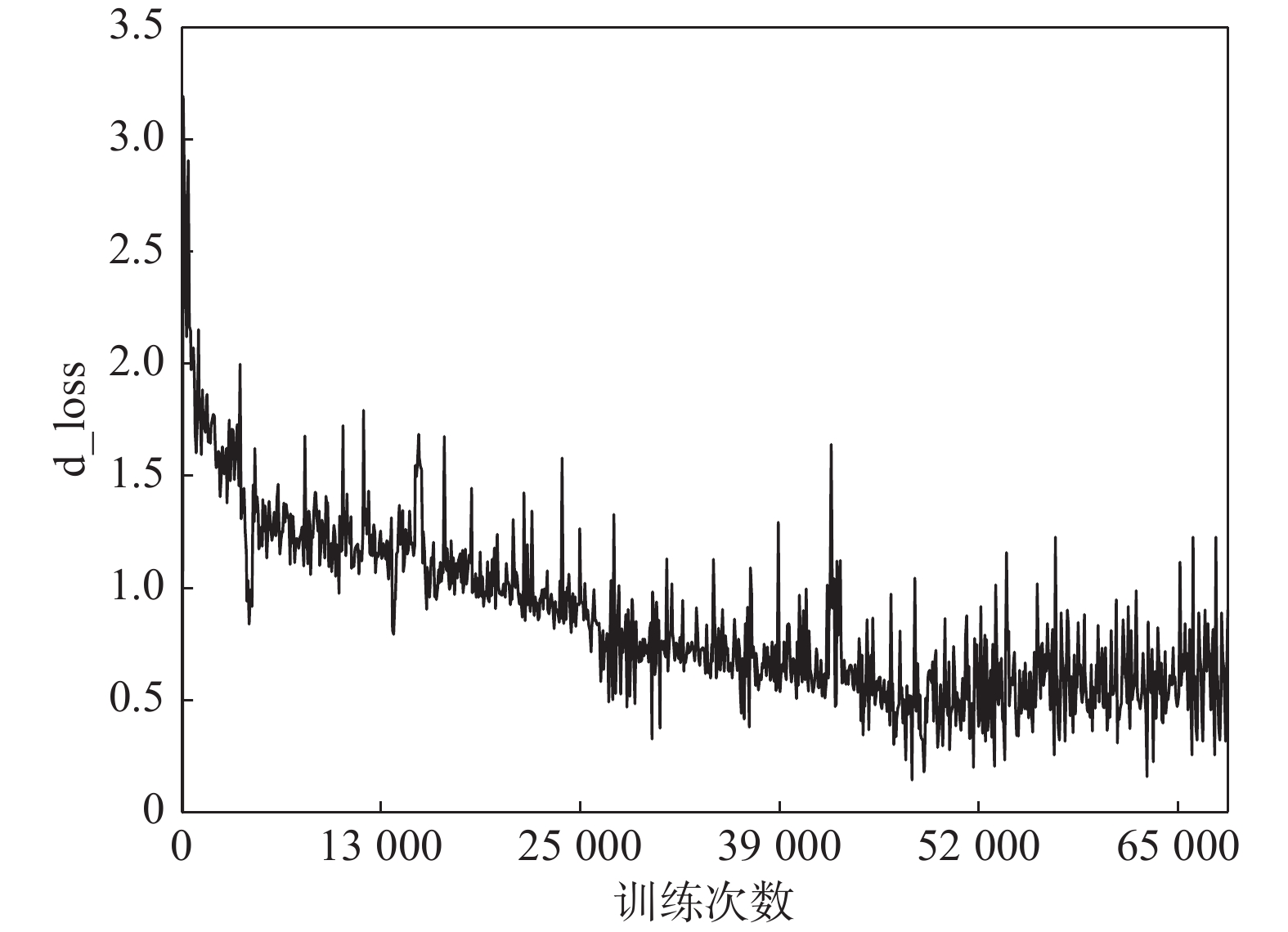
实验中采用VGG11作为目标网络F生成对抗样本用于训练模型的情况下, 在模型训练的过程中, AC-DefGAN在MNIST数据集上判别器的损失函数(d_loss)随训练次数增加而变化情况如图6所示. 从图6可以看出, 判别器在训练初期比较平滑, 随着训练次数的增加, 其损失函数出现了较大幅度震荡的状态, 这是因为判别器和生成器这两个网络结构互相对抗. 在总体趋势上, 判别器的损失函数处于逐渐下降的状态, 在对抗过程中, 判别损失函数越来越小, 意味着判别器对样本的真假判定及分类能力越来越强.
实验中, 我们对比分析了FGSM、BIM、DeepFool、CW-
${L_2}$ 、PGD等各类攻击方法对各类目标网络及AC-DefGAN进行攻击的攻击成功率, 如表1所示. 训练后的AC-DefGAN判别器对MNIST数据集的误分类率为0.3%, 对原始样本分类正确率高. 目标网络以及AC-DefGAN对各类攻击方法所产生的对抗样本的误分类率如表1所示, VGG11对FGSM所产生的攻击样本的误分类率为95.31%, AC-DefGAN对此攻击样本的误分类率为0.66%, AC-DefGAN对该攻击产生的对抗样本误分类率很低, 这表明经过训练后的AC-DefGAN可以有效防御该攻击. 从表1中可以看出, 各类攻击方法对目标网络进行攻击时, 目标网络对对抗样本的分类正确率很低; 对于不同攻击方法产生的对抗样本, AC-DefGAN的误分类率有所不同, 但在总体上其误分类率都较低. 实验结果表明, 就简单数据而言, 本文提出的方法能对原始样本和对抗样本进行正确分类, 能达到防御各类对抗攻击的目的.表 1 MNIST数据集中各目标网络、AC-DefGAN对各类对抗样本的误分类率Table 1 Misclassification rates of various adversarial examples for target models and AC-DefGAN on MNIST攻击方法 VGG11 ResNet-18 Dense-Net40 InceptionV3 Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN FGSM(%) 95.31 0.66 87.83 0.58 79.32 0.56 92.91 0.61 BIM(%) 95.70 0.78 88.51 0.69 82.01 0.64 93.12 0.73 DeepFool(%) 96.24 1.42 89.74 1.13 88.61 1.10 93.80 1.25 C&W(%) 99.37 1.79 97.52 1.71 96.21 1.68 98.93 1.75 PGD(%) 98.13 1.61 95.81 1.52 93.26 1.37 97.15 1.58 AC-DefGAN能够自定义神经网络分类器需要防御的攻击算法, 来完成分类器的防御训练, 使得分类器能够抵挡来自多种攻击算法产生的对抗样本的攻击. 在实验中, 采用FGSM、BIM、DeepFool、C&W、PGD等多种不同的攻击方法组成攻击算法集合来产生对抗样本, 用于模型的训练中. 表2为多种攻击同时对目标网络进行攻击产生对抗样本, AC-DefGAN对该对抗样本的误分类率. 如表2所示, 当多种攻击方法同时存在时, AC-DefGAN对其对抗样本的误分类率在0.59%到1.11%, 这表明 AC-DefGAN对这些对抗样本能保持较高的分类正确率, 具有较强的防御能力. 实验结果表明AC-DefGAN对多种攻击同时进行时的防御能力强, 能够有效抵御各类攻击方法的攻击.
表 2 MNIST数据集中AC-DefGAN对各攻击算法所生成对抗样本的误分类率Table 2 Misclassification rates of multiple adversarial examples for AC-DefGAN on MNIST攻击方法 VGG11 ResNet-18 Dense-Net40 InceptionV3 BIM、FGSM(%) 0.69 0.64 0.59 0.67 BIM、DeepFool(%) 1.11 0.91 0.87 1.01 FGSM、DeepFool(%) 1.05 0.86 0.81 0.93 BIM、FGSM、DeepFool(%) 1.01 0.84 0.79 0.89 为分析本文所提算法的有效性, 将AC-DefGAN与已有防御模型的防御效果进行比较分析. 表3为本文方法与其他防御方法分类结果的对比, 即在MNIST数据集中对同一目标网络F, 攻击算法的扰动阈值等超参数一致的情况下, 将AC-DefGAN与MagNet、对抗训练[17] (Adv. training)、APE-GAN、Defence-GAN等防御模型对各攻击算法所产生对抗样本的误分类率的进行对比分析. 如表3所示, 在MNIST数据集上, 对于攻击算法FGSM产生的对抗样本, APE-GAN与相应网络[33]训练得到的APE-GANm的误分类率为2.80%, Defence-GAN与分类器结合[30]训练得到的Defence-GAN-Rec的误分类率为1.11%, 对抗训练算法Adv. Training的误分类率为18.40%, MagNet的误分类率为80.91%, AC-DefGAN的误分类率为0.66%. 在这几个防御网络中, AC-DefGAN对FGSM所产生的对抗样本分类准确率最高, 且AC-DefGAN模型中的判别器可直接对样本进行分类; 而Defence-GAN、APE-GAN等可视为分类之前的附加或预处理步骤, 需与分类器结合对样本进行分类; 对抗训练的防御效果不如AC-DefGAN、Defence-GAN等方法; 而对于MagNet防御方法, 攻击者对分类器网络结构、参数等的了解程度会极大地影响其防御效果. 由实验结果可知, 在几种防御机制中, AC-DefGAN 对各类对抗样本的误分类率最低, 对于各攻击方法防御效果好.
当未确定或无法确定样本是否为攻击样本时, 利用AC-DefGAN可直接对样本进行正确分类, AC-DefGAN对与原始样本和对抗样本分类准确率都较高. 若利用Defence-GAN、APE-GAN进行处理后再通过分类器进行分类, 当样本为对抗样本时, 也可得到较为可靠的分类结果, 但当样本并非对抗样本时, 利用Defence-GAN、APE-GAN进行处理后再利用分类器分类则过于冗杂, 耗费时间与资源. 在MNIST数据集上, 对于Defense-GAN方法, 重构MNIST图像的时间平均约为0.675s, 再通过某一分类器进行图像分类, 分类器完成图像分类的具体时间视分类器模型而定. 而AC-DefGAN直接对图像进行分类, 时间远远短于Defense-GAN方法重构图像加上分类器再进行图像分类的时间. 在同等设备和相关条件下, 训练过程中, AC-DefGAN完成一轮训练所用时间约为23.43min, 而Defense-GAN完成一轮训练所用时间约为31.24 min, AC-DefGAN训练时间开销更小. AC-DefGAN在训练和分类时, 时间开销更小, 特别是在对对抗样本进行分类时, 可以直接进行分类, 无需进行图像重构再进行分类, 更加节省资源.
表 3 MNIST数据集中AC-DefGAN与各防御模型对各类对抗样本的误分类率Table 3 Misclassification rates of various adversarial examplesfor AC-DefGAN and other defense strategies on MNIST攻击方法 MagNet Adv. training APE-GANm Defence-GAN-Rec AC-DefGAN FGSM(%) 80.91 18.40 2.80 1.11 0.66 BIM(%) 83.09 19.21 2.91 1.24 0.78 DeepFool(%) 89.93 23.16 2.43 1.53 1.42 C&W(%) 93.18 62.23 1.74 2.29 1.67 粗体表示最优值. 为分析验证模型的泛化性能, 对于模型对未在模型攻击算法集合中的攻击算法的防御性能进行实验分析. 在MNIST 数据集实验中, 利用PGD攻击算法生成的对抗样本输入到模型中训练得到判别器, 该判别器对于PGD对抗样本的误分类率为1.87%, 而对于BIM对抗样本的误分类率为9.42%. 由次可以看出, 在MNIST 数据集中, 对于模型未接触过的攻击算, 模型的防御能力较强.
实验通过比较目标网络和AC-DefGAN对不同扰动阈值FGSM攻击目标网络时产生的对抗样本的误分类率, 以分析FGSM攻击方式下不同扰动大小对AC-DefGAN防御效果的影响. 表4展示了不同扰动阈值FGSM攻击中目标网络和AC-DefGAN对对抗样本的误分类率, 实验证明, 在简单数据集中, 对于不同扰动阈值的FGSM攻击, AC-DefGAN的分类准确率均保持在较高水平, FGSM攻击的扰动大小对AC-DefGAN防御效果影响不大.
表 4 MNIST数据集中目标网络、AC-DefGAN对不同扰动阈值$\varepsilon $ 的FGSM所产生对抗样本的误分类率(%)Table 4 Misclassification rates of adversarial examples generated from FGSM with different$\varepsilon $ for target model and AC-DefGAN on MNIST (%)FGSM的扰动阈值$\varepsilon $ 目标网络 AC-DefGAN $\varepsilon = 0.1$ 96.29 0.68 $\varepsilon = 0.2$ 96.98 0.83 $\varepsilon = 0.3$ 97.35 0.91 $\varepsilon = 0.4$ 98.76 1.69 综上, 对于MNIST数据集, AC-DefGAN可以对各类对抗攻击算法达到很好的防御效果, 可以自定义分类器需要防御的攻击算法来完成分类器的防御训练, 使得分类器能够抵挡来自各类攻击算法产生的对抗样本的攻击. 训练完成后, 判别器可以直接对原始样本和对抗样本进行正确分类, 模型鲁棒性较强, 且对于对抗攻击的防御效果比Defence-GAN等已有防御方法效果好.
3.2 CIFAR-10实验
在CIFAR-10实验中, 与 MNIST 数据集的实验相似, 采用类似图3和图4的结构作为AC-DefGAN的生成器和判别器. 训练过程中, 超参数设置如下:
1. Batch设置为32;
2. Epoch 设置为75轮;
3. 为了保持对抗平衡, 设置判别器与生成器的迭代次数为2: 1;
4. Adam 优化器的学习速率设置为 0.0002, 一阶矩估计的指数衰减率为 0.5, 二阶矩估计的指数衰减率为 0.9;
5. 根据各个损失函数对训练结果的影响, 为保证训练生成器完美的学习到训练样本的分布且生成的样本具有攻击性, 以使得训练得到的判别器对于原始样本和对抗样本具有较强的分类, 将损失函数
${L_{tf}}(G)$ 、${L_{cls}}(G)$ 和${L_{adv}}(G)$ 的权重$\alpha $ :$\beta $ :$\delta $ 设置为10:10:1.3.2.1 CIFAR-10实验结果分析
在CIFAR-10数据集, 实验采用VGG19作为目标网络F生成对抗样本用于训练的情况下, 在模型训练的过程中, AC-DefGAN在CIFAR-10数据集上判别器的损失函数(d_loss)随训练次数增加而变化情况如图7所示. 从图7可以看出, 判别器的损失函数处于逐渐下降的状态, 判别器训练前期相对平滑, 后期震荡明显, 这一现象说明随着训练次数的增加两个网络不断变得成熟, 由于两者之间对抗的关系, 因此判别器的损失函数出现了较大幅度震荡的状态. 在总体趋势上, 判别器的损失函数逐渐下降, 趋于稳定, 即在对抗过程中, 判别损失函数越来越小, 判别器对样本的真假判定及分类能力越来越强. 如图8所示, 对于CIFAR-10数据集, 在训练过程中, AC-DefGAN对于对抗样本
$x_{real}^{adv}$ 的分类准确率越来越高, 并逐渐趋于稳定. 这意味着随着训练次数增加, AC-DefGAN对于对抗攻击的防御能力逐渐增强, 最终达到防御攻击的目的.为分析AC-DefGAN对较为复杂的数据样本的分类能力, 我们对比分析了在CIFAR-10数据集下, 各类目标网络及AC-DefGAN对于FGSM、BIM、DeepFool、CW-
${L_2}$ 、PGD等各类攻击方法产生的对抗样本的误分类率, 如表5所示. 训练后的AC-DefGAN判别器对CIFAR-10数据集的误分类率为6.73%, 即本文所提出的算法能对原始样本进行正确分类. 目标网络以及AC-DefGAN对各类攻击方法所产生的对抗样本的误分类率如表5所示, VGG19对BIM所产生的攻击样本的误分类率为84.73%, AC-DefGAN对此攻击样本的误分类率为19.80%; 对于BIM对ResNet-18、Dense-Net40、InceptionV3其他目标网络所产生的对抗样本, AC-DefGAN对其误分类率在10%-20%之间, 这表明经过训练后的AC-DefGAN可以有效防御该攻击. AC-DefGAN及各分类网络对其余FGSM、DeepFool、C&W、PGD等各类攻击方法产生的对抗样本的误分类率见表5, 从表5中可以看出, 各类攻击方法对目标网络进行攻击时, 目标网络对对抗样本的分类正确率很低, 但AC-DefGAN对对抗样本的误分类率较低, 分类效果好. 从实验中可以看到, 在MNIST数据集AC-DefGAN对对抗样本的分类能力比在CIFAR-10 数据集上强, 这是训练数据本身的分布导致的. MNIST 数据集的样本是 (1,28,28) 的灰度样本, 而CIFAR-10 数据集的样本为 (3,32,32) 的彩色样本, CIFAR-10 数据分布的复杂程度远大于MNIST, 而数据分布的复杂程度是影响分类器对样本特征学习能力的重要因素之一, 对于越复杂的数据, 生成器越难以学习到数据的完整分布, 因此经过对抗训练后, 分类器对于样本的识别能力不如简单样本; 对于攻击算法而言, 在更复杂的数据集上, 在分类器上更容易找到对抗空间. 综上, 该实验结果表明, 对于复杂数据而言, 本文提出的方法能对原始样本和对抗样本进行正确分类, 能达到防御各类对抗攻击的目的.类似于在MNIST 数据集上的实验, 表6为在CIFAR-10数据集上当多种攻击同时对目标网络进行攻击时, AC-DefGAN对对抗样本的误分类率. 如表6所示, 当多种攻击方法同时对目标网络进行攻击时, AC-DefGAN对其对抗样本的误分类率在13.18%到22.14%, 这表明 AC-DefGAN对这些对抗样本能保持较高的分类正确率, 具有较强的防御能力. 实验结果表明, 在复杂数据集上, AC-DefGAN对多种攻击同时进行时的防御能力强, 能够有效抵御各类攻击方法的攻击.
为分析本文所提算法的在CIFAR-10数据集上的有效性, 将AC-DefGAN与已有防御模型的防御效果进行比较分析. 表7为本文方法与其他防御方法分类结果的对比, 在CIFAR-10数据集中对同一目标网络F, 攻击算法的扰动阈值等超参数一致的情况下, 比较分析目标模型与AC-DefGAN、 APE-GAN、Defence-GAN、Adv. training等防御模型对各攻击算法攻击目标模型时所产生对抗样本的误分类率. 如表7所示, 在CIFAR-10数据集上, 对于攻击算法FGSM、BIM产生的对抗样本, AC-DefGAN与APE-GAN、Defence-GAN、Adv. training等防御模型相比其误分类率最低; 对于攻击算法DeepFool, AC-DefGAN对对抗样本的误分类率低于Defence-GAN, 与APE-GAN接近. 在这几个防御网络中, 总体上AC-DefGAN对各攻击算法所产生的对抗样本分类准确率最高, 且Defence-GAN、APE-GAN等可视为分类之前的附加或预处理步骤, 需与分类器结合对样本进行分类, 而AC-DefGAN模型中的判别器可直接对样本进行分类. 综上, 在CIFAR-10数据集上的这几种防御机制中, AC-DefGAN 对各对抗攻击的防御效果最佳, 且训练结束后, 其判别器可以作为分类网络, 直接对样本进行正确分类.
表 5 CIFAR-10数据集中各目标网络、AC-DefGAN对各类对抗样本的误分类率Table 5 Misclassification rates of various adversarial examples for target models and AC-DefGAN on CIFAR-10攻击方法 VGG19 ResNet-18 Dense-Net40 InceptionV3 Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN FGSM(%) 77.81 16.95 74.92 13.07 73.37 13.74 76.74 15.49 BIM(%) 84.73 19.80 75.74 14.27 76.44 13.83 79.52 18.93 DeepFool(%) 88.52 23.47 83.48 22.55 86.16 21.79 88.26 23.15 C&W(%) 98.94 31.13 92.79 30.24 96.68 29.85 97.43 30.97 PGD(%) 87.13 28.37 86.41 26.29 86.28 25.91 87.04 26.74 表 6 CIFAR-10数据集中AC-DefGAN对各攻击算法所生成对抗样本的误分类率Table 6 Misclassification rates of multiple adversarial examples for AC-DefGAN on CIFAR-10攻击方法 VGG19 ResNet-18 Dense-Net40 InceptionV3 BIM、FGSM(%) 19.62 13.73 13.18 16.45 BIM、DeepFool(%) 21.71 18.65 17.42 22.14 FGSM、DeepFool(%) 20.95 15.21 16.35 19.78 BIM、FGSM、DeepFool(%) 21.37 17.56 16.93 20.81 表 7 CIFAR-10数据集中AC-DefGAN与各防御模型对各类对抗样本的误分类率Table 7 Misclassification rates of various adversarial examplesfor AC-DefGAN and other defense strategies on CIFAR-10攻击方法 目标网络F Adv. training APE-GANm Defence-GAN-Rec AC-DefGAN FGSM(%) 82.83 32.68 26.41 22.50 16.91 BIM(%) 89.75 39.49 24.33 21.72 19.83 DeepFool(%) 93.54 44.71 25.29 28.09 25.56 C&W(%) 98.71 78.23 30.50 32.21 30.24 粗体表示最优值. 为分析验证模型的泛化性能, 对于模型对未在模型攻击算法集合中的攻击算法的防御性能进行实验分析. 在CIFAR-10 数据集实验中, 利用PGD攻击算法生成的对抗样本输入到模型中训练得到判别器, 该判别器对于PGD对抗样本的误分类率为29.13%, 而对于FGSM对抗样本的误分类率为23.74%. 由实验结果可以看出, 在CIFAR-10 数据集中, 与MNIST 数据集相似, 对于模型未接触过的攻击算法, 模型的防御能力较强. PGD攻击是比较强大的一阶攻击(直接利用梯度进行攻击), 在某种意义上在深度学习中依赖于一阶信息的攻击是比较普遍的, 对于该类攻击, 若训练网络对于PGD具有较强的防御效果, 则该网络能对其他攻击具有鲁棒性. 并且, 在本文模型中可以设置多种攻击算法生成对抗样本来训练网络, 这使得训练得到的分类器对于目前大多数的攻击算法具有较好的抵抗力.
表8展示了在CIFAR-10数据集上不同扰动阈值FGSM攻击中目标网络和AC-DefGAN对对抗样本的误分类率, 以分析FGSM攻击方式下不同扰动大小对AC-DefGAN防御效果的影响. 实验证明, 与在MNIST数据集上的实验结果相似, 在CIFAR-10数据集上, 对于不同扰动阈值的FGSM攻击, AC-DefGAN的分类准确率均保持在较高水平, FGSM攻击的扰动大小对AC-DefGAN防御效果影响不大.
表 8 CIFAR-10数据集中目标网络、AC-DefGAN对不同扰动阈值$\varepsilon $ 的FGSM所产生对抗样本的误分类率(%)Table 8 Misclassification rates of adversarial examples generated from FGSM with different$\varepsilon $ for target model and AC-DefGAN on CIFAR-10 (%)FGSM的扰动阈值$\varepsilon $ 目标网络 AC-DefGAN $\varepsilon = 0.1$ 77.82 12.92 $\varepsilon = 0.2$ 80.89 17.47 $\varepsilon = 0.3$ 82.33 18.86 $\varepsilon = 0.4$ 84.74 24.13 上述几个实验证明了在CIFAR-10数据集上, AC-DefGAN可以对各类对抗攻击算法达到很好的防御效果, 可以自定义分类器需要防御的攻击算法来完成分类器的防御训练, 使得分类器能够抵挡来自各类攻击算法产生的对抗样本的攻击. 训练完成后, 判别器可以直接对原始样本和对抗样本进行正确分类, 模型鲁棒性较强, 且对于对抗攻击的防御效果比Defence-GAN等已有防御方法效果好.
3.3 ImageNet实验
在ImageNet实验中, 与 MNIST 数据集的实验相似, 采用类似图3和图4的结构作为AC-DefGAN的生成器和判别器. 训练过程中, 超参数设置如下:
1. Batch设置为32;
2. Epoch 设置为125轮;
3. 为了保持对抗平衡, 设置判别器与生成器的迭代次数为2: 1;
4. Adam 优化器的学习速率设置为 0.0002, 一阶矩估计的指数衰减率为 0.5, 二阶矩估计的指数衰减率为 0.9;
5. 根据各个损失函数对训练结果的影响, 为保证训练生成器完美的学习到训练样本的分布且生成的样本具有攻击性, 以使得训练得到的判别器对于原始样本和对抗样本具有较强的分类, 将损失函数
${L_{tf}}(G)$ 、${L_{cls}}(G)$ 和${L_{adv}}(G)$ 的权重$\alpha $ :$\beta $ :$\delta $ 设置为10:10:1.3.3.1 ImageNet实验结果分析
为分析AC-DefGAN对更为复杂的数据样本的分类能力, 我们对比分析了在ImageNet9 (Top 1) 下, 各类目标网络及AC-DefGAN对于各类攻击方法产生的对抗样本的误分类率, 如表9所示. 训练后的AC-DefGAN判别器对ImageNet 数据集的误分类率为20.2%, 即本文所提出的算法能对原始样本进行正确分类. VGG19对于C&W产生的对抗样本误分类率为97.39%, 而AC-DefGAN对C&W攻击样本的误分类率为39.1%, 这表明经过训练后的AC-DefGAN可以有效防御C&W攻击. 从表9中AC-DefGAN及各分类网络对FGSM、DeepFool、C&W等各类攻击方法产生的对抗样本误分类率可以看出, 各目标网络对各类攻击方法防御能力低, 对对抗样本误分类率高, 而AC-DefGAN对各类对抗样本的误分类率较低, 抵御攻击的效果好. 综上, 该实验结果表明, 在高维数据空间中, 本文提出的方法能达到防御对抗攻击的目的.
表 9 ImageNet数据集中各类目标网络、AC-DefGAN对各类对抗样本的误分类率Table 9 Misclassification rates of various adversarial examples for target models and AC-DefGAN on ImageNet攻击方法 VGG19 ResNet-18 Dense-Net40 InceptionV3 Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN Target model AC-DefGAN FGSM(%) 71.21 39.42 69.14 38.52 68.42 37.92 69.65 38.77 DeepFool(%) 88.45 44.80 85.73 42.96 86.24 43.17 87.67 44.63 C&W(%) 97.39 39.13 96.19 36.75 95.84 36.74 96.43 38.68 为分析本文所提算法的在ImageNet数据集上的有效性, 将AC-DefGAN与APE-GAN的防御效果进行比较分析. 表10为本文方法与其他防御方法分类结果的对比, 在ImageNet 数据集中对于同一目标网络F, 攻击算法的扰动阈值等超参数一致的情况下, 将目标模型与AC-DefGAN、 APE-GAN防御模型对各攻击算法产生对抗样本的误分类率进行比较分析. 如表10所示, 在ImageNet数据集上, AC-DefGAN对于各对抗样本的误分类率相较于APE-GAN更低, 也远远低于目标网络对于对抗样本的误分类率, 这表明AC-DefGAN在ImageNet数据集上能有效抵御对抗攻击. 且AC-DefGAN模型中的判别器可直接对样本进行分类, 而APE-GAN需与分类器结合对样本进行分类, AC-DefGAN相较于APE-GAN防御效果更好更有效.
综上, 在ImageNet数据集上, AC-DefGAN对各类对抗攻击算法防御效果显著, 训练完成后, 判别器可以直接对样本进行正确分类, 且对于对抗攻击的防御效果比APE-GAN等已有防御方法效果好.
表 10 ImageNet数据集中AC-DefGAN与各防御模型对各攻击算法对抗样本的误分类率(%)Table 10 Misclassification rates of various adversarial examples for AC-DefGAN and other defense strategies on ImageNet (%)攻击方法 目标网络 APE-GANm AC-DefGAN FGSM 72.92 40.14 38.94 C&W 97.84 38.70 36.52 BIM 76.79 41.28 40.78 DeepFool 94.71 45.93 44.31 粗体表示最优值. 4. 总结与展望
针对现有的对抗攻击防御方法耗费大量时间、资源, 且鲁棒性较差, 无法同时防御多种攻击算法等缺点, 本文利用生成对抗模型在数据表达和分布学习上的优势, 将生成对抗网络和现有的攻击算法结合, 提出一种更为高效、鲁棒性更强的对抗攻击防御模型. 在本文所提出的方法中, 一旦网络训练完成, 判别器可对对抗样本进行正确分类, 该模型可以自定义分类器需要防御的攻击算法, 产生对应对抗样本完成分类器的训练, 使得分类器能够防御来自多种对抗样本的攻击. 在该模型中, 生成器提供了关于数据分布的更多信息, 可以改善对抗训练过程, 提高训练后分类器的鲁棒性, 使得该模型具有更强的泛化能力. 通过将对抗样本加入到生成对抗网络的训练中, 可以利用GAN在数据学习上的优势提高分类器在对抗训练中的鲁棒性, 同时对抗攻击也能提高GAN的优化效率从而更好地训练生成器判别器, 提高训练后判别器的鲁棒性, 使得该模型具有更强的泛化能力. 本文方法AC-DefGAN在MNIST数据集、CIFAR-10数据集和ImageNet数据集上进行实验, 均取得优异的实验成果, 实验证明了本文所提出的基于生成对抗网络的对抗攻击防御模型AC-DefGAN在对抗攻击防御领域的有效性及优势. 总的来说, 本文所提方法中判别器在训练后对原始样本和对抗样本均具有很高的分类正确率, 相比于其他方法, 本文所提方法可以防御的多种攻击算法, 鲁棒性好, 判别器可以直接对原始样本和对抗样本进行正确分类, 且防御效果优于已有防御方法. 本文仍有不足之处, 在接下来的工作中, 在实验条件与设备允许的情况下, 将在生成器和判别器的内部网络的设计上使用更复杂的网络, 进一步探究提高判别器的分类正确率与鲁棒性. 训练集中对抗样本的设置对于判别器训练的影响也是一个非常值得继续探讨的方向, 在下一步工作中将继续探究训练集中对抗样本的设置对分类器训练的影响, 进一步探究提高判别器的分类正确率与鲁棒性. 同时, 在本文的训练细节中, 关于超参数的设置方面等方面, 需要通过经验化设计的超参数比较多, 在接下来的研究工作中, 将优化重点放在超参数和测试指标相关联上, 以最终实现超参数的自适应调节.
-
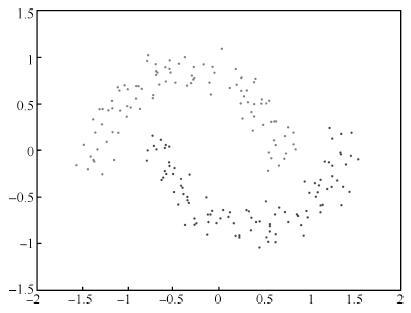
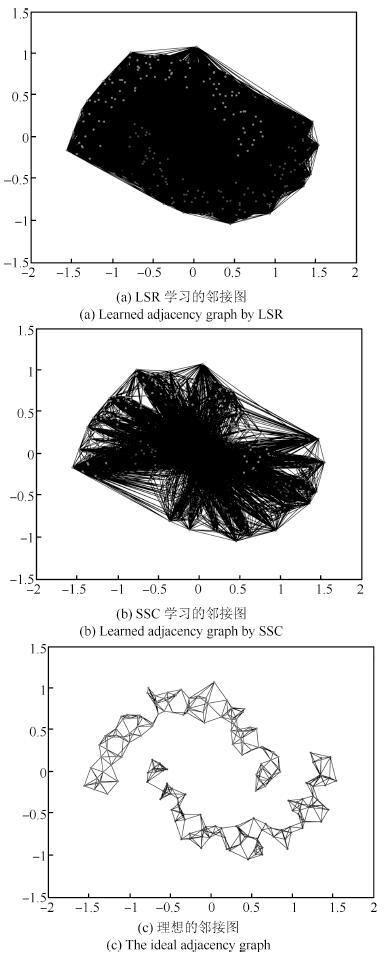
表 1 双月形数据上聚类准确率(%)和运行时间(s)的对比
Table 1 Clustering accuracy (%) and running time (s)comparison on the two-moon synthetic data
HC K-means LRR SSC LSR BD-LRR RLLRR SMR LSSC LLSR ACC 100.00 72.00 53.50 53.50 50.00 50.00 52.00 51.50 100.00 100.00 (0.001) (0.005) (0.0001) (0.08) (0.1) (0.001) (0.0001,5) (0.0001,5) Time 0.0010 0.0026 1.89 4.80 0.0008 19.15 0.33 0.045 0.94 0.10 表 2 数据集描述
Table 2 Summary of the data sets
数据集 样本 长 宽 类别 ORL10P 100 112 92 10 PIX10P 100 100 100 10 PIE10P 210 55 44 10 Umist 575 28 23 20 USPS 1 000 16 16 10 COIL20 1 440 32 32 20 表 3 聚类准确率
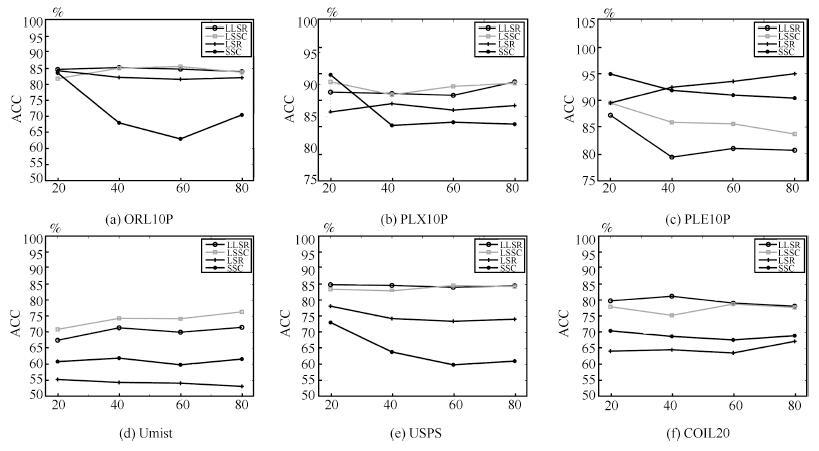
Table 3 Clustering accuracy (%)
HC K-means LRR SSC LSR BD-LRR RLLRR SMR LSSC LLSR ORL10P 41.00 73.40 79.00 71.00 83.00 70.30 74.70 78.00 86.00 87.00 PIX10P 77.00 79.90 87.00 86.00 85.00 76.80 56.10 88.00 96.00 97.00 PIE10P 70.95 32.95 100.00 90.00 90.00 80.00 79.43 100.00 98.57 100.00 Umist 45.57 47.58 52.17 61.57 52.35 48.35 50.96 69.91 76.87 74.09 USPS 10.90 73.14 78.60 60.80 71.30 63.90 65.50 77.10 81.20 91.20 COIL20 53.47 60.10 65.69 72.01 63.40 67.72 68.80 67.15 78.26 79.58 表 4 运行时间的对比(s)
Table 4 Running time (s) comparison
HC K-means LRR SSC LSR BD-LRR RLLRR SMR LSSC LLSR ORL10P 0.0011 0.0071 0.54 0.21 0.00078 7.29 1.69 0.014 0.14 0.034 PIX10P 0.00095 0.0062 1.04 0.33 0.00073 7.60 1.72 0.012 1.25 0.035 PIE10P 0.0023 0.011 4.51 2.53 0.0015 22.23 2.14 0.057 0.32 0.13 Umist 0.011 0.037 25.14 62.27 0.015 240.70 13.48 0.71 1.52 0.92 USPS 0.034 0.091 130.61 124.57 0.044 884.42 120.33 4.53 3.57 2.75 COIL20 0.072 0.071 423.54 2.51 1446.67 926.78 134.86 18.92 18.97 5.69 表 5 数据集描述
Table 5 Summary of the data sets
数据集 样本 基因 类别 Leukemia1 72 5 327 3 SRBCT 83 2 308 4 Lung_Cancer 203 12 600 5 Prostate_Tumor 102 10 509 2 表 6 聚类准确率(%)
Table 6 Clustering accuracy (%)
HC K-means LRR SSC LSR BD-LRR RLLRR SMR LSSC LLSR Leukemia1 54.17 69.31 86.11 58.33 77.78 79.17 54.17 77.78 90.28 90.28 SRBCT 36.14 53.73 68.43 40.12 54.22 60.24 46.99 63.68 74.70 74.46 Lung_Cancer 78.33 83.50 87.39 83.74 92.61 85.22 84.24 90.64 91.63 92.61 Prostate_Tumor 51.96 63.73 62.75 56.86 62.75 60.78 60.78 59.80 66.67 69.61 -
[1] Yang A Y, Wright J, Ma Y, Sastry S S. Unsupervised segmentation of natural images via lossy data compression. Computer Vision and Image Understanding, 2008, 110(2):212-225 doi: 10.1016/j.cviu.2007.07.005 [2] Vidal R, Tron R, Hartley R. Multiframe motion segmentation with missing data using power factorization and GPCA. International Journal of Computer Vision, 2008, 79(1):85-105 doi: 10.1007/s11263-007-0099-z [3] 王卫卫, 李小平, 冯象初, 王斯琪. 稀疏子空间聚类综述. 自动化学报, 2015, 41(8):1373-1384 http://www.aas.net.cn/CN/abstract/abstract18712.shtmlWang Wei-Wei, Li Xiao-Ping, Feng Xiang-Chu, Wang Si-Qi. A survey on sparse subspace clustering. Acta Automatica Sinica, 2015, 41(8):1373-1384 http://www.aas.net.cn/CN/abstract/abstract18712.shtml [4] Hong W, Wright J, Huang K, Ma Y. Multiscale hybrid linear models for lossy image representation. IEEE Transactions on Image Processing, 2006, 15(12):3655-3671 doi: 10.1109/TIP.2006.882016 [5] Vidal R, Favaro P. Low rank subspace clustering (LRSC). Pattern Recognition Letters, 2014, 43:47-61 doi: 10.1016/j.patrec.2013.08.006 [6] Elhamifar E, Vidal R. Sparse subspace clustering. In:Proceedings of the 2009 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Miami, FL, USA:IEEE, 2009.2790-2797 https://www.computer.org/csdl/proceedings/cvpr/2009/3992/00/index.html [7] Liu G C, Lin Z C, Yu Y. Robust subspace segmentation by low-rank representation. In:Proceedings of the 27th International Conference on Machine Learning (ICML). Haifa, Israel, 2010.663-670 [8] Lu C Y, Min H, Zhao Z Q, Zhu L, Huang D S, Yan S C. Robust and efficient subspace segmentation via least squares regression. In:Proceedings of the 12th European Conference on Computer Vision (ECCV). Florence, Italy:Springer, 2012.347-360 [9] Zhang H Y, Lin Z C, Zhang C, Cao J B. Robust latent low rank representation for subspace clustering. Neurocomputing, 2014, 145:369-373 doi: 10.1016/j.neucom.2014.05.022 [10] Hu H, Lin Z C, Feng J J, Zhou J. Smooth representation clustering. In:Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Columbus, OH, USA:IEEE, 2014.3834-3841 [11] Soltanolkotabi M, Elhamifar E, Candés E J. Robust subspace clustering. The Annals of Statistics, 2014, 42(2):669-699 doi: 10.1214/13-AOS1199 [12] Feng J S, Lin Z C, Xu H, Yan S C. Robust subspace segmentation with block-diagonal prior. In:Proceedings of the 2014 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). Columbus, OH, USA:IEEE, 2014.3818-3825 [13] Tibshirani R. Regression shrinkage and selection via the lasso. Journal of the Royal Statistical Society. Series B (Methodological), 1996, 58(1):267-288 http://cn.bing.com/academic/profile?id=2135046866&encoded=0&v=paper_preview&mkt=zh-cn [14] Hoerl A E, Kennard R W. Ridge regression:biased estimation for nonorthogonal problems. Technometrics, 1970, 12(1):55-67 doi: 10.1080/00401706.1970.10488634 [15] Fan J Q, Li R Z. Variable selection via nonconcave penalized likelihood and its oracle properties. Journal of the American statistical Association, 2001, 96(456):1348-1360 doi: 10.1198/016214501753382273 [16] Lee S R, Heo G S, Lee C Y. Representation and symbolization of motion captured human action by locality preserving projections. Applied Mathematics & Information Sciences, 2014, 8(1):441-446 http://cn.bing.com/academic/profile?id=2324349122&encoded=0&v=paper_preview&mkt=zh-cn [17] Roweis S T, Saul L K. Nonlinear dimensionality reduction by locally linear embedding. Science, 2000, 290(5500):2323-2326 doi: 10.1126/science.290.5500.2323 [18] Tang Y Y, Yuan H L, Li L Q. Manifold-based sparse representation for hyperspectral image classification. IEEE Transactions on Geoscience and Remote Sensing, 2014, 52(12):7606-7618 doi: 10.1109/TGRS.2014.2315209 [19] Boyd S, Parikh N, Chu E, Peleato B, Eckstein J. Distributed optimization and statistical learning via the alternating direction method of multipliers. Foundations and Trends in Machine Learning, 2011, 3(1):1-122 http://cn.bing.com/academic/profile?id=2164278908&encoded=0&v=paper_preview&mkt=zh-cn [20] Shi J B, Malik J. Normalized cuts and image segmentation. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2000, 22(8):888-905 doi: 10.1109/34.868688 [21] Cai D, He X F, Wu X Y, Han J W. Non-negative matrix factorization on manifold. In:Proceedings of the 8th IEEE International Conference on Data Mining (ICDM). Pisa:IEEE, 2008.63-72 [22] Hou C P, Nie F P, Yi D Y, Tao D C. Discriminative embedded clustering:a framework for grouping high-dimensional data. IEEE Transactions on Neural Networks and Learning Systems, 2015, 26(6):1287-1299 doi: 10.1109/TNNLS.2014.2337335 期刊类型引用(15)
1. 马盈仓,吴也凡,邢志伟,袁林. 基于k-近邻局部线性邻域重建的多视角聚类算法. 纺织高校基础科学学报. 2023(03): 75-83 .  百度学术
百度学术2. 简彩仁,翁谦,夏靖波. 系数增强最小二乘回归子空间聚类法. 计算机工程与应用. 2022(20): 73-78 .  百度学术
百度学术3. 郑毅,马盈仓,杨小飞,续秋霞. 基于k-近邻与局部相似度的稀疏子空间聚类. 计算机工程与应用. 2020(04): 99-108 .  百度学术
百度学术4. 王雨思,路德杨,李海洋. 基于分式函数约束的稀疏子空间聚类方法. 计算机工程与应用. 2020(07): 39-47 .  百度学术
百度学术5. 朱恒东,马盈仓,杨婷,张要. 基于ε-邻域和拉普拉斯矩阵秩约束的谱聚类算法. 纺织高校基础科学学报. 2020(01): 88-94 .  百度学术
百度学术6. 肖金安,贺兴时,王燕. 自适应逐步混合截尾MOEE分布的统计分析. 纺织高校基础科学学报. 2020(01): 95-99 .  百度学术
百度学术7. 蒋忆睿,裴洋,陈磊,王文乐,代江艳,易玉根. 多局部约束自表示的谱聚类算法. 计算机工程与应用. 2020(11): 172-178 .  百度学术
百度学术8. 贾瑞玉,宋飞豹,汤深伟. 双精英遗传策略的基因聚类算法. 小型微型计算机系统. 2020(07): 1375-1380 .  百度学术
百度学术9. 陈晓云,廖梦真. 基于稀疏和近邻保持的极限学习机降维. 自动化学报. 2019(02): 325-333 .  本站查看
本站查看10. 邱云飞,费博雯,刘大千,刘兴. 稀疏条件下的重叠子空间聚类算法. 计算机应用研究. 2019(03): 657-662 .  百度学术
百度学术11. 邱云飞,刘畅. 基于加权集成Nystr?m采样的谱聚类算法. 模式识别与人工智能. 2019(05): 420-428 .  百度学术
百度学术12. 邱云飞,费博雯,刘大千. 局部加权最小二乘回归的重叠子空间聚类算法. 模式识别与人工智能. 2018(02): 114-122 .  百度学术
百度学术13. 许福,徐建. 基于组合增量聚类的数据流异常检测研究. 计算机与数字工程. 2017(08): 1473-1478+1508 .  百度学术
百度学术14. 邱云飞,费博雯,刘大千. 基于概率模型的重叠子空间聚类算法. 模式识别与人工智能. 2017(07): 609-621 .  百度学术
百度学术15. 张鹏涛,陈晓云. 弹性核子空间聚类. 模式识别与人工智能. 2017(09): 779-790 .  百度学术
百度学术其他类型引用(21)
-




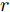
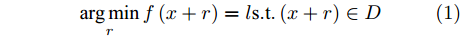
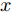
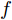
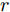
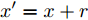
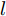
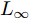
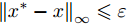
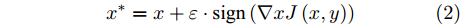
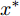
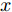
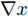

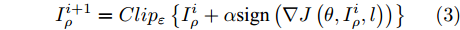


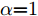
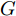
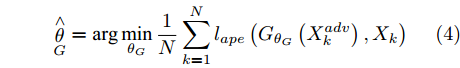
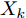
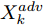
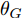
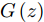
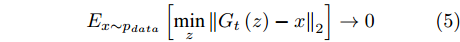
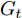

 下载:
下载:
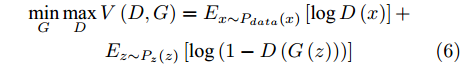
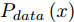
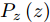
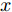
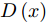
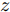
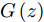
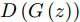
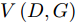
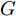
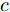
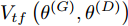
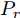
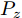
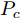
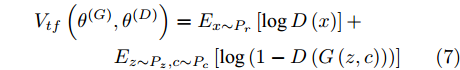
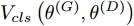
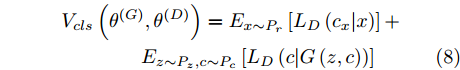
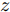
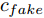


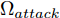
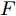
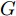
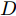
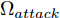


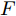
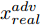
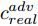
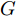
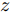
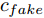
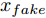
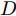
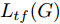
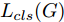
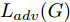
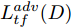
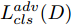
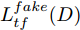
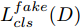
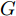
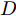
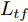
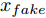
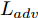
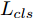
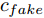
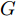
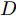
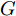
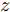
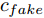
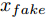
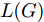
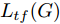
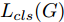
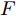
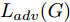
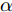
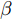
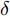
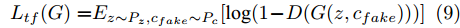
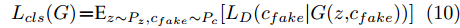
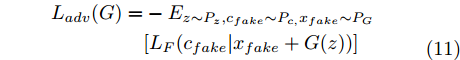
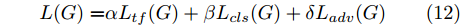
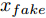
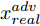
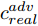
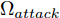

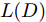
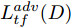
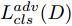
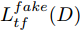
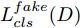


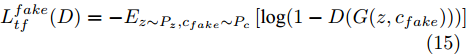
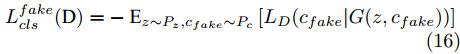
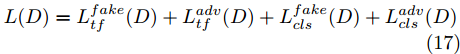
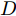
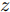
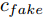
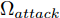

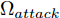
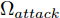
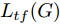
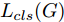
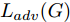
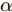
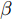
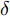
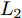


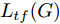
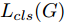
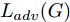
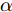
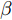
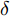
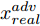

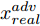
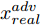
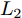


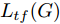
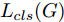
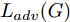
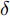
 下载:
下载: